
大家好,我是来自paipai队的徐一帆,Github ID IvanaXu [01],相关研究工作都会开源在上面,欢迎大家关注!
以下是关于本次“云上进化”2022全球AI生物智药大赛 [02]:赛道二“抗原抗体结合Epitope和Paratope精准确定”的参赛分享。
一、团队介绍

paipai队,取自 PAIN + AI,我们团队先后打过很多不同领域的算法比赛,之前参加过“创新大师杯”冷冻电镜蛋白质结构建模大赛 [03],为继续深入生物计算相关技术,进而参加了本次比赛。我们同时打了本次“云上进化”2022全球AI生物智药大赛:赛道一“基于AI算法的SARS-CoV-2广谱中和抗体药物设计” [04]、赛道二“抗原抗体结合Epitope和Paratope精准确定” [05]。
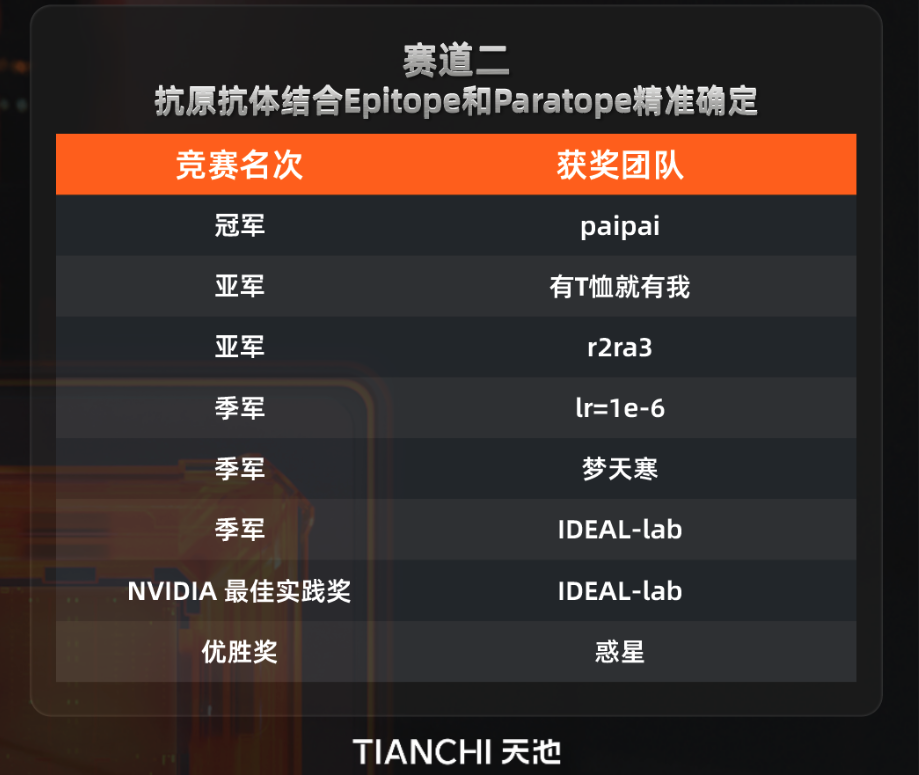
其中,赛道一初赛Rank14、复赛Rank10;赛道二我们是较早出成绩的,初赛阶段0.29159/2022-10-09,0.3264/2022-10-25,之后由于时间关系就没更新了,所幸Rank15边缘晋级;复赛阶段我们进行了方案的主要优化,取得0.4410/2022-12-20,拿下Rank01;决赛阶段由于春节假期的时间投入,最终优化至0.4607/2023-02-06。
经2023-02-23 决赛答辩后 [06],

承蒙各位评委老师的错爱,我们有幸取得本次比赛 赛道二“抗原抗体结合Epitope和Paratope精准确定” 冠军!
接下来,介绍主要算法方案。
二、主要方案
2.1 问题拆解
表位(Epitope)是存在于抗原表面的,决定抗原特异性的特殊性结构的化学基团称为抗原决定簇,又称表位。抗原通过表位与相应淋巴细胞表面抗原受体结合,从而激活淋巴细胞,引起免疫应答;抗原也借此与相应抗体或致敏淋巴细胞发生特异性结合。单个抗原分子可具有一种或多种不同的表位,其大小相当于相应抗体的抗原结合部位,每种表位只有一种抗原特异性。因此,表位是被免疫细胞识别的靶结构,也是免疫反应具有特异性的基础,其性质、数目和空间构型决定着抗原的特异性。与之对应的抗体结合部位(Paratope)是与抗原表位相结合的抗体上的位点。
表位和相应的抗体结合部位的确定的重要性,可想而知,它揭示了免疫系统对抗病毒的具体的机理。举个例子,几天前,一个新发现的新冠病毒抗体SP1-77及其谱系成员表现出非常广泛的活性。是什么使SP1-77抗体拥有如此广泛的中和作用?结构研究表明,SP1-77与当前抗体的工作原理大相径庭。研究人员解释说,现有许多抗体通过结合特定位置的刺突蛋白受体结合域(RBD)来防止新冠病毒与人体细胞的血管紧张素转化酶2(ACE2)受体结合。SP1-77抗体也与RBD结合,但方式完全不同,不会阻止病毒与ACE2受体结合,但SP1-77可防止病毒外膜与标靶细胞膜融合,这阻碍了病毒感染最后一个必要步骤。这就是结构基础上的表位和抗体结合部位的确定,能为人们解码抗体与病毒作用的细节方式。但是由于新冠病毒的表位不多,很容易被误打误撞地猜到,所以我们这个赛题不聚焦在新冠病毒。而是针对各种各样的抗原和抗体复合物。
根据比赛赛题以及数据解析 [07],
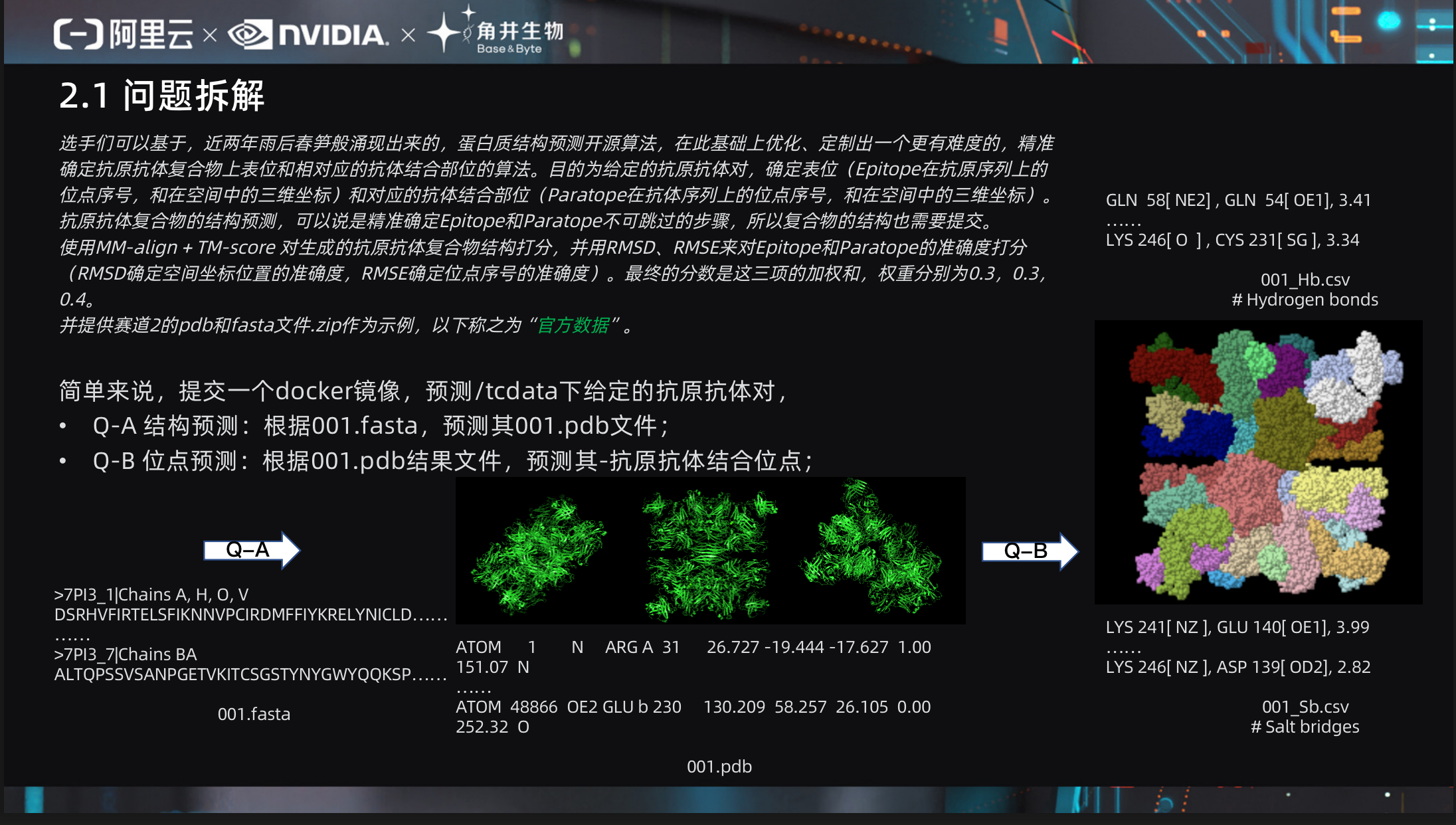
选手们可以基于,近两年雨后春笋般涌现出来的,蛋白质结构预测开源算法,在此基础上优化、定制出一个更有难度的,精准确定抗原抗体复合物上表位和相对应的抗体结合部位的算法。目的为给定的抗原抗体对,确定表位(Epitope在抗原序列上的位点序号,和在空间中的三维坐标)和对应的抗体结合部位(Paratope在抗体序列上的位点序号,和在空间中的三维坐标)。抗原抗体复合物的结构预测,可以说是精准确定Epitope和Paratope不可跳过的步骤,所以复合物的结构也需要提交。
使用MM-align + TM-score 对生成的抗原抗体复合物结构打分,并用RMSD、RMSE来对Epitope和Paratope的准确度打分(RMSD确定空间坐标位置的准确度,RMSE确定位点序号的准确度)。最终的分数是这三项的加权和,权重分别为0.3、0.3、0.4。[08][09]
简单来说,我们需要提交一个Docker镜像 [10],预测/tcdata下给定的抗原抗体对,并完成如图所示:
- 任务A-结构预测,即根据001.fasta,预测其001.pdb文件;
- 任务B-位点预测,即根据001.pdb结果文件,预测其抗原抗体结合位点;
2.2 结构预测
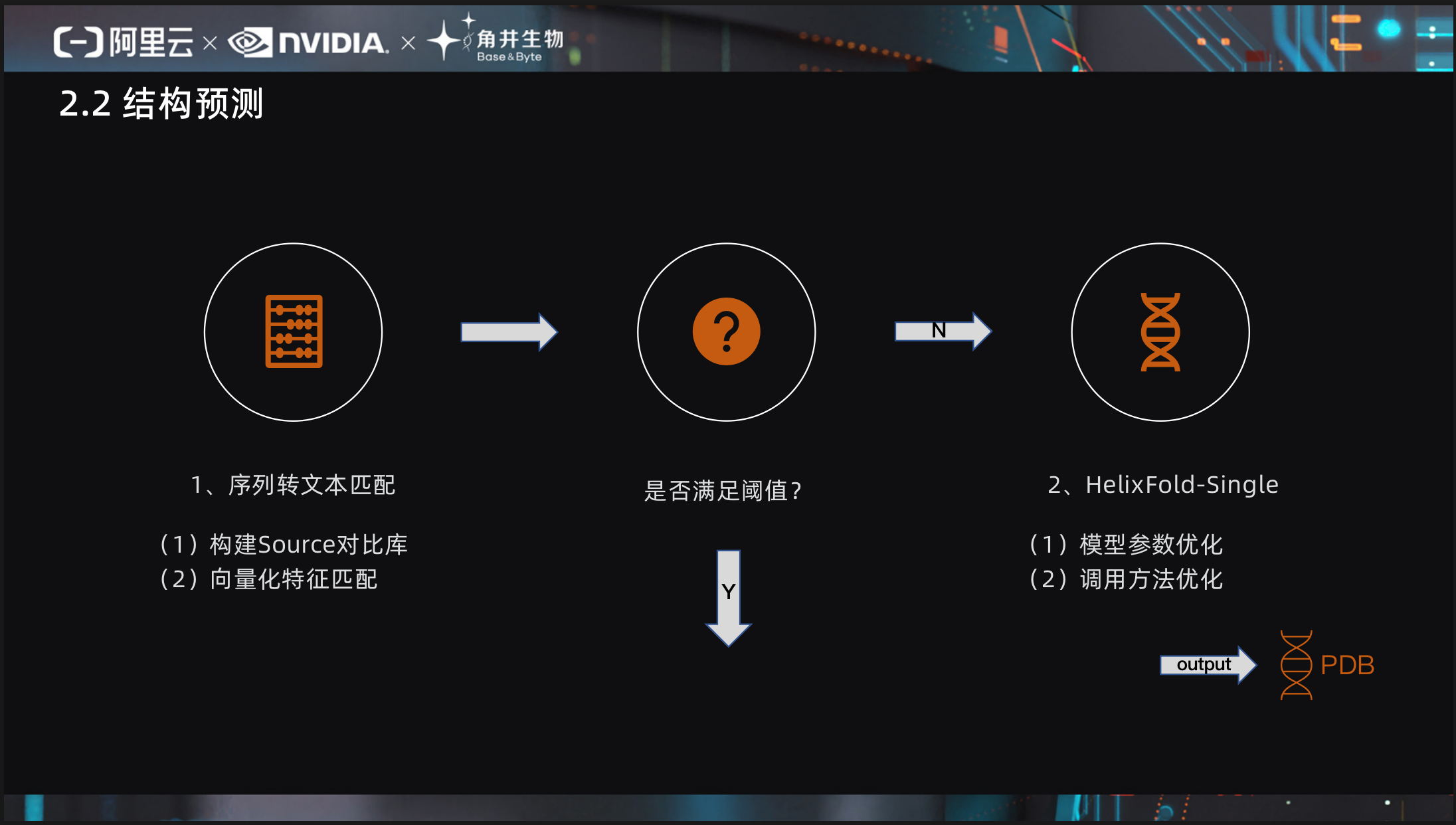
如图流程所示,我们先完成1、序列转文本匹配,预先构建Source对比库,并进行向量化特征匹配,若此时输出结果满足设定阈值,则直接输出,反之我们调用2、HelixFold-Single [11],百度类AlphaFold [12] 提出蛋白质结构预测方法 [13],但我们选用了其Single版本。并对其进行了一定程度上的优化:(1)模型参数优化、(2)调用方法优化等。
- 也有幸在决赛答辩环节认识HelixFold其开发者 @IDEAL-lab团队,在此留一个大写的赞!
2.2.1 序列转文本匹配
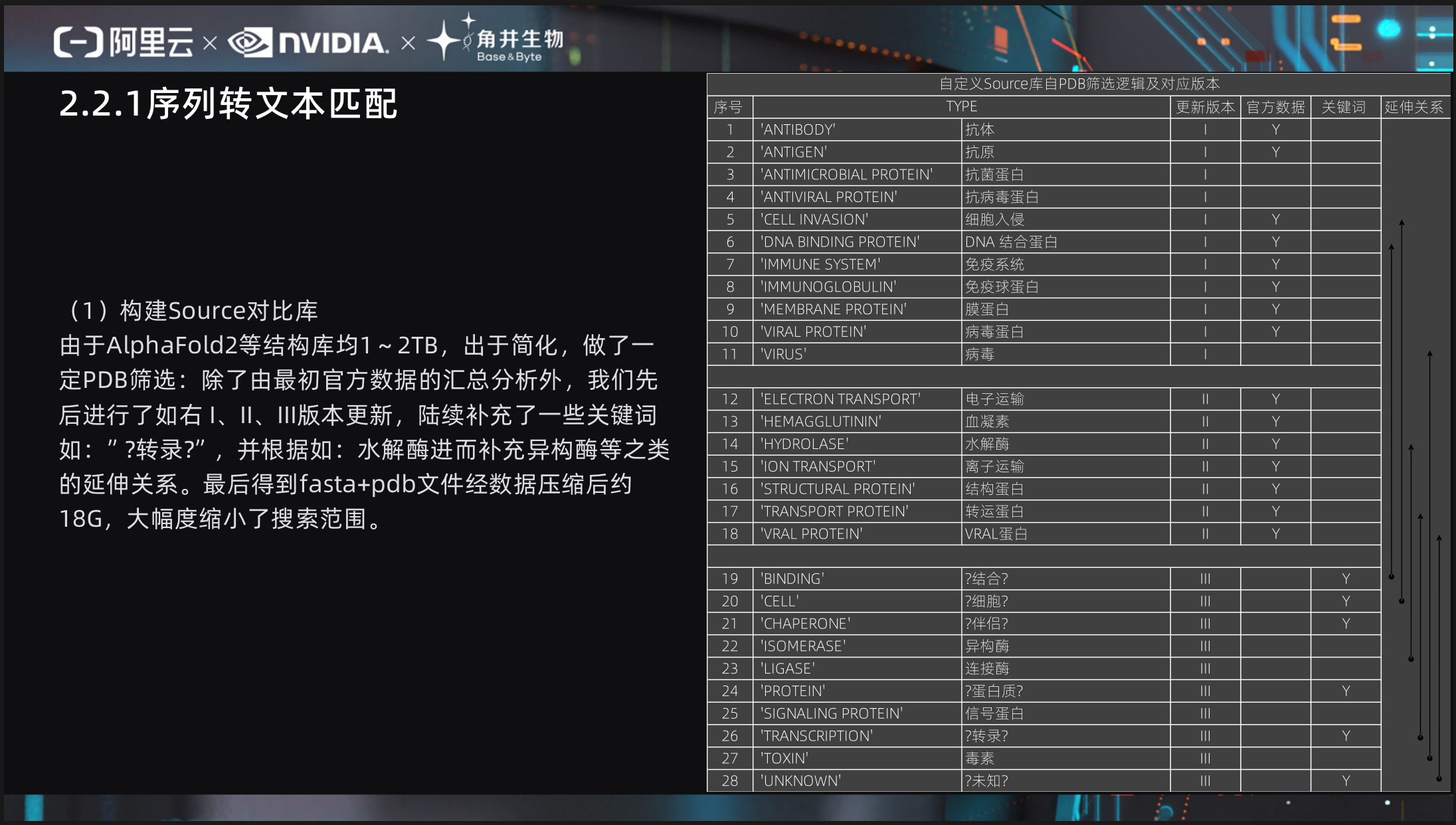
(1)构建Source对比库

如上,由于AlphaFold2 [14]等结构库均1~2.2TB,出于简化,做了一定PDB筛选:
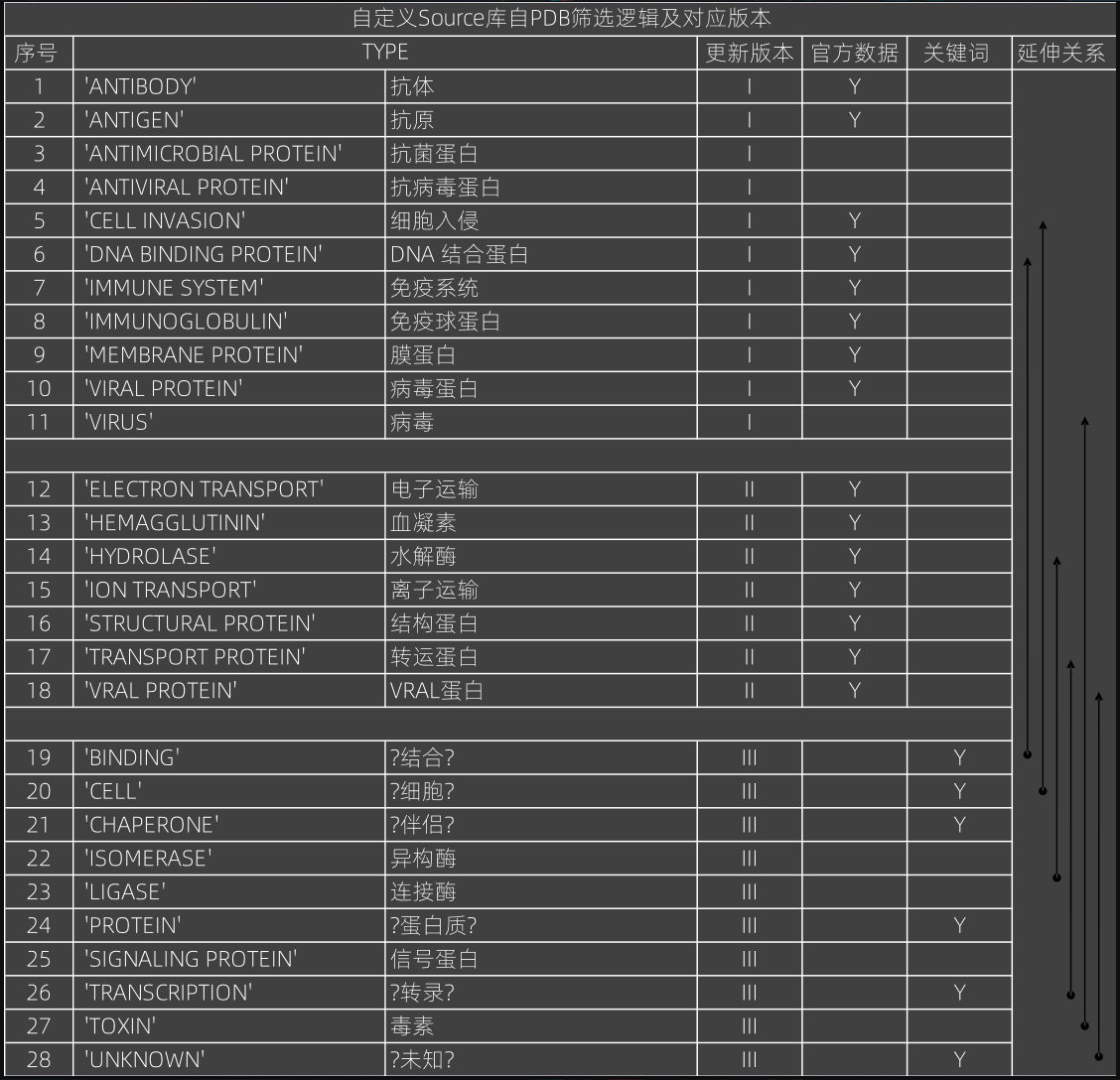
除了由最初官方数据的汇总分析外,我们先后进行了如右 I、II、III版本更新,陆续补充了一些关键词如:”?转录?”,并根据如:水解酶进而补充异构酶等之类的延伸关系。最后得到fasta+pdb文件经数据压缩后约18G,大幅度缩小了搜索范围。
对应的Source对比库也有 I [15]、II [16]、III [TODO] 版本,我们开放了部分。
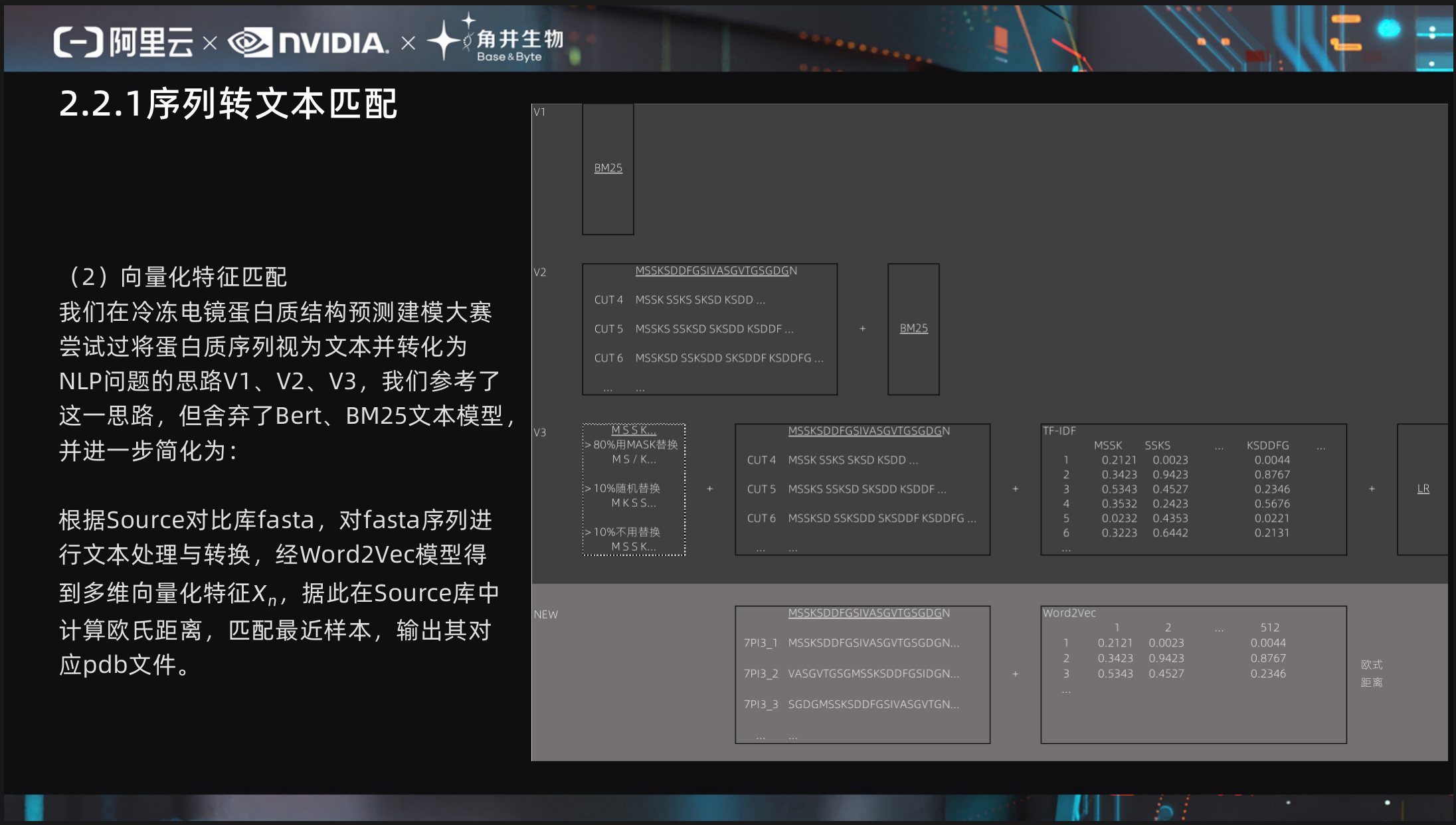
(2)向量化特征匹配
我们在冷冻电镜蛋白质结构预测建模大赛尝试过将蛋白质序列视为文本并转化为NLP问题的思路V1、V2、V3,如图:
- V1、直接走BM25文本模型;
- V2、对文本进行CUT预处理后,走BM25文本模型;
- V3、引用Bert MASK思想 + CUT预处理后,切入TF-IDF特征,导入LR模型;
我们参考了这一思路,但舍弃了Bert [17]、BM25文本模型 [18]。根据KISS原则 [19]”Keep It Simple, Stupid“,进一步简化为:根据Source对比库fasta,对fasta序列进行文本处理与转换,经Word2Vec模型 [20]得到多维向量化特征$X_n$,实际我们使用了512维,据此在Source库中计算欧氏距离 [21],匹配最近样本,输出其对应pdb文件。

2.2.2 HelixFold-Single
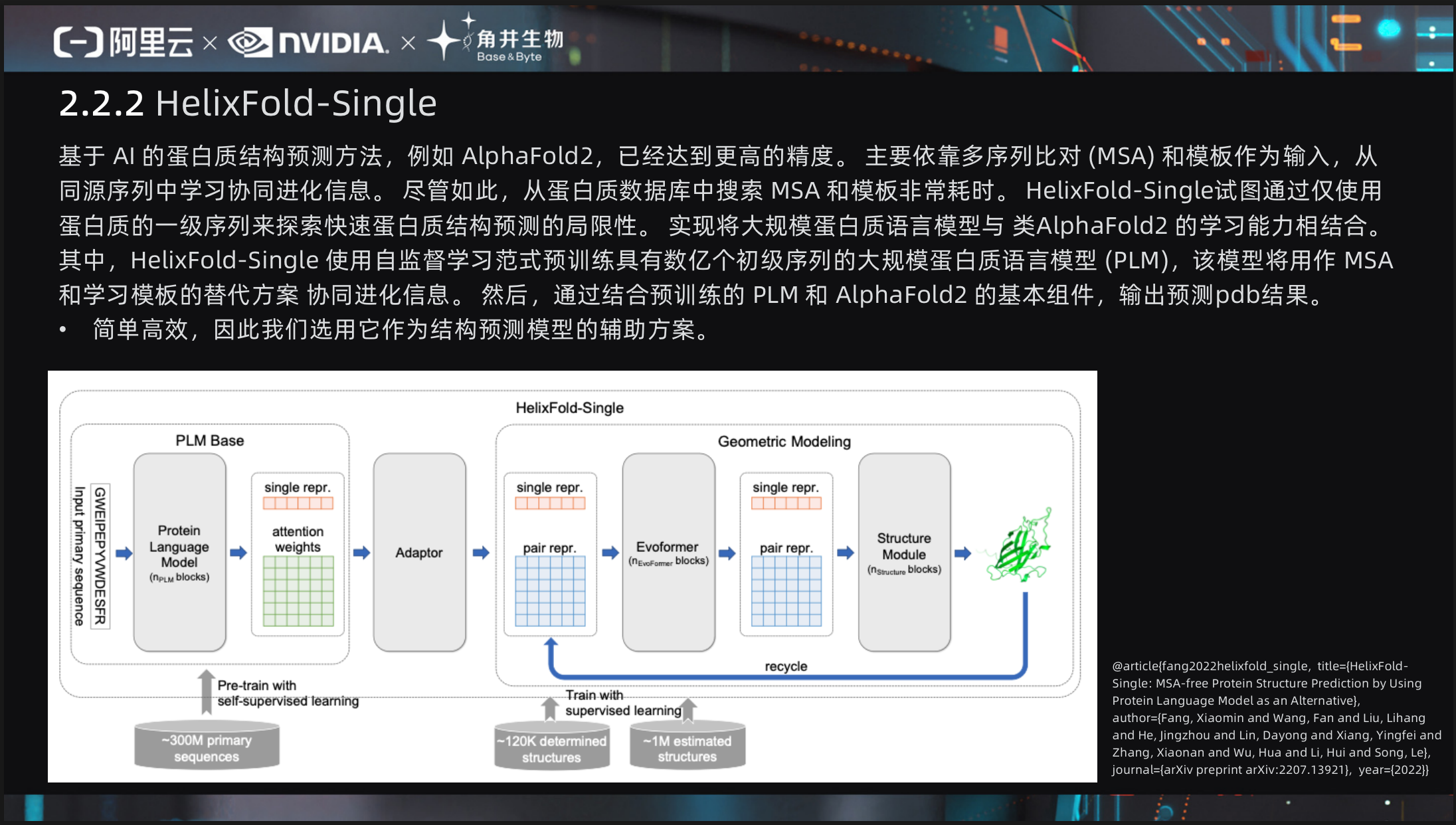
基于 AI 的蛋白质结构预测方法,例如 AlphaFold2,已经达到更高的精度。 主要依靠多序列比对 (MSA) [22]和模板作为输入,从同源序列中学习协同进化信息。 尽管如此,从蛋白质数据库中搜索 MSA 和模板非常耗时。 HelixFold-Single试图通过仅使用蛋白质的一级序列来探索快速蛋白质结构预测的局限性。 实现将大规模蛋白质语言模型与 类AlphaFold2 的学习能力相结合。 其中,HelixFold-Single 使用自监督学习范式预训练具有数亿个初级序列的大规模蛋白质语言模型 (PLM) [23],该模型将用作 MSA 和学习模板的替代方案 协同进化信息。 然后,通过结合预训练的 PLM 和 AlphaFold2 的基本组件,输出预测pdb结果。
我们注意到HelixFold-Single仅应用了蛋白质的一级序列,且也应用NLP技术,采用了大规模蛋白质语言模型/PLM作为AlphaFold2的文本化版本。其基本思想与我们类似,也是类似ESMFold [24]的大规模模型,但预训练给了更强的推理速度。
- 简单高效,因此我们选用它作为结构预测模型的辅助方案。
2.2.3 优化版HelixFold-Single
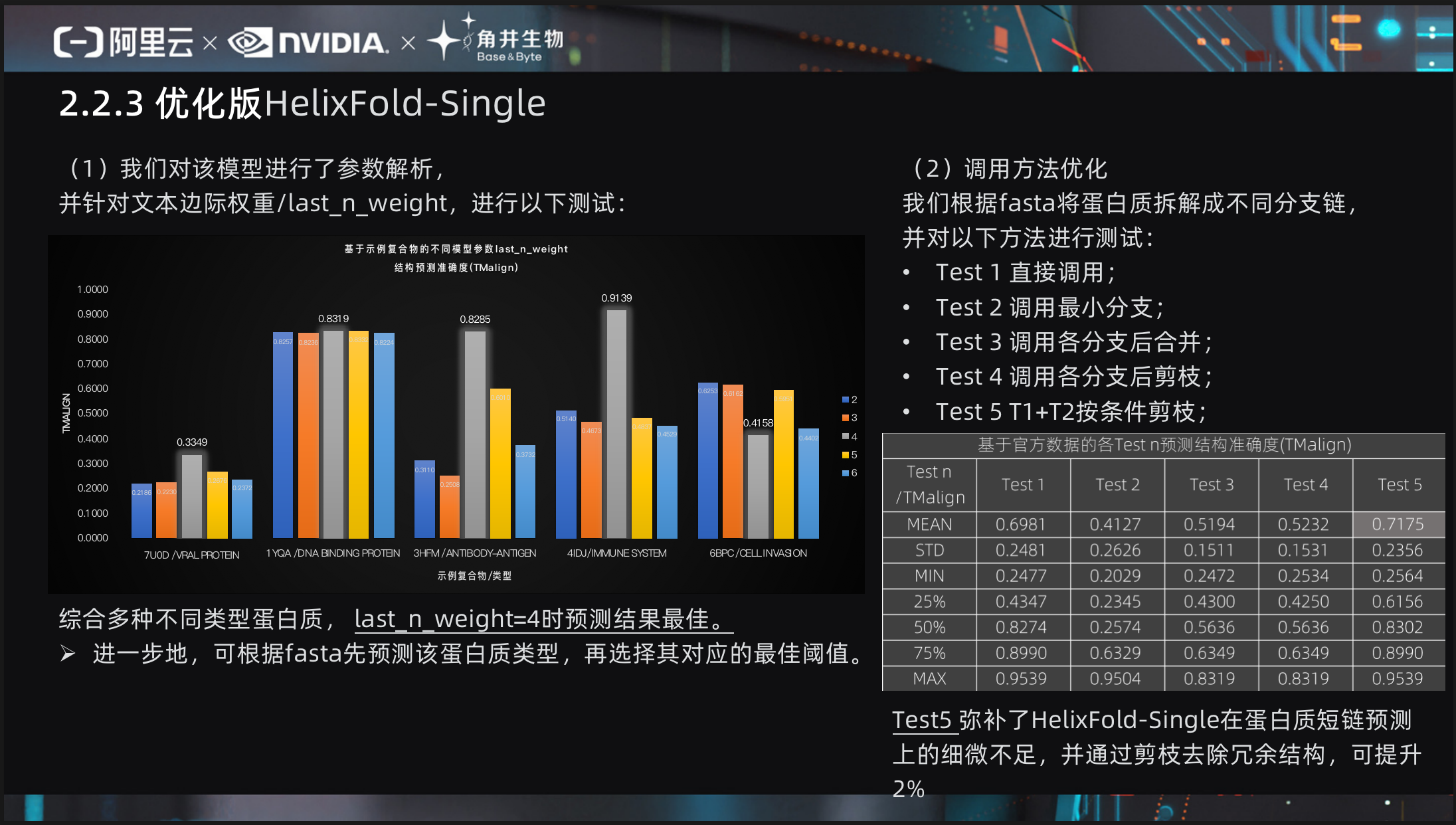
(1)我们对该模型进行了参数解析,
并针对文本边际权重/last_n_weight [25],进行如图测试:综合多种不同类型蛋白质,示例中病毒蛋白 [26]、DNA构建蛋白 [27]、抗体抗原蛋白 [28]、免疫系统蛋白 [29]、细胞侵入 [30]等, last_n_weight=4时预测结果最佳。
进一步地,如细胞侵入last_n_weight=2更佳,这样我们可根据fasta先预测该蛋白质类型,再选择其对应的最佳阈值。
(2)调用方法优化
我们根据fasta将蛋白质拆解成不同分支,或者说蛋白质链,并对以下方法进行测试:
- Test 1 直接调用;
- Test 2 调用最小分支;
- Test 3 调用各分支后合并;
- Test 4 调用各分支后剪枝;
- Test 5 T1 + T2按条件剪枝;
Test5 弥补了HelixFold-Single在蛋白质短链预测上的细微不足,并通过剪枝去除冗余结构,可提升2%!
特别说明,这里剪枝的思想来自于冷冻电镜蛋白 @nils 团队 [31]
2.3 位点预测
关于位点预测,我们主要有方法:
1、取默认值;
2、基于官方给的Baseline [32]进行优化;
3、通过ePISA [33]采集数据进行PDB匹配;
4、引入图模型 [34] GL model *;
其中,Baseline主要:
- 读取结构预测后的 pdb 文件
- 区分 pdb 文件中,分别属于抗原和抗体的部分
- 根据Hydrogen bonds/氢键 [35],Salt bridges/盐桥 [36]的定义,识别相互作用区域的位点
- 将pdb和epitope文件夹内结果保存为result.zip
实际使用,我们对方法2、方法3进行double check,以下介绍:
2.3.1 Baseline优化
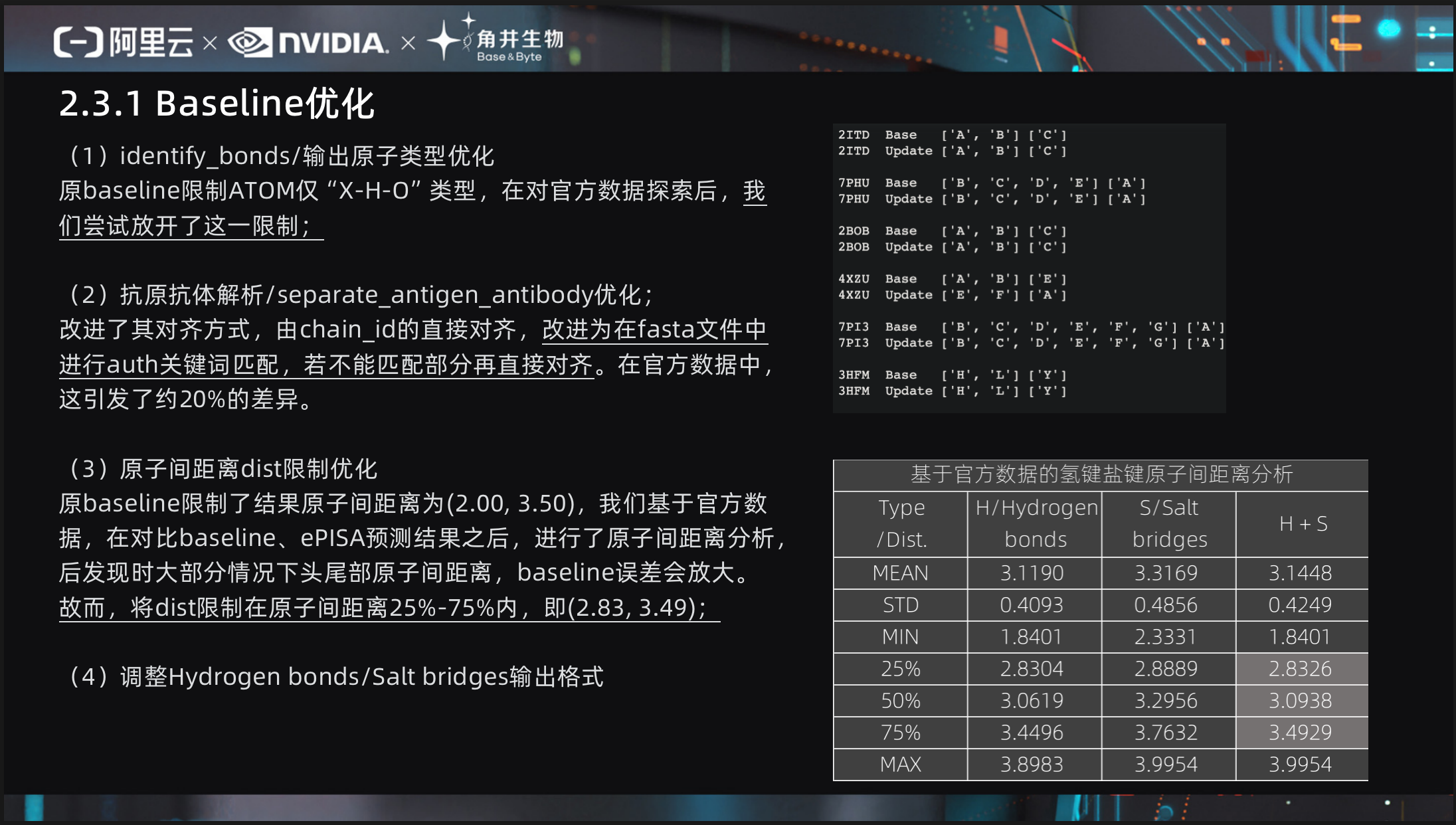
(1)identify_bonds/输出原子类型优化
原baseline限制ATOM仅“X-H-O” [37]类型,在对官方数据探索后,我们尝试放开了这一限制;
(2)抗原抗体解析/separate_antigen_antibody优化;
改进了其对齐方式,由chain_id的直接对齐,改进为在fasta文件中进行auth关键词匹配,若不能匹配部分再直接对齐。在官方数据中,这引发了约20%的差异。
- 这个bug其实很多团队都发现了,优化后可使得抗原抗体以及其链是对应的。
(3)原子间距离dist [38]限制优化
原baseline限制了结果原子间距离为(2.00, 3.50),我们基于官方数据,在对比baseline、ePISA预测结果之后,进行了原子间距离分析,后发现时大部分情况下头尾部原子间距离,baseline误差会放大。
故而,将dist限制在原子间距离25%-75% [39]内,即(2.83, 3.49);
(4)调整Hydrogen bonds/Salt bridges输出格式
2.3.2 PDBePISA
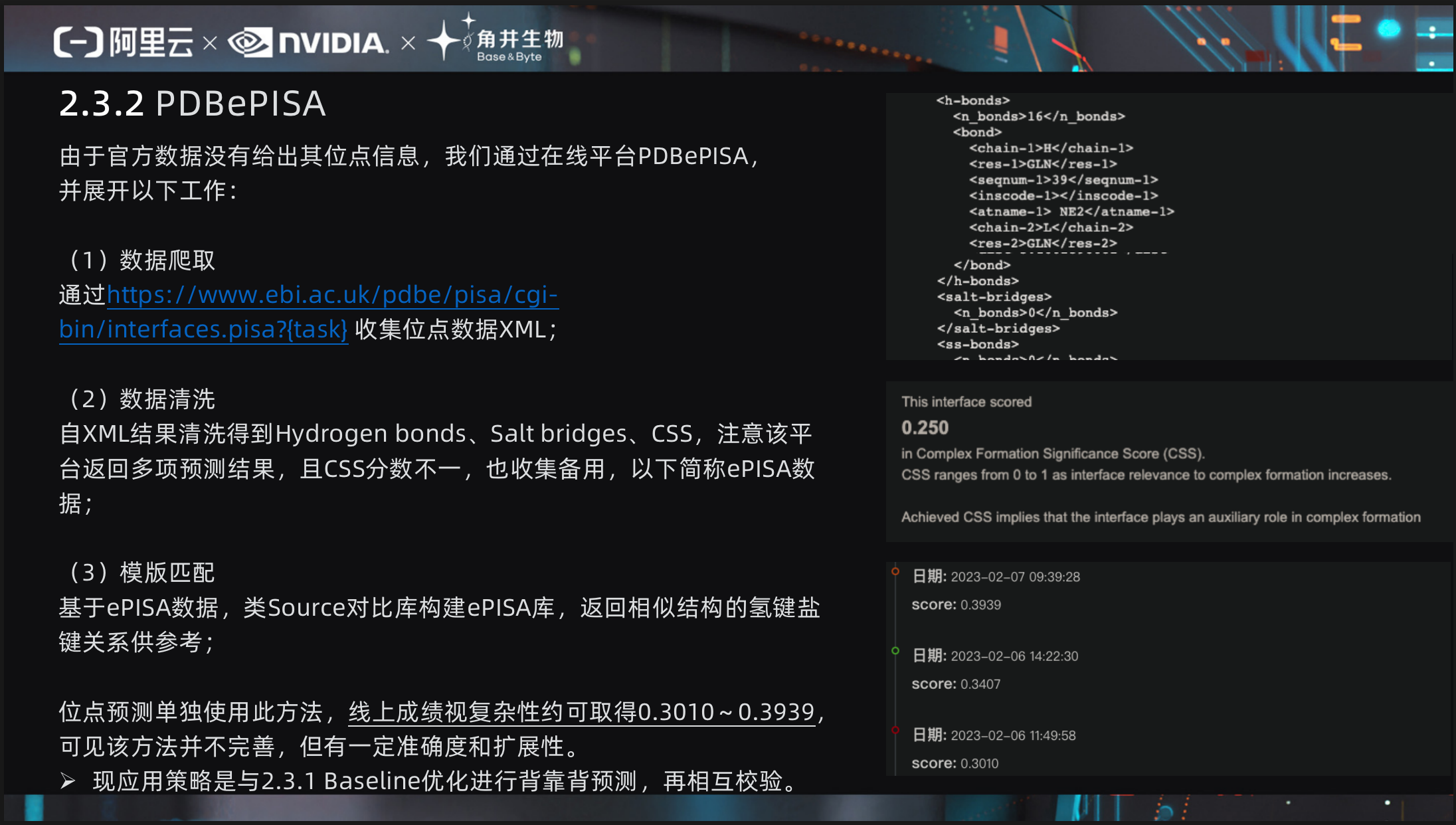
由于官方数据没有给出其位点信息,我们通过在线平台PDBePISA,并展开以下工作:
(1)数据爬取
通过https://www.ebi.ac.uk/pdbe/pisa/cgi-bin/interfaces.pisa?{task} 收集位点数据XML [40];
(2)数据清洗
自XML结果清洗得到Hydrogen bonds、Salt bridges、CSS [41],注意该平台返回多项预测结果,且CSS分数不一,也收集备用,以下简称ePISA数据;
(3)模版匹配
基于ePISA数据,类Source对比库构建ePISA库,返回相似结构的氢键、盐桥关系供参考;
位点预测单独使用此方法,线上成绩视复杂性约可取得0.3010~0.3939,可见该方法并不完善,但有一定准确度和扩展性。
这里我们对位点预测部分较为严谨,希望尽可能预测准确,故而:
- 现应用策略是与2.3.1 Baseline优化进行背靠背预测,再相互校验。
如果改进此应用策略,或许整体模型效果会更好。








:数据链路层协议)







)


