前记
目前,已经有很多种玉米miRNA结合位点预测工具可供选择,以下几种比较常用:
1、psRNATarget:该工具是由华盛顿州立大学开发的,可以用来预测植物miRNA和靶基因之间的相互作用。用户可以使用该工具来预测玉米miRNA和结合位点以及靶基因,同时可以输出预测结果。
psRNATarget: A Plant Small RNA Target Analysis Server (2017 Update) (zhaolab.org)
https://www.zhaolab.org/psRNATarget/home 2、miRanda:该工具由麻省理工学院开发,可以用来预测miRNA和靶基因之间的相互作用。用户可以使用该工具来预测玉米miRNA和结合位点以及靶基因,同时可以输出预测结果。
miRanda (microRNA.org) | miRToolsGallery
http://mirtoolsgallery.tech/mirtoolsgallery/node/1055 3、TargetFinder:该工具由威斯康星大学开发,可以用来预测miRNA和靶基因之间的相互作用。用户可以使用该工具来预测玉米miRNA和结合位点以及靶基因,同时可以输出预测结果。
GitHub - carringtonlab/TargetFinder: Plant small RNA target prediction tool
https://github.com/carringtonlab/TargetFinder 这些工具使用方法大多相似,用户需要输入miRNA序列和靶基因序列,然后进行计算、分析和输出预测结果。用户可以根据自己的需求和使用习惯来选择最适合自己的工具。
本文以psRNATarget为例,演示如何预测基因的miRNA结合区域。
一、psRNATarget页面介绍
打开官网后,如下所示:
psRNATarget是一个在线工具,用于预测植物小RNA和宿主基因的互作关系。它基于植物小RNA和宿主基因的序列数据,使用一个准确的算法进行分析和预测。用户可以输入小RNA和宿主基因的序列,或者直接上传相应的FASTA文件。该页面会显示预测结果,包括小RNA的靶向位点,以及宿主基因的功能注释和调控网络。用户还可以进行进一步分析,例如查看小RNA和宿主基因的共表达网络,以及预测小RNA的调控效应。psRNATarget页面简单易用,对于研究植物非编码RNA的功能与调控机制具有重要意义。

二、 查询玉米基因的miRNA靶点
首先,点击主页面的菜单栏Analysis选项,进入以下界面:
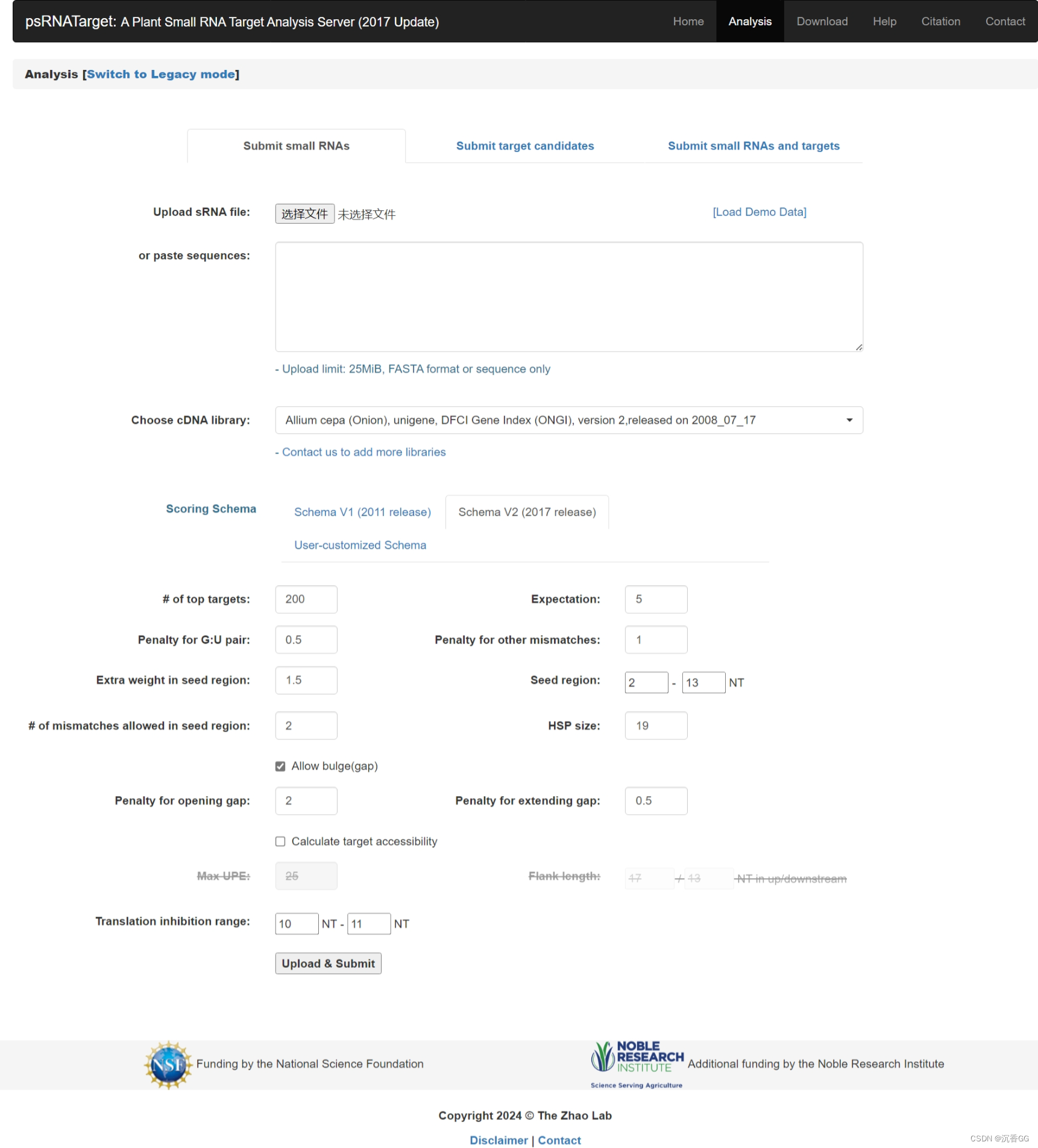
然后,选择Summit target candidates,如下所示:
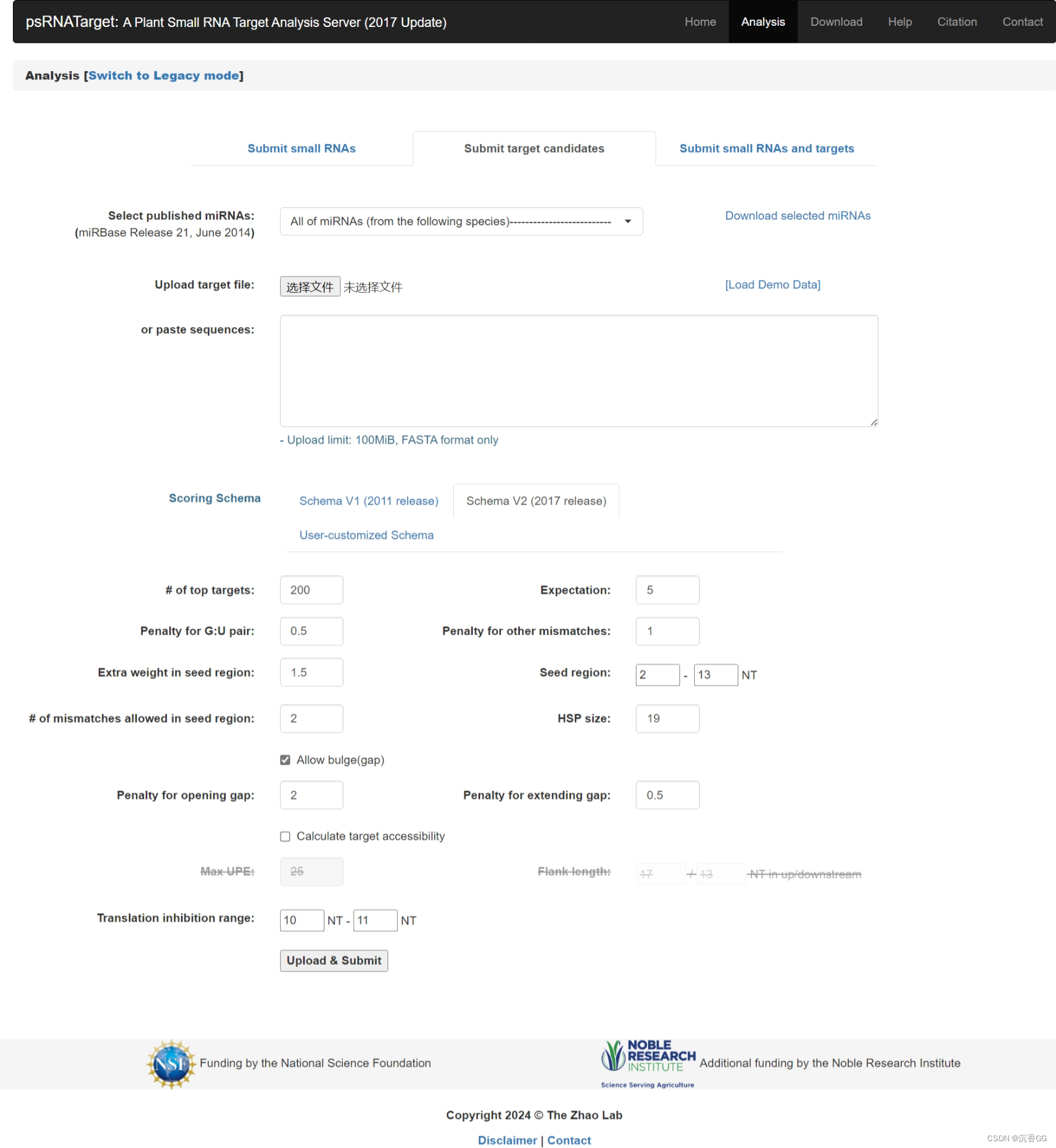
这里,我们设置好需要查询的miRNA数据库(玉米),在框内粘贴好自己的基因序列(使用fasta格式)。如下所示:
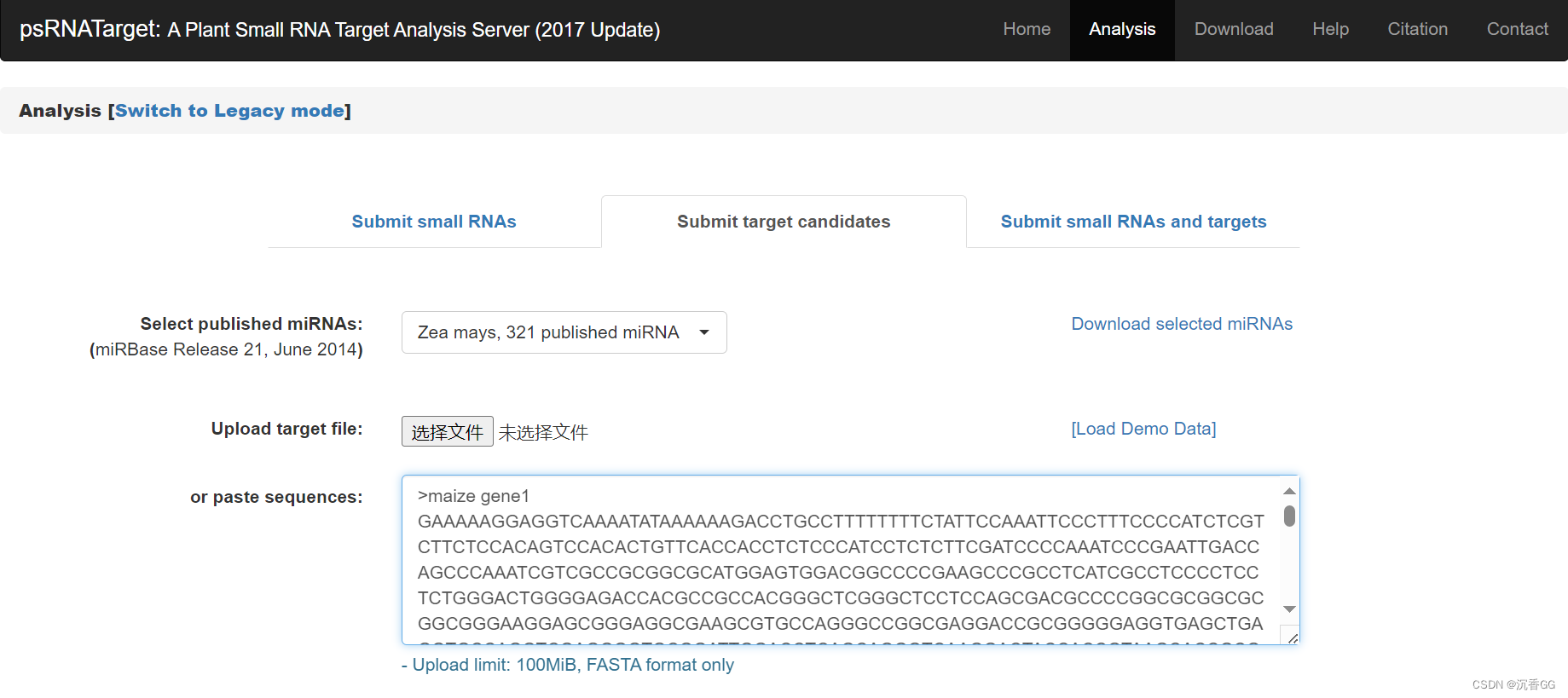
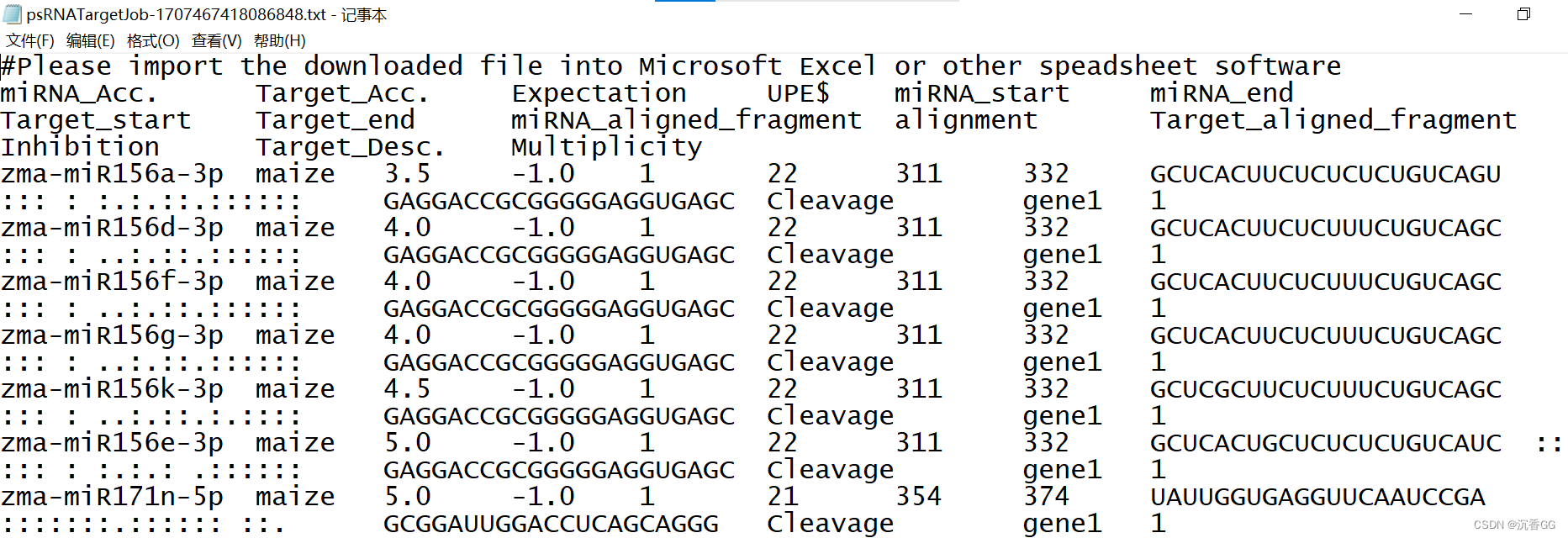
设置完成后,点击下方提交按钮即可。几秒后,输出预测的结果: 
可以看到miR156可能靶向我们的目的基因,但这些结果还需要后续试验验证。
三、预测结果的导出
在结果页面中,点击左上方的download选项,即可下载所有的结果。如下所示:
后记
以上是目的基因miRNA的靶点预测过程,使用方法较为简单,供大家参考。
2024.2.9----CXGG
千里之行,始于足下。
(cp --help)(各选项参数用法解释))















)


