

GO和KEGG富集分析是我们在筛选出差异表达基因之后,都会去做的套路性分析。然鹅……我相信,总有那么一些“倒霉孩子”会遇到跟我一样的窘境吧,好不容易筛选出来的差异基因,尝试了DAVID(https://david.ncifcrf.gov/)、Metascape(https://metascape.org/)、g:Profiler(https://biit.cs.ut.ee/gprofiler/)等之后,仍然富集不出来通路,这真是个令人悲伤的故事嘞~

就在我万念俱灰的时候,我神奇的发现了一个网站,居然富集到了好几条通路哦,心情顿时开朗许多~~

现在,就把这个最强大的基因富集分析在线工具分享给大家啦~揭开它神秘面纱的时候到了,它就是——KOBAS!!!
KOBAS(KEGG Orthology Based Annotation System),网址:http://kobas.cbi.pku.edu.cn,是由北京大学魏文丽课题组开发的数据库,主要功能是用于基因/蛋白质功能注释和功能富集。随着数据量不断增加,KOBAS至今为止共经历了3次升级,除了1.0版本的KEGG代谢通路之外,2.0版本增加了PID Curated,PID BioCarta,PID Reactome,BioCyc,Reactome和Panther等数据库,除此之外还增加了疾病的查询,使得KOBAS的功能更加强大。而3.0版本更是增加了lncRNA的鉴定、注释以及富集功能,还建立GSEA,GSA和 PADOG等功能。
KOBAS的功能最主要分为两个部分,Annotate(注释)和Enrichment(富集)。
接下来我们就以“Annotate”为例来体验看看如何利用KOBAS做GO和KEGG的富集分析吧!
点击“Annotate”选项,进入注释界面。
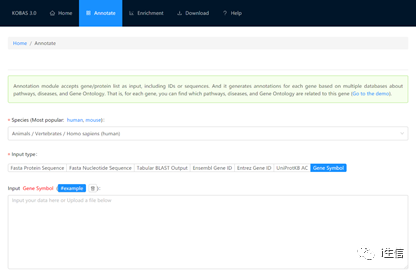
“Species”为物种,可根据自己测序组织来源进行选择,如人,大鼠,小鼠等。
“Input Type”为输入格式/类型,支持如图Fasta Protein Sequence,Gene Symbol等七种格式,可根据自己的输入数据进行选择。
输入数据后,点击“Run”进行提交,等待数秒即可出结果。
得到注释结果,如下图所示,点击“Detail”可查看基因注释的Pathway通路、疾病及GO功能分类。注释结果下方还提供链接进行结果下载哦。
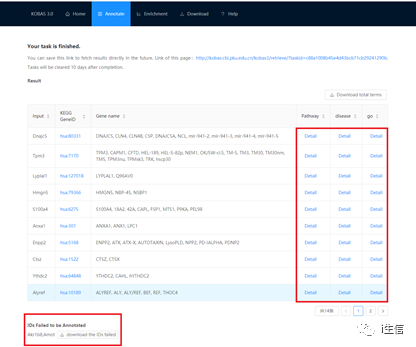
点击第二列“KEGG Gene ID”可以直接链接到KEGG数据库查看具体信息。
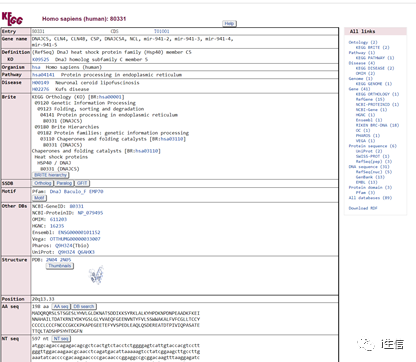
点击第三列“Disease”中“Detail”注释结果如下所示,可具体了解与差异表达基因相关的疾病信息哦~
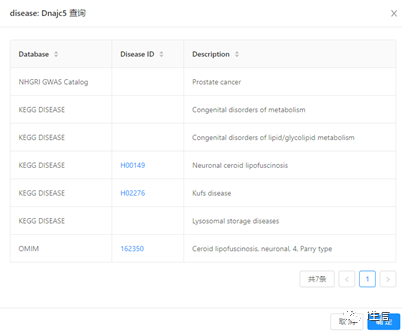
“Gene-list Enrichment”部分,“Input Type”, “Species”及“Input”与注释时基本一致。
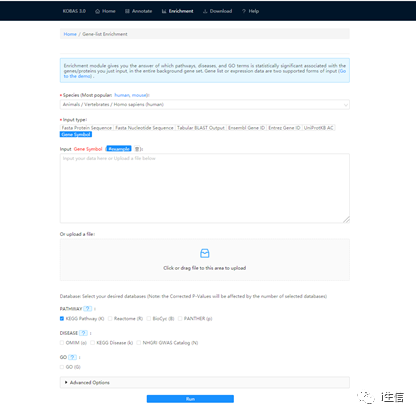
“Datebase”中可选择自己感兴趣的数据库进行分析。
“Advance Options”中可自行选择参数,包括背景基因、统计学方法及校正方法等。
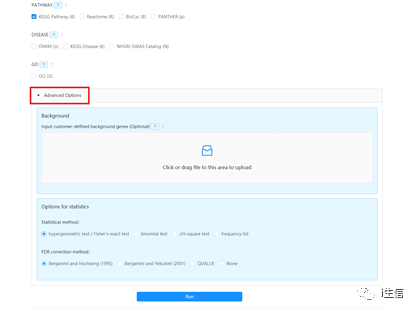
与“Annotate”一样,设置好所有参数后,点击“Run”,等待数秒即可得到分析结果哦,且结果都可进行下载,方便又快捷。
此外,KOBAS除在线使用外,还可将下载到本地,具体可点击官网菜单栏“Download”进行详细了解哦,在此不做详细介绍啦。
总之,KOBAS不仅仅可以得到GO和KEGG富集分析结果,还可以了解与之相关的疾病信息。学习了这么多,赶紧把你手中筛选到的差异基因操作分析起来吧,说不定马上就会得到意想不到的结果哦~~
注:此推文未经许可禁止转载!
阅读推荐:
工具篇|MNDR:递“瓜”小能手,扒一扒ncRNA与疾病之间的那些事
工具篇 | TransCirc:一个预测分析circRNA翻译的数据库
工具篇|一站搞定多种形式的韦恩分析可视化,需要哪个选哪个
工具篇|最新出炉的国产m6A修饰精准预测数据库m6A-Atlas
工具篇|神器一出,作图无忧:一个完爆R语言的作图网站




















