0. 简介
GMM和Kmeans一样也属于聚类,其算法训练流程也十分相似,Kmeans可认为是“硬聚类”,GMM是“软聚类”。
给定数据集X,Kmeans算法流程是这样的----- a 初始化:随机初始k个中心(即k个点,记为μ);b 矫正数据归属:计算X中每个点与k个中心的距离,并将其归为相距最近的那个中心;c 矫正中心:计算每个中心(共k个)所有点的均值,并将其更新为中心值;d 完成整体训练:循环b和c,直到聚类到“足够好”。
GMM算法流程和Kmeans基本一致,区别在于:a 除了初始化k个中心(μ)外,每个中心还对应一个协方差矩阵(Σ)和混合概率(π),其中μ代表高斯分布的中心,Σ代表高斯分布形状,π代表高斯函数值的大小;b 矫正数据归属,GMM中每个数据点并不完全归属某个中心,而是归属每个中心,只是归属的概率不同;c 矫正中心,每个中心矫正更新时考虑数据集X中的所有点,而非某一部分数据点。
以下使用鸢尾花数据集按照a~d的流程解析GMM;导入鸢尾花数据集如下;
from sklearn import datasets
import numpy as npiris = datasets.load_iris()
X = iris.data
N, D = X.shape
display(X.shape, X[:10])
(150, 4)
array([[5.1, 3.5, 1.4, 0.2],[4.9, 3. , 1.4, 0.2],[4.7, 3.2, 1.3, 0.2],[4.6, 3.1, 1.5, 0.2],[5. , 3.6, 1.4, 0.2],[5.4, 3.9, 1.7, 0.4],[4.6, 3.4, 1.4, 0.3],[5. , 3.4, 1.5, 0.2],[4.4, 2.9, 1.4, 0.2],[4.9, 3.1, 1.5, 0.1]])即鸢尾花数据集是一个150行4列的矩阵。
1. 初始化
定义聚类数量为3类,每一类都初始化一个中心μ、一个协方差矩阵Σ和混合概率π;
mus = X[np.random.choice(X.shape[0], 3, replace=False)]
covs = [np.identity(4) for i in range(3)]
pis = [1/3] * 32. 矫正数据归属
普通高斯概率函数(只有一个中心)如下,其中D是数据维度,此处D=4;
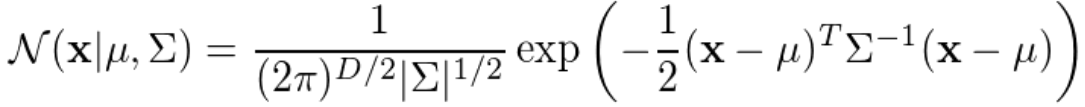
表示数据点x归属该中心(μ、Σ)的概率,代码如下;
def gaussian(X, mu, cov):diff = X - mureturn 1 / ((2 * np.pi) ** (D / 2) * np.linalg.det(cov) ** 0.5) * np.exp(-0.5 * np.dot(np.dot(diff, np.linalg.inv(cov)), diff))当用混合高斯函数(即多个中心)时,表示一个数据点n归属中心k(μ_k、Σ_k)的概率函数如下,其中k表示第k个中心,此处总共有3个中心,即K=3;
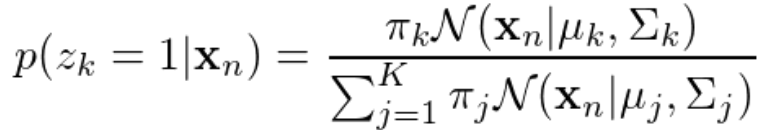
将上式定义为γ_z_nk,即
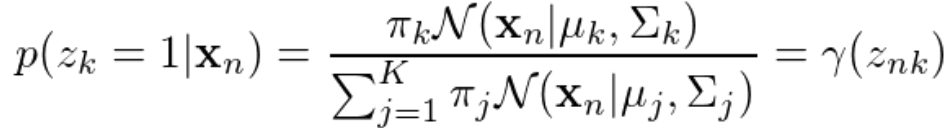
代码如下:
gammas = []
for mu_, cov_, pi_ in zip(mus, covs, pis):# loop each centergamma_ = [[pi_ * gaussian(x_, mu_, cov_)] for x_ in X]# loop each pointgammas.append(gamma_)
gammas = np.array(gammas)
gamma_total = gammas.sum(0)
gammas /= gamma_total3. 矫正中心
根据2.中的gammas值,更新μ、Σ和π值,公式如下,其中N表示数据总个数,此处N=150;
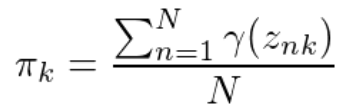
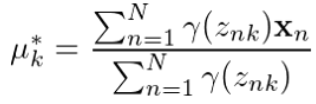
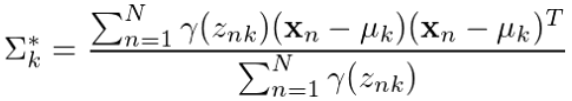
代码如下;
mus, covs, pis = [], [], []
for gamma_ in gammas:#loop each centergamma_sum = gamma_.sum()pi_ = gamma_sum / Nmu_ = (gamma_ * X).sum(0) / gamma_sumcov_ = []for x_, gamma_i in zip(X, gamma_):diff = (x_ - mu_).reshape(-1, 1)cov_.append(gamma_i * np.dot(diff, diff.T))cov_ = np.sum(cov_, axis=0) / gamma_sumpis.append(pi_)mus.append(mu_)covs.append(cov_)4. 完成整体训练
将2.~3.作为一个循环单元,写成一个函数;
def train_step(X, mus, covs, pis):gammas = []for mu_, cov_, pi_ in zip(mus, covs, pis):# loop each centergamma_ = [[pi_ * gaussian(x_, mu_, cov_)] for x_ in X]# loop each pointgammas.append(gamma_)gammas = np.array(gammas)gamma_total = gammas.sum(0)gammas /= gamma_totalmus, covs, pis = [], [], []for gamma_ in gammas:#loop each centergamma_sum = gamma_.sum()pi_ = gamma_sum / Nmu_ = (gamma_ * X).sum(0) / gamma_sumcov_ = []for x_, gamma_i in zip(X, gamma_):diff = (x_ - mu_).reshape(-1, 1)cov_.append(gamma_i * np.dot(diff, diff.T))cov_ = np.sum(cov_, axis=0) / gamma_sumpis.append(pi_)mus.append(mu_)covs.append(cov_)return mus, covs, pis训练50次;
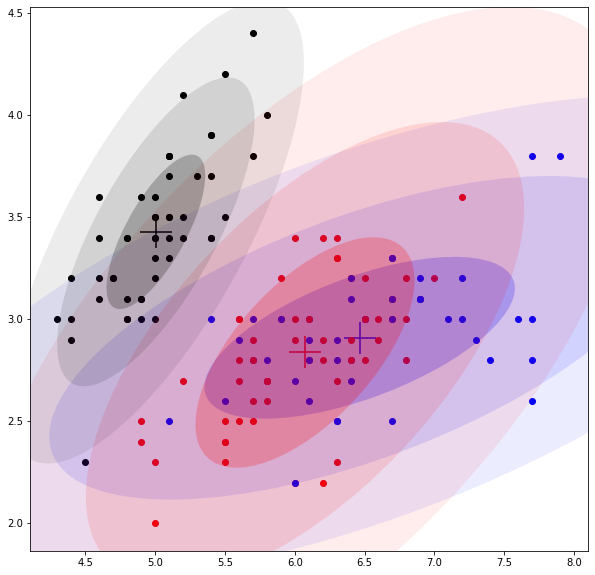
for _ in range(50):mus, covs, pis = train_step(X, mus, covs, pis)训练完成后,会得到3个中心,可计算每个点归属这三个中心的概率(即γ_z_nk,第n个点归属第k个中心的概率),并将其归属于概率最大的那个中心;因为数据集是4维,无法可视化,仅选择前两维度进行可视化展示如下;
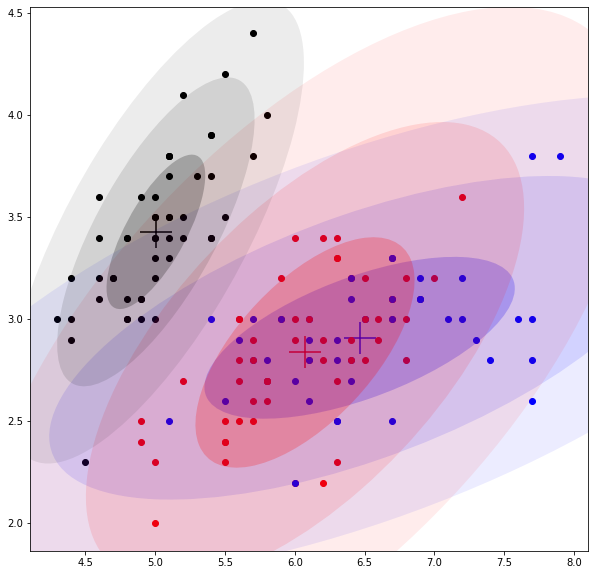
整个训练过程的动态图如下;
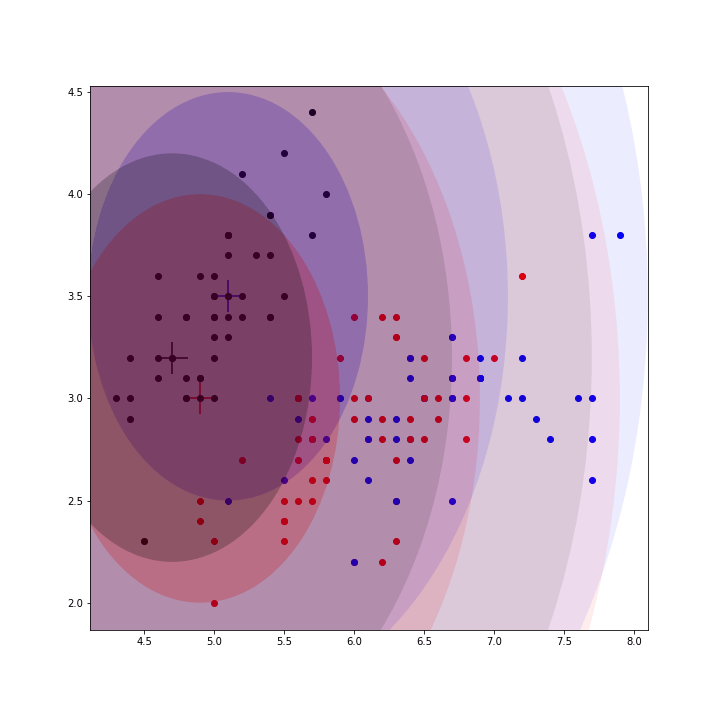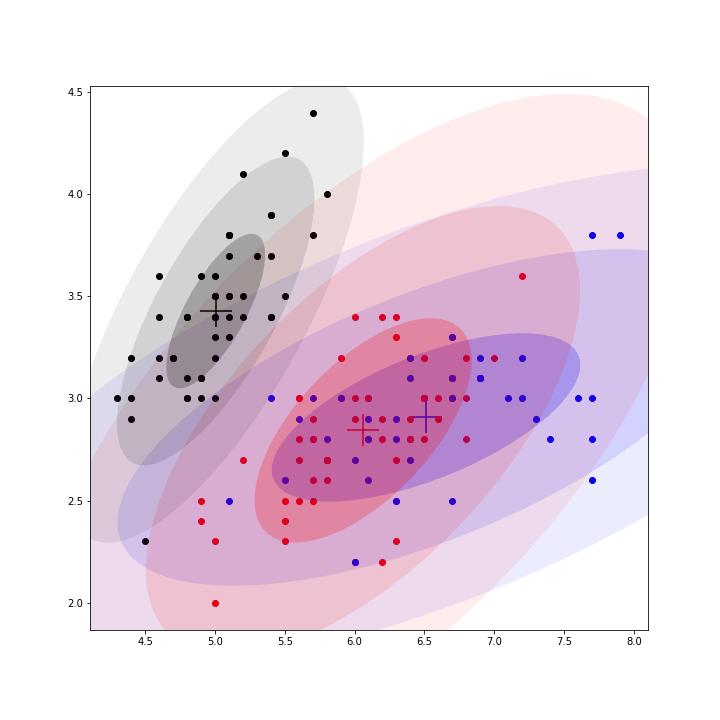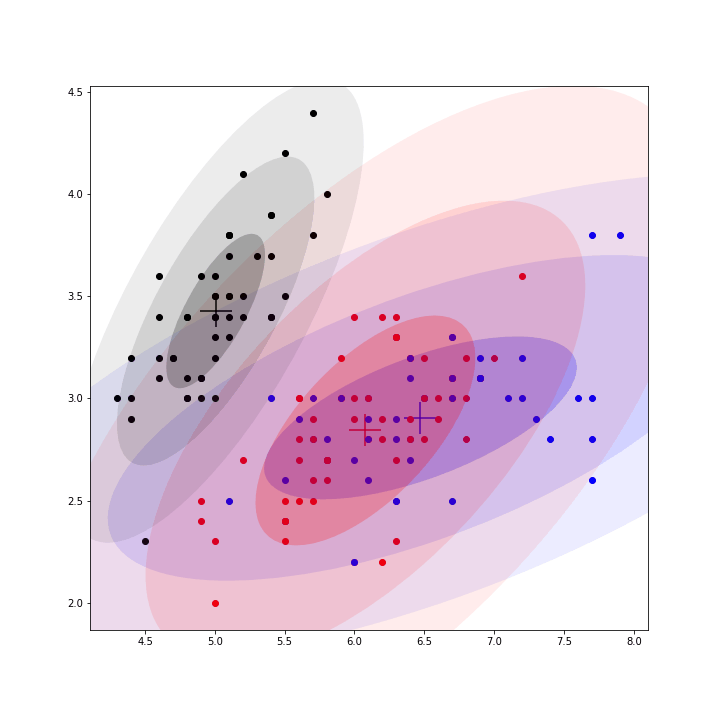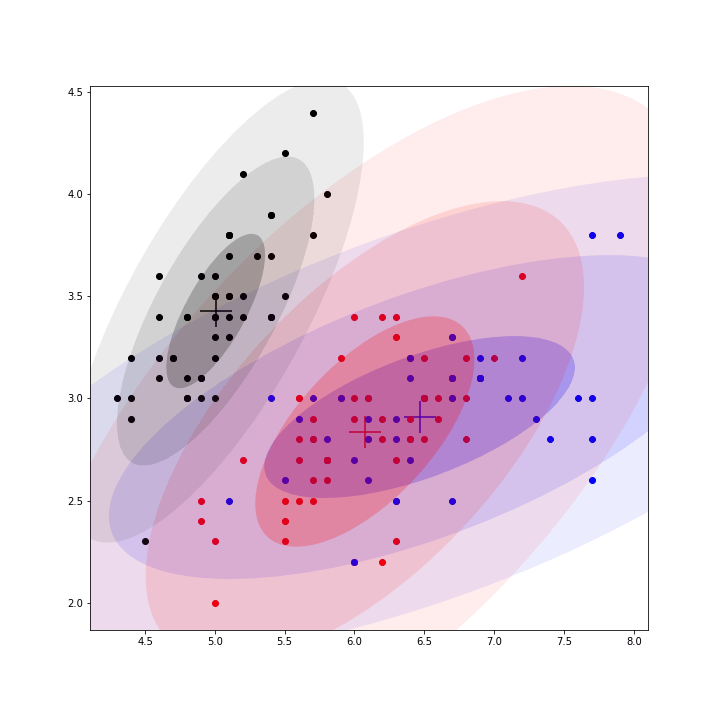
完整代码如下;
from sklearn import datasets
import numpy as np
import matplotlib.pyplot as pltiris = datasets.load_iris()
X = iris.data
N, D = X.shapemus = X[np.random.choice(X.shape[0], 3, replace=False)]
covs = [np.identity(4) for i in range(3)]
pis = [1/3] * 3def gaussian(X, mu, cov):diff = X - mureturn 1 / ((2 * np.pi) ** (D / 2) * np.linalg.det(cov) ** 0.5) * np.exp(-0.5 * np.dot(np.dot(diff, np.linalg.inv(cov)), diff))def get_likelihood(gamma_total):return np.log(gamma_total).sum()def train_step(X, mus, covs, pis):gammas = []for mu_, cov_, pi_ in zip(mus, covs, pis):# loop each centergamma_ = [[pi_ * gaussian(x_, mu_, cov_)] for x_ in X]# loop each pointgammas.append(gamma_)gammas = np.array(gammas)gamma_total = gammas.sum(0)gammas /= gamma_totalmus, covs, pis = [], [], []for gamma_ in gammas:#loop each centergamma_sum = gamma_.sum()pi_ = gamma_sum / Nmu_ = (gamma_ * X).sum(0) / gamma_sumcov_ = []for x_, gamma_i in zip(X, gamma_):diff = (x_ - mu_).reshape(-1, 1)cov_.append(gamma_i * np.dot(diff, diff.T))cov_ = np.sum(cov_, axis=0) / gamma_sumpis.append(pi_)mus.append(mu_)covs.append(cov_)return mus, covs, pis, gamma_totallog_LL = []
for _ in range(50):mus, covs, pis, gamma_total = train_step(X, mus, covs, pis)log_LL.append(get_likelihood(gamma_total))
plt.plot(log_LL)
plt.grid()![[html] 对于rtl网站的适配有哪些方案?](http://pic.xiahunao.cn/[html] 对于rtl网站的适配有哪些方案?)



![[html] 如何通过表单下载文件?](http://pic.xiahunao.cn/[html] 如何通过表单下载文件?)

)
)

微信号对接)

朗读)

编写一个vue页面用于复制淘口令)

)
使用IMoniker)


