
首先,小编先给大家介绍一些关于组装的历史。大家肯定也知道,这个组装技术的发展是依赖于测序技术的。首先在一代测序的时候,测序数据量较少,成本较高,人们对于组装的结首先,小编先给大家介绍一些关于组装的历史。大家肯定也知道,这个组装技术的发展是依赖于测果预期也比较低,弄到contig水平就可以啦,还有就是这个组装的物种基因组都偏小。因此那个时候的组装软件都是基于overlap的。然后呢,二代测序技术来了,数据量超高,成本也便宜啦。科研工作者就想啊,现在都二代了,这个组装的预期得提高啊,得与时俱进啊。但是基于overlap的组装不了太长,而且超级慢,又加之,二代数据量太多,overlap扛不住啊。因此有人就开发出了基于图论的组装算法。然后呢,三代测序技术这两年出来了,科学家一看,我x,这么长的read,那还搞毛图论的算法,直接简单粗暴点,用overlap多简单,于是基于overlap的软件又火了,去年一个三代基于图论的软件发表,将基于图论的组装再次放在了风口浪尖。
大家好,我是生信人小编XIXI,与青壬同学遥相呼应地,坐标魔都西南……平时做项目总会遇到一些很有意思的文章或者算法,或者一些很有用的工具,会有拍案叫绝的冲动(当然更多时候有想摔桌子的冲动),所以呢希望能通过这个平台和大家一起分享。
最近有在看一些关于从头拼接(de novo)相关的文章。De novo的方法大部分是当基因组、转录组或者蛋白组信息缺失严重或者想要研究修饰的时候使用,它可以不用借助任何参考组直接进行推测拼接,但相对来说准确度会比较低一些。
其实不难发现,大部分de novo的文章都是基于图论分析,比如大部分基因组和转录组de novo;蛋白组的质谱数据分析属于“看图说话”,以前的很多工具也是基于图论,最近有出来很多基于机器学习的工具,运行速度也有大幅提升,如果大家感兴趣后边也可以给大家讲讲。
最近想使用SOAPdenovo-Trans来进行RNA-seq的de novo分析
所以对它的原理及运行过程进行了一下简单摸索。在转录组方面,如果是测序比较完全的生物,大多数人是会选择将测序的read直接匹配到参考基因组上边的(还记得大明湖畔的Tophat和cufflinks么);但是如果是一些参考基因组缺失的或者基因组非常庞大的物种,de novo的方法则可以很好地解决这个问题。
De Bruijn graph (DBG)可以说基本是大部分组装方法的核心所在,有很大一部分的转录组拼接软件(Oases,SOAPdenovo-Trans等)都是依赖于这个算法的。这里给大家做一个比较通俗的介绍。
比如我们手上有真实的RNA序列AUCGAAUCCCGAA,通过 RNA-seq可以得到的6条read:

那怎么从这6个小片段推导出它真实的序列信息呢?(如果你一眼看出来了,请原谅我宽容地排列整齐……)
首先,假如我们设定K-mers为3-mers,也就是说我们可以将所有的read都切割3个碱基长度的子序列,如下:

接着,我们可以将每个子序列都可以看成一个结点,每个read中的子序列结点可以依次用箭头连接,依次建立这样一张有向图:

最后一张即为最终的de Bruijn graph (DBG)。那么怎么通过这张图得到相应的序列呢?
学过图论的旁友们大概可以会心一笑,那就是解欧拉路径(Eulerian Tour)!也就是在最后这张图里使得每条路径被经过且仅经过一次。这个例子里可以很简单的可以看出来(真实的案例里大约看的出来算我输……):
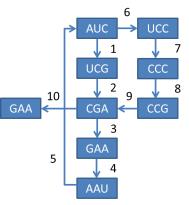
串起来就可以得到:AUCGAAUCCCGAA,也就是我们最开始的RNA序列了!具体解欧拉途径的算法图论中应该会有比较多的讨论,有兴趣的大神可以亲手实现一下。
在该算法中K的选择是有一定讲究的,一般来说K小于等于最短read的长度,如果太短拼接效果又会比较差。SOAPdenovo-Trans中一般会设定~35作为K值。
De Bruijn graph的组装方法相对时间复杂度比较低,为O(min(N,G)),G=genome length,N= total length of reads,与重叠方法的拼接过程相比可以大大节约时间成本;但它存在的问题是很可能你得到的路径和你输入的read是不匹配的,另外对于重复序列的处理也不是很棒。
更多套路,生信分析需求,请加微信:13895744602
欢迎关注生信人
TCGA | 小工具 | 数据库 |组装| 注释 | 基因家族 | Pvalue
基因预测 |bestorf | sci | NAR | 在线工具 | 生存分析 | 热图
生信不死 | 初学者 | circRNA | 一箭画心| 十二生肖 | circos
舞台|基因组 | 黄金测序 | 套路 | 杂谈组装 | 进化 | 测序简史






- 完成新闻页面)













