molUP是一个VMD扩展,提供了一个简单的方式来加载和保存高斯文件,并分析相关的结果。
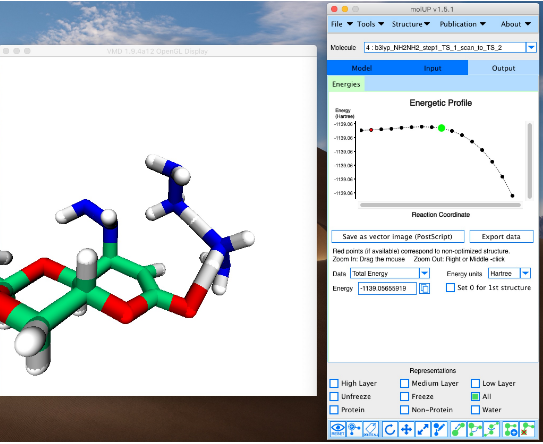
molUP为VMD提供了一个图形界面,用户可以加载和保存高斯文件格式的化学结构。这个扩展包括一组工具来设置高斯支持的任何计算,包括ONIOM通过互动图分析能量;生动的振动频率;画出与这些频率相关的向量;修改键、角和二面体;并收集关于所用方法的文献信息。
主要特点:
加载高斯输入(.com)和输出文件(.log)上的VMD;
分配ONIOM层(High-, Medium-, and Low-level);
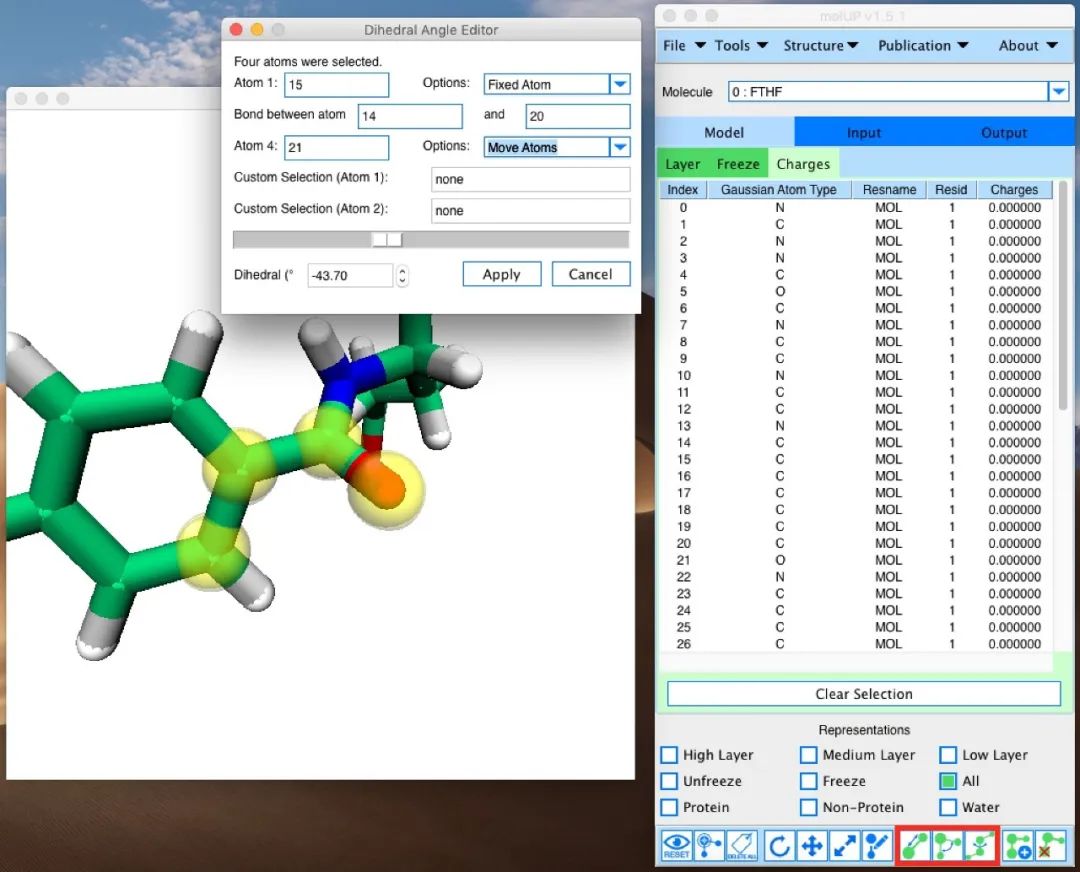
在几何优化期间选择自由和固定原子(冻结状态Freeze status);
分析和编辑原子电荷;
使用高斯输入部分设置新的高斯计算;
跨反应坐标进行负载扫描,并绘制结构能量图;
加载高斯频率计算,动画振动模式,并显示各自的向量;
展示快速有用的表示法,例如蛋白质、不同的离子层和固定的原子;
结构操控:调整键长、角度幅度、二面角扭转;
根据计算中采用的方法和功能类型,提供完整的参考书目列表;
添加新的原子和分子碎片;
移除新原子;
除其他外。
软件官网:
https://biosim.pt/molup/
源代码:
https://github.com/BioSIM-Research-Group/molUP
联系作者:hfernandes@med.up.pt
引用:
S. Fernandes, H., M.J. Ramos, and N. M. F. S. A. Cerqueira, molUP: A VMD plugin to handle QM and ONIOM calculations using the gaussian software. Journal of Computational Chemistry DOI:http://doi.org/10.1002/jcc.25189
安装最低要求
操作系统:macOS、Linux和Windows
可视化分子动力学(VMD) 1.9.3或更高版本
自动安装
可以通过vmdStore快速安装molUP.
vmdStore:
https://github.com/BioSIM-Research-Group/vmdStore
https://sourceforge.net/projects/vmdstore/
手动安装
手动安装molUP:
下载软件包。
将molUP目录复制到计算机上的某个位置。
复制“install.txt”文件中的文本并粘贴到环境变量文件:.vmdrc文件(macOS和Linux)或vmd.rc文件(Windows)。
(该文件位于主目录中)
将字符串“$::vmdStorePath/plugins/molUP”替换为molUP文件安装位置。
保存.vmdrc或vmd.rc文件。
重启VMD。

(可选)从bash运行molUP
molUP现在支持直接从命令行加载高斯文件(适用于macOS和Linux)。
教程:
将下面一行添加到的.bashrc (Linux)或者.bash_profile (macOS)文件。
(该文件位于主目录中)
编辑"path of the bashScript.tcl file"字段,该字段要写成位于molUP文件安装位置中文件“molUP/lib/bashScript.tcl”的路径:
alias molUP='vmd -e "path of the bashScript.tcl file" -args'从命令行调用molUP:
使用标准程序打开高斯文件:
molUP "Gaussian file"打开高斯输出文件,读取所有可用的结构:
molUP "Gaussian file" -all本公众号不定期发布有关DFT计算相关内容,主题多变且不固定。
欢迎分享本公众号推送,将教程与经验传播给需要的人。
如对教程内容有疑问,或者有需要咨询,可后台留言
 - Java)

——rtsp收流(tcp方式))






)
![[DASCTF 2023 0X401七月暑期挑战赛] web刷题记录](http://pic.xiahunao.cn/[DASCTF 2023 0X401七月暑期挑战赛] web刷题记录)

)




;Spring控制反转(IOC);解释一下Spring AOP里面的几个名词;Spring 的 IoC支持哪些功能)
)
