
基因组挖掘指导天然药物分子的发现

摘要
天然产物是临床药物的主要来源,也是新药研发过程中先导化合物结构设计和优化的灵感源泉。但传统策略天然药源分子的发现却遭遇了瓶颈,新颖天然产物的数量逐渐无法满足现代药物开发的需求和应对全球多药耐药的威胁。随着测序技术的快速迭代,生物学的研究进入了基因组时代,基因组挖掘指导天然产物定向发现的策略得以确立,成功摆脱了传统天然产物发现策略对于生物样本生物量的依赖,极大提高了活性天然产物发现的特异性和成功率。本文简述了基因组挖掘以及相关数据库和生物信息学工具的发展,详细介绍了包括基于核心基因或后修饰基因的经典挖掘手段,自抗性机制、进化理论指导的基因组挖掘和人工智能在活性天然产物发现中的具体应用,并对基因组挖掘在药物发现和多学科交叉领域的影响和发展进行了展望。基因组信息中蕴藏着无可估量的化学潜能,促进基因组挖掘与其他学科间的交叉融合,提升对遗传信息的处理和分析能力,增强下游基因簇表达通量和产物结构预测能力,可实现天然小分子高通量、高新颖性和高效率的发现,为开发具有自主知识产权的新药物、新化学品和新型酶催化剂服务。
有机生命体产生的化学物质不仅包括核酸、蛋白质等初级代谢中的生物大分子,也包含大量结构类型多样的小分子次级代谢物,又称天然产物[1]。这些天然产物及其衍生物是临床药物的主要来源,也是新药研发过程中先导化合物结构设计和优化的灵感源泉。为了获得潜在的天然药源分子,研究者们建立了一套直接从生物样本出发,以活性或特殊的理化性质为指导的传统天然产物发现策略,这一策略在20世纪取得了巨大成功,发现了一大批至今仍在临床使用的药物[2-4],如开创抗生素治疗新纪元的青霉素[5]、他汀类降脂药物洛伐他汀[6]、抗癌药物紫杉醇[7]和抗疟药物青蒿素[8]等。然而,由于对样本生物量的高度依赖,该策略的研究对象受限于易获得、易培养的生命体。同时,天然产物在生物样本中的含量差异较大,低丰度的分子无法满足结构解析和活性研究的需求,而高丰度的化合物又常常被重复发现,这些弊端造成了新颖天然产物发现的步伐日趋放缓,逐渐无法满足现代药物的开发和应对多重耐药威胁的需求[9]。
随着分子生物学的快速发展,大量微生物的遗传操作体系逐步成熟,尤其是天然产物丰富的链霉菌(Streptomyces)[10],研究者们开始探寻天然产物合成的遗传信息。1984年David Hopwood课题组[11]将天蓝色链霉菌(S.coelicolor)中一段推测可能负责放线紫红素(Actinorhodin)合成的DNA导入微小链霉菌(S.parvulus)后,成功实现了放线紫红素的异源生产,使得放线紫红素成为了第一个与基因组信息关联起来的微生物天然产物。进入新世纪后,测序技术快速迭代,获取基因组信息的成本大幅降低,天然产物的生物合成研究迎来了基因组时代。随着大量天然产物生物合成过程的解析,研究者们发现,微生物中负责天然产物生物合成的基因在基因组上的邻近区域聚集成簇,形成了天然产物的生物合成基因簇(biosynthetic gene cluster,BGC),负责相同骨架结构合成的基因常常十分相似,非核糖体肽合成酶(nonribosomal peptide synthetase,NRPS)和Ⅰ型聚酮合酶(polyketide synthase,PKS)的结构域排列顺序与产物的合成过程一一对应[12-13]。这些发现为逐步建立基因序列与天然产物结构之间的直接联系提供了理论依据,使得从基因组水平预测生物样本的化学潜能成为了可能[1]。由此,直接从遗传信息入手,定位天然产物的BGC,通过对基因簇的原位激活或异源表达,进而实现天然产物定向发现的策略得以确立,称之为天然产物的基因组挖掘[14]。该策略摆脱了传统方法对样本生物量的依赖,显著简化了分离纯化过程,极大提高了天然产物发现的特异性和成功率。随着生物合成研究在真菌和动植物领域的深入,高等生命体来源的天然产物的生物合成过程也在逐渐明晰,这些合成路径的解析也为基因组挖掘在高等生命体中的应用铺平了道路。
更重要的是,通过对公共数据中细菌基因组和宏基因组的分析,研究者发现了数目和多样性极为庞大的天然产物BGC,其种类远超现阶段已经分离鉴定的天然产物数量,这充分暗示了有机生命体合成新颖天然产物的巨大潜能仍待开发[15],这为基因组挖掘提供了广阔的空间和巨大的机遇。本文首先简述了生物信息数据库和工具算法对基因组挖掘的促进作用,然后详细介绍了基因组挖掘发现活性天然产物的经典案例,并对基因组挖掘的后续发展方向、药物开发和学科间的交互影响等进行了展望。
1 基因组挖掘的数据基础和工具算法
21世纪初,针对天蓝色链霉菌[16]和阿维链霉菌(S.avermitilis)[17]最早完成了全基因组测序,随后其他放线菌的全基因组陆续得到公布,研究者们发现放线菌基因组中存在着大量产物未知的BGC,其多样性远高于已发现的天然产物类型,这体现了基因组数据对于天然产物发现的重要意义[14]。随着测序技术的发展,测序得到的DNA数据正呈现指数级的增长,为了合理且高效地利用这些数据资源,大量专业化的数据库和生物信息学工具也相继诞生,进一步加速了天然产物基因组挖掘的发展。
1.1 基因组挖掘的数据基础
测序技术的发展是基因组数据增长的主要支撑,自第一代测序技术使用的双脱氧链终止法[18]诞生以来,测序技术已历经三代。由于双脱氧链终止法在人类基因组计划[19]实施中的低效表现,直接催生了第二代的高通量测序技术[20],包括了高通量焦磷酸测序法和Illumina染色测序法。为了弥补第二代测序技术读长较短的缺陷,第三代单分子测序技术[21]应运而生,使得测序读长达到了1万个碱基对以上,这对于宏基因组测序的后续组装十分有利。随着测序技术的迭代,测序成本逐年递减,测序效率却逐年递增,形成的大规模基因组数据又催生了各种类型数据库的建立和完善。这些数据库涵盖了核酸序列数据库、蛋白质序列数据库和天然产物生物合成基因簇数据库等。
美国国家生物信息中心(NCBI,https://www.ncbi.nlm.nih.gov)的GenBank数据库[22]是全球最大的核酸序列数据库,包含近26万个物种的核酸序列信息。基于相对冗余的GenBank数据,NCBI选取测序质量更加优异和注释信息更为全面的条目组建了RefSeq数据库[23],该数据库非冗余、广泛交联和注释信息丰富的特点正逐渐受到研究者们的青睐。其他的核酸数据库还包括由美国能源部联合基因组研究中心(JGI,https://genome.jgi.doe.gov/portal)创立的综合性微生物基因组数据库IMG/M[24]。
值得注意的是,大量公布的宏基因组测序数据,拓宽了天然产物基因组挖掘的数据来源,使得天然产物的发现模式不再局限于纯培养的微生物。截至2023年,NCBI的assembly数据库[25]收录的组装后宏基因组达到了2145个,涵盖了水体、土壤、动物消化道等众多环境样本。作为人类基因组计划的延伸,人类微生物组计划已进入第2阶段[26-27],产生的宏基因组数据为研究微生物群对人类健康和疾病的影响发挥了重要的作用,同时也为天然产物的基因组挖掘提供了新的数据来源。基于人类微生物组的数据,Michael A. Fischbach团队从中发现了抗革兰氏阳性菌的活性硫肽类分子lactocillin[28],和多种具有蛋白酶抑制活性的二肽醛类化合物[29]。Shinichi Sunagawa等[30]通过1038个海水样本的宏基因组测序,结合可培养海洋微生物基因组数据,系统研究了海洋中微生物BGC的多样性和新颖性,揭示了大约40 000个潜在的新颖BGC,并搭建了一个交互式的数据分享平台microbiomics-ocean(https://microbiomics.io/ocean/)。这些研究展示了宏基因组数据在挖掘不可培养微生物来源的天然活性分子方面的潜能。
目前最大的蛋白数据库是Uniprot(https://www.uniprot.org)的UniprotKB蛋白质序列数据库,该库收录了超过57万个经文献核实的具有功能注释的蛋白序列条目,和超过2亿条普通序列[31]。基于蛋白质序列数据库,又形成了包括Pfam[32-33]、InterPro[34]等蛋白质家族数据库,这些数据库依据功能和序列特征将蛋白质分为不同的家族,并记录同一家族的序列保守性,这些信息极大促进了蛋白质功能预测工具的开发和基因组挖掘过程中基因功能的预测。
随着越来越多的天然产物生物合成基因簇被报道,天然产物生物合成基因簇数据库应运而生。其中,由150位科学家联合创立的生物合成基因簇的最小信息数据库(MIBiG,https://mibig.secondarymetabolites.org)[35],截止到2023年10月,较为全面地收录了2502条已鉴定的天然产物生物合成基因簇,使之成为开发基因组挖掘工具的重要数据基础,该数据库也为生物合成基因簇及其编码产物的信息收录建立了参考标准。IMG-ABC天然产物生物合成基因簇数据库(https://img.jgi.doe.gov/cgi-bin/abc-public/main.cgi)基于JGI-IMG/M平台,成为目前工具最为齐全的数据库,囊括了已知(主要来源于MIBiG)和antiSMASH v5预测的共411 407条生物合成基因簇。作为应用最为广泛的次级代谢产物基因簇预测工具antiSMASH,其开发者基于antiSMASH软件[36]对RefSeq核酸数据库中微生物天然产物基因簇的预测,组建了基因簇数据库antiSMASH-DB(https://antismash-db.secondarymetabolites.org),2023年9月发布的第四版中,已包含了231 534条次级代谢产物生物合成基因簇[37]。
1.2 用于基因组挖掘的经典算法与工具
天然产物基因组挖掘的核心在于有效识别和区分BGC,本质上是对基因或蛋白质序列的分析。经典的用于生物序列比对和聚类的算法在基因组挖掘中均有应用,如基本局部相似性比对工具(basic local alignment search tool,BLAST)[38],该算法可以实现探针序列和多序列数据库的比对,在最短的时间内寻找最优的匹配序列,从而发现探针序列的同源物。该算法目前是NCBI网站指定的线上搜索工具,目前已衍生出了PSI-BLAST、PHI-BLAST和DELTA-BLAST等功能更加丰富的工具[39]。另一个较为常用的比对算法是隐马尔可夫模型(hidden Markov model,HMM),该算法可以对相似性的蛋白质序列数据集进行分析,形成序列位置上20种氨基酸残基出现频率的矩阵模型,该模型体现的是多序列的保守信息,如蛋白质的结构域(domain),利用该模型作为探针,可以更加准确和全面地发现同类蛋白质,避免单一条目检索时带来的亲缘物种的偏好性和非保守区域的无效匹配。聚类算法中较为突出的是CD-HIT(Cluster Database at High Identity with Tolerance)[40],该算法的核心是通过组建“最长序列优先”列表来删除超过某个一致性阈值的序列,以减少冗余或高度相似序列,生成非冗余的输出结果,展现与目的序列密切相关的蛋白质家族的成员。
此外,特异性地用于天然产物生物合成基因簇预测和注释的软件在过去的20年间层出不穷,常用的分析工具包括ClustScan[41]、CLUSEAN[42]、NP.searcher[43]、antiSMASH[36]和PRISM[44]等。ClustScan可快速、半自动地对编码模块化生物合成酶的DNA序列进行注释,包括聚酮合酶(PKS)、非核糖体肽合酶(NRPS)和PKS-NRPS杂合酶,同时也能预测NRPS和PKS产物的化学结构[41]。antiSMASH是目前使用最广泛的基因组挖掘工具,可用于古菌、细菌、真菌(fungiSMASH)和植物(plantiSMASH)基因组中天然产物生物合成基因簇的快速识别、注释和分析。最新版本的antiSMASH 7[36]将基因簇的类型拓展至81种,并且对产物结构预测、酶装配线可视化和放线菌转录调控因子结合位点预测等功能进行了优化和改进。PRISM除了有类似antiSMASH的基因簇预测和天然产物结构预测的功能外,还通过引入深度学习算法来进行产物及活性预测,将基因簇和天然产物潜在的生物学功能融合在一起[44]。
然而,这些工具对于编码核糖体翻译后修饰肽(RiPPs)基因簇的识别仍然有所欠缺,主要是由于前体肽序列过短且缺乏保守特征,从而难以准确地定位和预测。为了解决这一问题,许多专门针对RiPPs的基因簇挖掘和识别工具相继问世。RODEO[45](https://www.ripp.rodeo)是首个结合了HMM分析,启发式评估和机器学习来预测前体肽的基因组挖掘工具,目前支持对套索肽类、Ⅰ型羊毛硫肽和硫肽类RiPPs的基因组挖掘和分析。RRE-Finder[46](https://github.com/Alexamk/RREFinder)是一种通过定位RiPP前体识别元件(RiPP precursor recognition element,RRE)的基因组挖掘工具,许多Ripps的后修饰酶依赖于RRE与前导肽的结合来发挥对核心肽的修饰作用。RRE-Finder有两种使用模式,在精准模式下能够检索到所有已表征的包含RRE的RiPPs类别;在探索模式下可以从UniProtKB蛋白数据库中调取到大量未表征的高可信度RRE蛋白序列,从而定位到新颖RiPPs的生物合成基因簇。DeepRiPP[47](http://deepripp.magarveylab.ca)使用深度学习算法从基因组中预测RiPPs等短肽序列。在无法获得完整基因簇的情况下,DeepRiPP也能仅通过短肽序列就判断出该短肽是否是RiPPs家族的前体肽,并根据已知RiPPs后修饰酶的特性预测出其结构;结合代谢组的质谱数据,DeepRiPP还能在代谢组中精确定位出RiPPs的信号。
同样为了克服了宏基因组等测序信息不完整的缺陷,Sugimoto等[48]开发了利用分段式隐马尔可夫算法(spHMM)的MetaBGC,不依赖于测序数据的组装,从测序数据可以直接获得天然产物合成基因簇信息。MetaBGC首先将保守的天然产物合成酶序列打碎成30个左右的氨基酸片段来模拟基因测序的片段,然后对这些片段分别构建pHMM并进行评估。接着,作者利用评分高的pHMM从宏基因组数据中直接找出天然产物生物合成基因。这种方法使得从碎片化严重的宏基因组数据中进行基因组挖掘变得简单而迅速。
2 针对生物合成核心基因的基因组挖掘
天然产物按化学结构和生源途径可简单划分为聚酮类、多肽类、萜类、生物碱类、嘌呤和嘧啶类以及苯丙素类等。各类别结构骨架的高度相似性暗示了同类别天然产物具有保守的生物合成逻辑,负责编码骨架形成的核心酶具有一定的保守性。因此,可以通过定位天然产物骨架的合成基因来实现对特定类别天然产物基因簇的发现,结合基因簇中其他基因的比较分析,从而挖掘出同一类别但结构更加丰富的天然产物,为药物的开发提供结构更加多样的先导化合物。
2.1 烯二炔类天然产物的基因组挖掘
烯二炔类天然产物因其独特的分子结构和超强的抗肿瘤活性而备受关注,其核心结构是一个由双键偶联两个炔键构成的烯二炔,依据其核心烯二炔环的大小,分为九元环和十元环烯二炔两种类型。烯二炔活性基团可通过伯格曼芳构化反应产生的苯双自由基,作用于DNA小沟区促使DNA链间交联或双链断裂,从而发挥抗肿瘤活性[49]。目前,烯二炔类化合物卡奇霉素(Calicheamicin)已开发成为FDA批准上市的抗体偶联药物(antibody-drug conjugate,ADC),而其他的烯二炔类天然产物仍然是后续ADC开发潜在的有效载荷[50]。基于已知报道的烯二炔类天然产物基因簇,该类天然产物的生物合成需要5个保守的聚酮合酶基因(E3/E4/E5/E/E10)(图1)。Shen Ben课题组[51]多年来一直致力于烯二炔类天然产物的发现和生物合成研究,他们通过分析Genbank数据库中4889个细菌基因组信息,发现了61个基因簇中含有上述合成烯二炔骨架结构特征的保守基因盒,其中10条来源于已报道的烯二炔类天然产物生物合成基因簇。为了快速发现潜在的同源基因簇,该团队[52]采用前期开发的高通量实时PCR技术,即预先建立未测序的细菌基因组文库,对负责特定天然产物合成的关键且保守的基因,设计简并引物,以基因组文库为模板,进行PCR,然后使用DNA荧光燃料SYBR Green Ⅰ对PCR产物进行熔解曲线分析,以得到的Tm值为指标,判断菌株基因组中是否含有该关键基因的同源物,再结合进一步的测序,从而高效地发现大量特定天然产物的同源基因簇。基于此,该团队针对烯二炔聚酮合酶(E5/E或者E/E10)基因设计简并引物,通过高通量的实时PCR技术,从3400株放线菌中筛选出81株潜在的烯二炔产生菌株,并对其中的31株菌进行全基因组测序,确证了该方法的可靠性。最终,他们[53]发现了C-1027的高产菌株,可满足后续在化学、生物学和临床等领域的研究,以及一类新颖的具有强效广谱抗肿瘤活性的烯二炔天然产物天赐霉素(Tiancimycin A)(图1)。
图1
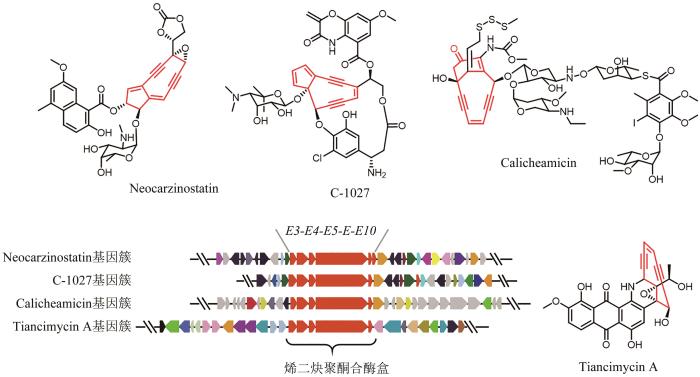
图1 烯二炔类天然产物生物合成基因簇及代表性化合物
2.2 脂肽类天然产物的基因组挖掘
脂肽是一大类具有生物活性的微生物天然产物,结构中通常包含一个亲水性环肽以及N端不同长度或官能团化的疏水性脂肪链,这种两亲性使脂肽高度功能化,比如作为生物表面活性剂;同时也赋予其不同的生态角色,包括物种间的防御、竞争和共生等[54-55]。目前,报道的大多数脂肽都是非核糖体来源,由多结构域的大型非核糖体肽合酶以装配线的形式线上合成。经典的最小NRPS模块包含识别氨基酸底物的腺苷化(A)结构域,催化肽键形成的缩合(C)结构域和挂载延伸肽链的硫酯(T)结构域。这些结构域以及在链延伸过程中可能存在的一些差向异构酶结构域或者甲基转移酶结构域均可通过生物信息学工具进行预测。基于此,Sean F. Brady课题组[56]近几年尝试利用基因组信息来预测非核糖体肽结构,建立了合成-生物信息天然产物(synthetic-bioinformatic natural products,syn-BNPs)发现新方法。
在最近的工作中,研究者系统分析了10 858条细菌基因组信息,从中找到35条可能编码多黏菌素类似物的非核糖体肽生物合成基因簇。多黏菌素为脂肽类天然产物,曾被誉为抵御革兰氏阴性菌的最后一道防线,而如今也面临肆虐全球的多黏菌素抗性基因的威胁[57-58]。研究者认为,自然进化产生多黏菌素同系物可能是对抗自然耐药性的一种策略,也极有可能抵御临床上的耐药性。研究者以腺苷化结构域信息为指导,选择了其中与多黏菌素结构差异最大的生物合成基因簇,不使用微生物的培养和基因簇的激活,而是利用固相多肽合成技术化学合成了化合物Macolacin(图2),发现其对临床上常见的几种耐多药(包括多黏菌素)细菌均具有高效的抑制活性[59]。syn-BNP方法利用生物信息学算法,从持续增长的海量微生物基因组中通过化学-酶合成方法快速获得数量庞大的syn-BNP化合物库,从中鉴定新的生物活性小分子,为针对耐药病原菌的抗生素发现开辟了一条资源获取新途径[60-62]。
图2
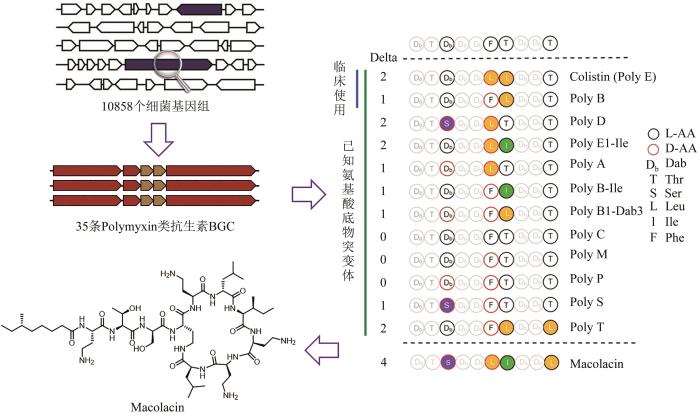
图2 从细菌天然基因簇的组成中获得启示,发现抗耐药株的新型多黏菌素类分子Macolacin[59]
2.3 膦酸盐类天然产物的基因组挖掘
膦酸盐类化合物是一类比较罕见的含碳磷键的天然产物,其能够模拟生物分子的磷酸酯或碳酸基团,影响细胞的代谢过程和信号转导,从而表现出抗菌、抗癌和除草的活性[63]。并且,不同于磷酸酯中易水解的氧磷键,碳磷键的高度稳定性使这类化合物同时具有抵抗化学和酶降解的能力。目前,已经有许多实现商业化的膦酸盐药物,包括临床上应用于细菌性膀胱炎的广谱抗生素磷霉素(Fosfomycin)、市售除草剂的主要成分草铵膦(Glufosinate)、治疗与艾滋病相关的巨细胞病毒和疱疹病毒感染的膦甲酸钠(Hosphonoformate)[64]等。膦酸盐类天然产物水溶性高,无法提取到有机溶剂中,且大多数为短肽,没有紫外吸收,想通过传统的天然产物发现策略获得膦酸盐无异于大海捞针。针对膦酸盐天然产物的生物合成研究表明,其生物合成过程均起始于磷酸烯醇式丙酮酸变位酶(PepM)催化磷酸烯醇式丙酮酸(PEP)转变为磷酸丙酮酸(PnPy),随后再经下游的酶催化产生后续的生物合成中间体,分流至不同类型膦酸盐天然产物的生物合成路径中[65-66]。因此,将pepM作为基因簇标志物应用于膦酸盐类天然产物基因组挖掘,成为了以生物信息学为导向的该类天然产物发现的新策略。2015年,Wilfred van der Donk和William W. Metcalf课题组[67]利用该方法,设计pepM的简并引物,通过大规模高通量聚合酶链反应(PCR),在1万株放线菌基因组中,鉴定出403株可能的膦酸盐产生菌株。随后通过基因组测序,确认其中278株包含了膦酸盐类天然产物的生物合成基因簇,并通过PepM的序列相似性网络分析和系统发育分析,从中发现了5个潜在的新类别基因簇,结合31P NMR磷谱的检测,至少有45株产生了膦酸盐类天然产物(图3)。凭借该策略,该团队最终发现了11种新颖的膦酸盐类天然产物。
图3

图3 从放线菌基因组中挖掘活性膦酸盐类天然产物
2.4 海洋动物来源的萜类化合物的基因组挖掘
萜类化合物是最丰富的天然产物,该类化合物主要来源于植物和微生物的次级代谢,并挖掘了许多重要临床药物如紫杉醇和青蒿素等。萜类合酶(terpene cyclase,TC)是萜类生物合成过程中塑造萜类骨架的核心酶,常作为萜类挖掘的主要切入点。依据形成碳正离子方式的不同,经典的萜类合酶可以分为两种类型,Ⅰ型萜类合酶的序列中含有保守的DDxxD基序和NSE/DTE基序,以异戊烯基焦磷酸前体上焦磷酸部分的离去而触发碳正离子的生成。Ⅱ型萜类合酶的序列中含有保守的DxDD基序,以质子化异戊烯基前体中烯键或环氧环来触发碳正离子的生成[68-69]。由于目前已知的萜类合酶在一级序列的相似性并不高,难以使用单一的特定序列来广泛发掘其他未知的萜类合酶,针对微生物来源的萜类合酶的基因组挖掘均采用了HMM算法生成的模型来进行挖掘,Haruo Ikeda团队[70]将HMM和系统发育分析结合,戈惠明团队[71]将HMM和序列相似性网络分析结合,均发现了大量细菌来源的萜类合酶及其产物。随着高等生物基因组和转录组数据的逐渐释放,针对动植物来源萜类的基因组挖掘也已取得新的进展。
八放珊瑚是萜类化合物的一个重要来源,已分离出超过4000种倍半萜和二萜类化合物,占已报道海洋天然产物的12%以上[72]。大多数海洋无脊椎动物依赖于共生菌产生的天然产物来进行捕食和防卫,而未发现八放珊瑚拥有丰富的共生微生物和藻类[73]。Bradley S. Moore课题组[74]对已表征的细菌和真菌Ⅰ型萜类合酶序列构建HMM,随后在已测序的八放珊瑚(Dendronephthagigantea)中进行检索,得到了一些得分较低的序列,利用这些序列对HMM进行优化后,对已公开的八放珊瑚基因组和转录组重新进行检索。结合系统发育分析,发现这些挖掘到的条目与植物和微生物来源的已知的萜类合酶不同,在进化树上单独成为一支。尽管如此,但所有珊瑚来源的萜类合酶序列都具有微生物Ⅰ型萜类合酶的保守基序。研究者利用大肠杆菌对这些八放珊瑚来源的萜类合酶进行异源表达,共获得了8个萜类骨架产物(图4),并通过体外酶反应验证了它们的功能,证明了从八放珊瑚中分离得到的许多萜类化合物是珊瑚自身编码的酶催化产物,而不是来源于共生的藻类或微生物。同期,Eric W. Schmidt课题组[75]同样采用HMM检索的方法在八放珊瑚E.caribaeorum中挖掘到多个萜类合酶,并表征了两个能形成eunicellane型二萜化合物关键前体的萜类合酶。两个研究团队都在这些珊瑚来源的萜类合酶基因附近发现了编码细胞色素P450酶、脱水酶和短链脱氢酶等的后修饰基因,这些基因可能形成了萜类的合成基因簇,佐证了珊瑚自身产生多样性萜类化合物的能力,也是探索新型珊瑚来源萜类天然产物的基因基础。
图4
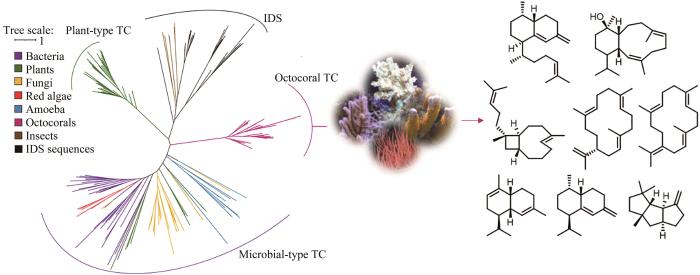
图4 针对萜类合酶的基因组挖掘发现八放珊瑚来源的萜类[74]
3 针对特定药效团的基因组挖掘
天然产物的结构中常包含一些对发挥生物学功能有重要影响的化学基团,称为药效团。药效团可能是一些亲电或亲核基团,直接与靶标蛋白共价结合,影响或破坏蛋白的功能;或是合适的大小或电负性促使该部分基团以非共价的形式有利占据蛋白的活性空腔,使蛋白无法发挥正常功能[76-77]。这些药效团往往由后修饰酶催化形成,包括在天然产物骨架结构中进行氧化、糖基化、卤化、硝化等特定类型的修饰,亦可催化天然产物骨架的重排、芳构化、大环化等,进一步拓展了天然产物结构的多样性和复杂性[78-82]。因此,以催化形成特定药效团的后修饰酶作为探针展开基因组挖掘,有助于发现活性更优的新颖天然产物,而且,以药效团为出发点而不是骨架合成基因为挖掘探针,可以有效突破天然产物类别的限制。
3.1 卤代天然产物的基因组挖掘
卤素基团是众多临床药物的药效团,化合物中卤素取代基可通过位阻效应、极性效应或与蛋白受体形成卤键来影响化合物的生物活性[83]。例如,人体所分泌的甲状腺激素结构上碘的数量是激素活性的重要因素[84];糖肽类抗生素万古霉素,当骨架上两个氯原子中任何一个缺失,都会导致抗菌活性的大幅下降[85];抗肿瘤海洋天然产物Salinosporamide A依赖于氯原子来发挥蛋白酶体的共价抑制活性[86]。生物体内的卤化过程由卤化酶所负责,其作用机制与生物氧化类似,主要包含有亲电性的血红素依赖性卤化酶、黄素依赖性卤化酶和钒依赖性卤化酶、自由基机理的α-KG依赖性卤化酶以及亲核性的SAM依赖性的氟化酶[87]。
为了更高效地从庞大的基因组序列中发现含卤天然产物,Pelzer课题组[88]以黄素依赖性卤化酶的保守序列为探针,对550株随机选择的放线菌进行PCR筛选,从中鉴定出103条可能编码卤化酶的基因,结合系统发育分析和质谱检测,分离出卤化的Ⅱ型聚酮化合物CBS40,其对多种革兰氏阳性菌都表现出强效的抑制活性。Xie Yunying课题组[89]采用类似的方法以黄素依赖性卤化酶GedL为探针在本地真菌数据库中进行tBLASTp分析,抽取周边基因信息利用antiSMASH进行注释,找到一条未被表征的含卤化酶的生物合成基因簇,通过发酵培养,最终分离得到含卤天然产物Pestalachlorides 1a/1b和2a/2b,这两对阻转异构体对一些临床上的耐药菌株均有不同程度的抑制活性[图5(a)]。
图5
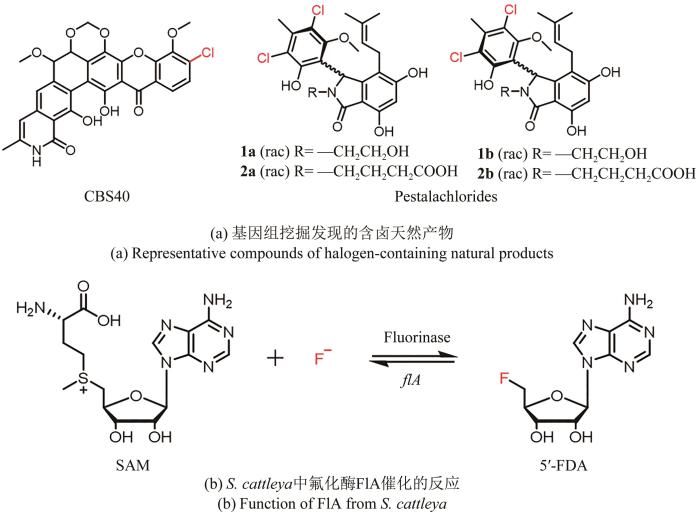
图5 卤代天然产物的基因组挖掘
虽然氟在自然界的丰度很高,但生物体内的氟化并不常见。目前,只有一类SAM依赖性氟化酶被报道直接催化C-F键的形成[图5(b)]。David O′Hagan课题组[90]利用BLAST工具从三株不同种属放线菌中定位到与已报道的来自卡特兰链霉菌(S.cattleya)的氟化酶基因flA高度同源的3个基因,并通过体外酶学实验和晶体学研究表征它们的功能,为含氟天然产物的发现和有机氟化物的生物转化提供了新的思路。
3.2 含特殊元素天然产物的基因组挖掘
硒是一种非金属元素,是动物体必需的微量元素和植物有益的营养元素[91-92]。目前,可通过硒代半胱氨酸和2-硒尿苷将硒引入蛋白质和核酸中,具体过程是硒磷酸合成酶(SelD)对硒化氢进行磷酸化生成硒磷酸(SeP),作为硒代半胱氨酸合酶(SelA)和硒尿苷合酶(SelU)的底物,将其引入到生物大分子中[93-95],而将硒特异性引入有机小分子的生化过程尚未报道。鉴于SeP是常见的硒供体,Mohammad R. Seyedsayamdost研究团队[92]推测含硒小分子可能遵循类似的生物合成逻辑,由于微生物中负责特定天然产物合成的基因常聚集成簇,以selD为切入点,在该基因的周边有可能存在与有机硒化物合成相关的基因,从而有可能从头发现一条有机硒化物的合成通路。基于这一假说,研究者展开了针对有机小分子硒化物的基因组挖掘。首先,通过在NCBI的保守蛋白结构域家族(CDD)中调取SelD家族COG0709的所有序列,使用CD-HIT工具进行聚类分析以去除冗余序列,再通过E-Direct工具获取selD基因周边4000 bp的区域,为了进一步提升周边基因与selD的相关性,研究者重点关注那些与selD同向且具有重叠区域的共定位基因,除了已表征的与硒代大分子生物合成和硒代谢或转运相关的基因外,一个编码功能尚不明晰的tigr04348家族糖基转移酶的基因引起了作者的关注。为了进一步获取更多的信息,研究人员继续寻找与selD和tigr04348两个基因共定位的第3个基因,从中发现了一个编码EgtB蛋白的同源基因,而EgtB负责催化麦角硫因生物合成中C-S键形成[96]。827个不同的细菌基因组中均保守地含有这3个紧邻排布的基因组合,表明其可能编码了一个全新而广泛存在的含硒代谢产物的生物合成途径。随后,研究人员通过代谢组学分析,从两株细菌的代谢物中鉴定到了麦角硫因(ergothioneine)和麦角硒因(selenoneine,SEN),后续的体外酶学表征,揭示了无机硒元素掺入有机天然产物之中的合成路径(图6),这一案例充分展示了基因组挖掘策略可以实现全新类型天然产物及其生物合成途径的从头发现,是近年来基因组挖掘研究中的标志性成果。
图6
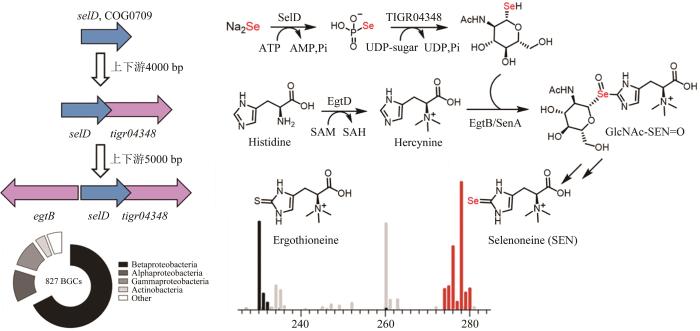
图6 通过基因组挖掘发现新型硒代天然产物
砷元素广泛存在于自然界中,无机砷俗称“砒霜”,通常有剧毒;而含有C-As键的有机砷类化合物毒性较低,并且可以作为化疗药物的候选药物[97]。目前,虽然已报道了300多个有机砷类天然产物,但对它们的生物合成认识依旧有限,大多可能是由As(Ⅲ) S-adenosylmethionine(SAM)甲基转移酶连续催化甲基化而形成终产物[97-98]。Hiroyasu Onaka课题组[99]从模式放线菌Streptomyceslividans A3(2)中鉴定了一个砷类次级代谢产物Bisenarsan,在阐明生物合成途径过程中发现其中间体具有抗菌活性,并确定了其中参与C-As键形成的磷酸甘油酸变位酶BsnN,而非其他有机砷生物合成过程中的由甲基转移酶催化砷的烷基化。随后,研究人员在RefSeq数据库中检索了100条同源基因进行系统发育分析,结果表明BsnN同源蛋白能够在进化上与经典的磷酸甘油酸变位酶实现功能区分。并且,BsnN同源蛋白集中分布于各种放线菌基因组上,暗示了放线菌中蕴藏着产生砷类天然产物的巨大潜力,可服务于后续通过基因组挖掘探索更多具有药用价值的有机砷类化合物。
3.3 含有N—N键天然产物的基因组挖掘
含有N-N键的天然产物较为罕见,但衍生出了许多复杂多样的官能团,包括哌嗪酸、重氮基团、亚硝胺和二醇二氮𬭩等,丰富的结构多样性赋予了这类天然产物多样的生物活性[100],比如作为DNA烷基化试剂的抗肿瘤药物链脲佐菌素(Streptozotocin)[101],具有MTAP缺陷肿瘤细胞抑制活性的丙氨菌素(L-alanosine)[102]和具有强效抗真菌活性的Kutzneride[103]等。
2017年,Katherine S. Ryan课题组[104]在含有哌嗪酸结构单元的非核糖体肽Kutzneride生物合成过程中,报道了首例以N-羟基鸟氨酸为底物催化N-N键形成的血红素依赖酶KtzT,并且发现ktzT同源基因在含有哌嗪酸合成砌块的天然产物生物合成基因簇中高度保守。随后,Raymond J. Andersen等[105]就以KtzT为探针,在NCBI数据库中定位到一条NRPS基因簇,通过发酵培养结合1H/15N HSQC-TOCSY分析,获得了含哌嗪酸的肽类天然产物Incarnatapeptin A和Incarnatapeptin B,其中Incarnatapeptin B在体外对前列腺癌细胞具有抑制活性。为了充分开发未测序菌株的生物合成潜力,Oh Dong-Chan研究团队[106]针对ktzT及其上游基因ktzI的保守区域设计了兼并引物,对包含2020株细菌基因组的DNA文库进行PCR筛选,鉴定出62株阳性菌株,通过发酵培养,最终分离表征了3个新颖的含哌嗪酸单元的天然产物,其中Lenziamide A具有抗结肠癌活性[图7(a)]。
图7
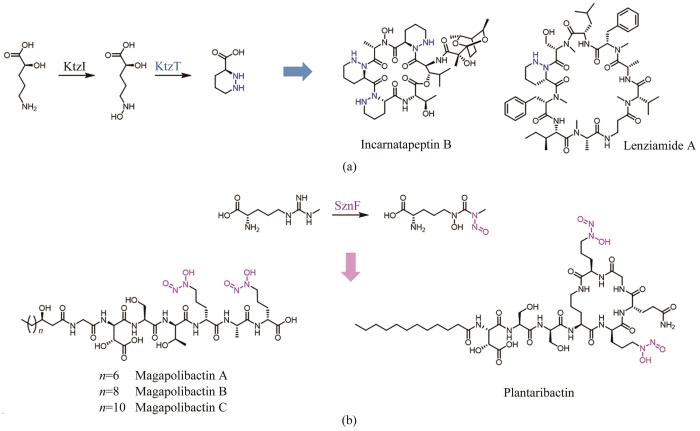
图7 基因组挖掘含哌嗪酸和二醇二氮𬭩结构单元的天然产物
2019年,Emily P. Balskus研究团队[107]在对含有亚硝胺结构片段的天然产物Streptozotocin进行生物合成研究过程中,发现了一个以N-甲基精氨酸为底物催化氧化重排形成N-N键的金属酶SznF。SznF是一个多结构域蛋白,包括了NADPH依赖的氧化还原结构域和C端的cupin结构域,分别行使对N-甲基精氨酸氧化和重排的催化功能。随后,Christian Hertweck课题组[108]以cupin结构域同源蛋白GrbD为探针,利用EFI-EST工具对可能含有亚硝胺结构单元的天然产物进行挖掘,定位到37条潜在基因簇,选取部分菌株进行发酵后,从中分离鉴定出10个含有二醇二氮𬭩结构的嗜铁素类天然产物[图7(b)]。
3.4 rSAM介导的RiPPs类环肽天然产物的挖掘
自由基SAM酶(rSAM)利用[4Fe-4S]簇和S-腺苷甲硫氨酸(SAM)产生5-脱氧腺嘌呤核苷自由基(dAdo•)中间体从而诱发后续各种各样的自由基反应,涉及天然产物生物合成、辅因子生物合成和蛋白质的翻译后修饰等生化过程[109]。对于核糖体翻译后修饰肽(ribosomally synthesized and post-translationally modified peptides,RiPPs)而言,rSAM可催化线性的多肽前体中多种类型氨基酸之间的交联反应。
Sactipeptides是一类具有抗菌、溶血等生物活性的RiPPs天然产物,其结构中Cα-S键的交联由rSAM催化。Douglas A. Mitchell课题组[110]从InterPro数据库中找到超过450 000条rSAM,用已表征的rSAM序列为探针,经PSI-BLAST四轮分析后,选取约4600条rSAM序列进行相似性网络分析(SSN)。对其中处于不同聚类的3条基因簇进行产物鉴定,分别得到新颖的具有生长抑制活性的Cα-S交联产物Huazacin、Cβ-S交联产物Freyrasin和Cγ-S交联产物Thermocellin。
2018年,Seyedsayamdost研究团队[111]提出链球菌的RaS-RiPPs网络分析,他们整合了2875株链球菌中所有由shp/rgg群体感应(QS)系统控制的包含rSAM的RiPPs基因簇,利用前体肽序列构建SSN,以底物特异序列来区分rSAM酶的潜在功能,最终形成16个类群。截至2022年,他们已对其中8个类群的rSAM酶的功能进行了挖掘和验证,发现了W-K、C-N、R-Y、T-Q、C-G、C-S以及S-H的环化交联产物[112-115](图8)。
图8
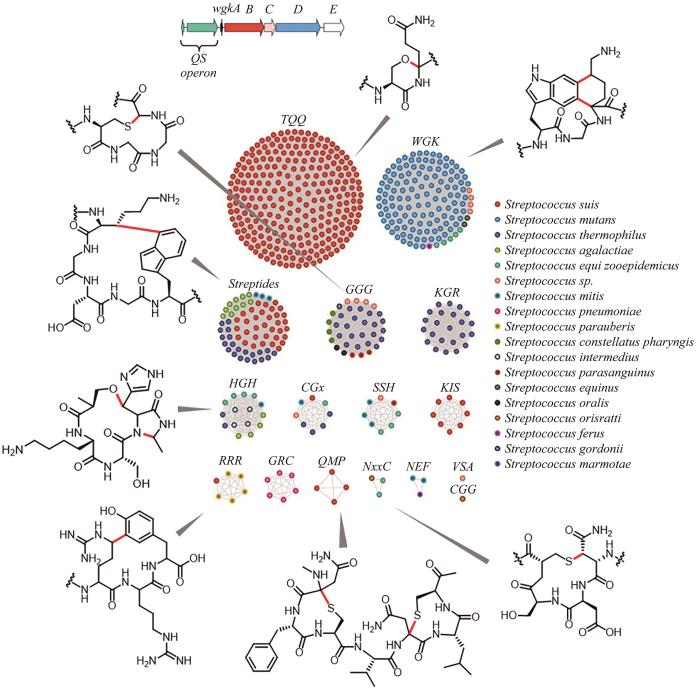
图8 RaS-RiPPs序列相似性网络和对应的交联产物[111-115]
(已表征类群的rSAM酶催化形成的交联由红色标注)
3.5 真菌RiPPs类环肽天然产物的挖掘
相较于细菌,真菌来源RiPPs类环肽天然产物的挖掘却十分少见,这主要受限于该类天然产物的发现和生物合成研究相对较少。目前,真菌来源的RiPPs类环肽天然产物可以分为Cycloamanides、Borosins和Dikaritins三个家族[116]。Cycloamanides类是以酰胺键方式首尾环化的小环肽,典型的代表是Cycloamanide B,该类前体肽序列的N端含有较为保守的MSDIN序列,一种保守的脯氨酰寡肽酶B(PopB)负责核心肽的释放和酰胺键形成,实现其头尾大环化[116](图9)。为了更多地挖掘这一类RiPPs天然产物,研究者以前体肽中较为保守的N端和C端氨基酸序列为探针,在不同种属的担子菌纲真菌的基因组中陆续发现了大量编码Cycloamanides类核糖体肽的基因簇[117],其中,Jonathan D. Walton团队[118]利用LC-MS/MS在剧毒真菌死亡帽(Amanitaphalloides)代谢物中发现了该家族的两个新成员Cycloamanide E和Cycloamanide F。Borosins家族成员的生成依赖于脯氨酰寡肽酶P(OphP)催化的核心肽释放和酰胺键形成,该家族的前体肽(OphMA)融合了氮甲基转移酶的功能,致使该家族产物结构中存在高度的氮甲基化[116],如Omphalotin H(图9)。基于此,Michael F. Freeman团队[119]通过使用ophMA的氮甲基转移酶结构域作为探针,最终从基干菌类和子囊菌类真菌中共寻找了54个与OphMA类似的前体肽,并通过LC-MS/MS对相应的产物进行了结构验证。Dikaritins家族环肽是通过醚键交联成环的小环肽,Ustiloxin B(图9)是该家族的第一个成员,其生物合成基因包括前体肽基因ustA,负责环化的DUF3328结构域蛋白基因ustYa/Yb和肽酶基因kex1/2[120]。基于此,Myco Umemura团队[121]通过ustY基因和ustA基因的重复序列在曲霉属真菌中进行检索,成功发现了94个同源基因簇,并通过异源表达和产物的分离,发现了该家族的新成员Asperipin-2a(图9)。2023年,谭仁祥团队[122]在一株昆虫的内生真菌Acauliumalbum H-JQSF中发现了一个由30个常见蛋白源L型氨基酸组成的新环肽产物Acalitide(图9),具有显著的抗帕金森病的活性。结合产生菌的基因组序列分析和合成基因在米曲霉(Aspergillusoryzae)宿主中的异源表达实验,发现这是一种新类别的真菌RiPP,催化线性前体肽发生环化的酶是一个注释为S53家族肽酶的蛋白AcaB,可以预见,该发现将会开启针对这一新型环肽的基因组挖掘研究。这些研究直接促进真菌来源RiPPs的发现和后续的生物合成研究,展示了有别于细菌的独特的RiPPs形成过程,为新型真菌环肽的发现开创了新的思路。
图9
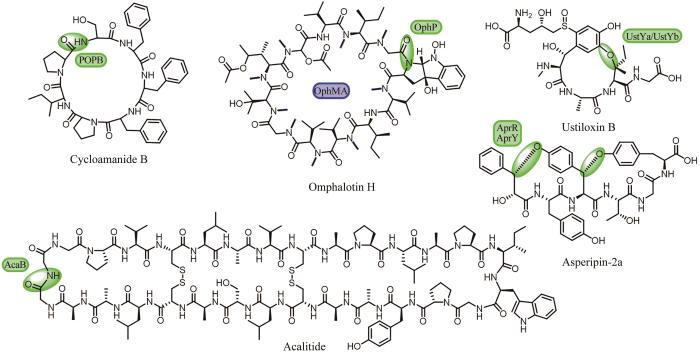
图9 真菌来源的RiPPs以及催化其环化的酶
3.6 植物RiPPs类自催化分支环肽天然产物的挖掘
植物产生的RiPPs种类远不及微生物丰富,但往往都具有显著且多样的药理活性。其中,有两类环肽的生物合成过程较为明晰,分别是由内肽酶或蛋白酶催化头尾相连的环肽(如Cyclotide[123]和Orbitide[124]等)以及由前体肽C端BURP结构域催化氨基酸之间发生偶联的分支环肽(如枸杞素[125]和Moroidin[126]等)。BURP(该家族蛋白的四个经典成员BNM2、USP、RD22和PG1β的首字母组成)结构域是一类铜离子依赖的C端蛋白,目前只在陆生植物中发现,通常与非生物胁迫反应相关[127]。Roland D. Kersten和Marnix H. Medema研究团队[128]合作开发了一套植物分支环肽的系统性发掘流程(图10)。他们首先利用液相色谱-质谱来获取植物组织有机粗提物的氨基酸亚胺离子碎片信息,将这些代谢组学数据集提交至包含已知分支环肽代谢组信息的分子网络中,随后将从分子网络获得的氨基酸信息结合植物基因组和转录组中的前体肽序列(是否含有BURP结构域)来判断潜在分支环肽的新颖度。按照此流程,分离鉴定了5类植物来源的分支环肽,同时也表征了前体肽中的BURP结构域为自催化的肽环化酶。鉴于BURP结构域与分支环肽骨架形成直接相关,Marnix H. Medema等在plantiSMASH[129]中寻找编码BURP结构域的基因并开发了一个RepeatFinder算法确认基因N端是否包含重复的核心肽序列;基于此,在苋菜、土豆和番茄基因组中找到了已知分支环肽枸杞素的前体肽序列,从而证明了利用该方法进行植物分支环肽基因组挖掘的可行性;随后,发现大豆基因组中的一段编码BURP结构域的序列GLYMA_04G180400包含了与已报道分支环肽区别较大的核心肽序列,将该基因合成后在本氏烟草(N.benthamiana)中进行瞬时表达成功产生了两个分支环肽。
图10
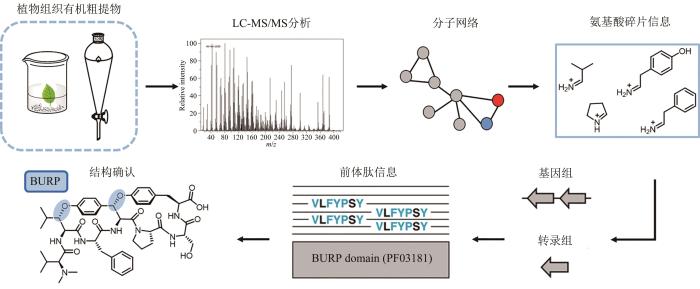
图10 植物分支环肽发掘流程
4 自抗性基因在基因组挖掘中的应用
上述以核心酶或修饰酶为探针的基因组挖掘均是一种结构导向的天然产物发现策略,这种策略难以预知目标产物的生物活性。这对于下游以活性为导向的药物开发十分不便。因此,建立一种通过基因组序列信息直接预测产物活性的方法,来实现目标活性天然产物的精准挖掘,则能极大加速药物开发的进程。
天然产物一般都具有特定的生理功能,有利于保障生产者在特定环境中的生存优势,例如,微生物产生活性次级代谢产物靶向竞争者体内重要代谢途径中的酶来抑制竞争者的生长。然而在抑制竞争者的同时,这些活性产物也有可能对生产者自身产生毒害作用。为了避免这种情况发生,生产者需要自身进化出自抗性机制。目前,已知的自抗性机制主要包括:①转运蛋白将毒性产物运输到胞外的外排泵机制[130];②抗性蛋白对细胞内的毒性产物进行捕获[131]或修饰[132];③靶点蛋白被修饰,使之不受毒性产物的抑制[133];④菌株自身编码了一个持家基因的突变体,也称为自抗性基因,使之既具有原始代谢酶的催化功能,又不被天然产物所抑制[134]。由于这种突变体与持家基因高度类似,且常常与天然产物生物合成基因成簇存在,因此可以通过对自抗性基因功能的生物信息学分析来直接预测基因簇产物的生物活性和作用靶点,实现活性导向的天然产物基因组挖掘(图11)。
图11
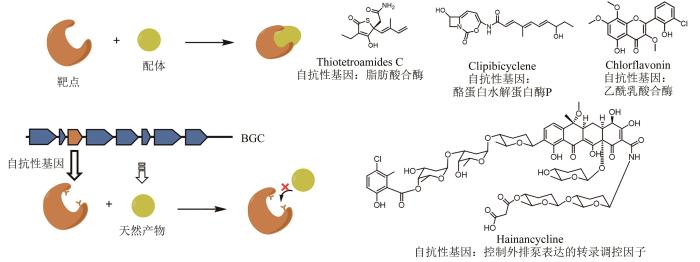
图11 自抗性基因导向的基因组挖掘获得活性天然产物
2013年,Gerard D. Wright研究团队[135]认为对某一类抗生素不敏感的菌株可能编码了一些针对该类抗生素的抗性基因,而这些抗性基因很可能是为了抵御自身产生的类似抗生素,这为发现特定类型的新抗生素提供了崭新的视角。为了验证猜想,他们构建了一个基于万古霉素抗性的放线菌筛选平台,以催化糖肽类天然产物交联的单加氧酶为探针,通过PCR筛选和系统发育分析,发现了一个新型糖肽类抗生素Pekiskomycin,首次证明了微生物自抗性机制可以作为新抗生素发现的依据。第一个详细阐述自抗性基因导向的基因组挖掘研究是由Moore团队[136]在2015年完成的,该团队在86个盐孢菌基因组中筛选具有双拷贝的管家基因,定位到一个坐落在PKS-NRPS生物合成基因簇上的脂肪酸合酶,将该基因簇在天蓝色链霉菌中异源表达后,产生了一系列脂肪酸合酶天然产物抑制剂Thiolactomycins(图11)。
酪蛋白水解蛋白酶P(ClpP)在原核及真核生物的线粒体中广泛存在且高度保守。Gerard D. Wright研究团队[137]以clpP作为可能的自抗性基因在Genbank数据库中进行基因组挖掘,定位到10条基因簇,期望从中找到潜在的ClpP天然抑制剂类抗生素。其中,有6条包含了一个双模块的NRPS,该双模块NRPS广泛分布于各种放线菌和临床致病菌,簇内大多编码了一个丝氨酸水解酶,推测该基因簇产物发挥丝氨酸水解酶的抑制作用。研究者发现其中1条除了含有双模块的NRPS外还罕见地包含了一个Ⅰ型PKS,于是将该基因簇在天蓝色链霉菌(S.coelicolor M1154)中进行异源表达,分离鉴定了一个ClpP共价抑制剂Clipibicyclene(图11)。
四环素是一类具有抗菌活性的广谱抗生素,目前报道的四环素天然产物仅有5个,其中2个已作为临床用药[138]。戈惠明团队[138]对已知四环素的生物合成基因簇进行分析,发现其中均存在由转录调控因子控制外排泵表达的抗性基因tetR,利用抗性基因和骨架合成基因双探针,在放线菌基因组数据库进行挖掘,结合链延长因子的系统发育分析,定位到30条四环素生物合成基因簇。随后,通过构建基因组相似性网络(genome neighborhood network,GNN)关注到一条包含大量新颖后修饰酶的四环素基因簇,经过发酵分离得到天然产物Hainancycline(图11),是30年来首次鉴定的新型四环素类天然产物,也是目前发现的修饰最为复杂的一类四环素天然产物。
合成支链氨基酸的关键酶乙酰乳酸合酶(ALS)是许多除草剂的作用靶点。吕雪峰研究团队[139]选择ALS为探针对459个测序真菌基因组进行扫描,在亮白曲霉(A.candidus)和蘑菇(A.campestris)中发现了两条同源的NRPS-PKS生物合成基因簇与ALS共定位。通过生物信息学分析和化学表征确定该基因簇产物为黄酮类天然产物氯黄酮(图11)。活性评价显示,氯黄酮具有抑制拟南芥种子萌发和抑制病原菌生长的活性,因而具有开发成为除草剂和抗生素的潜力。
值得注意的是,为了便于研究者们开展自抗性基因导向的基因组挖掘,Nadine Ziemert团队[140]在2017年推出了一个可以在线使用的分析网站:ARTS(Antibiotic Resistant Target Seeker,https://arts.ziemertlab.com)。该网站首先使用antiSMASH考察天然产物基因簇中是否存在重复拷贝的管家基因,以及该基因的系统发育分析来确认其是否为自抗性基因,进而识别出可以产生活性天然产物的基因簇。2020年,该工具的2.0版本[141]进一步增强了分析能力,可以实现对所有细菌和宏基因组数据的自动挖掘。2022年,该团队[142]发布了ARTS-DB数据库,该数据库收录了ARTS 2.0对共计70 000多个基因组和宏基因组的分析结果,并提供了配套的查询工具,从而免去了大量冗余的重复分析。
5 进化理论在基因组挖掘中的应用
天然产物结构多样性是生物合成基因簇持续进化的结果,而构建分子系统发育树是追溯特定基因进化足迹和确定同源基因进化关系的常用手段。基于分子系统发育的天然产物发现策略正是构筑在生物合成基因可以作为基因簇进化标志的基本假设之上,从而以点及面地体现基因簇之间的进化关系以及对应产物的结构多样性和新颖性[143]。具体而言,如果一个未知功能的核心基因与已知的同源基因进化距离较远,那么它的生物合成基因簇可能编码了一个与已知天然产物骨架完全不同的新颖产物;反之,如果该基因与一些基因紧密分布在一个进化分支之内,那么这些基因所在的基因簇的产物将十分类似(图12)。
图12
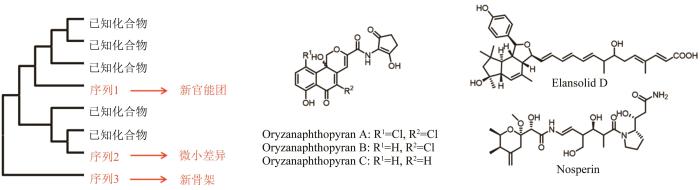
图12 构建系统发育树指导新颖天然产物的发现[143]
进化理论指导的基因组挖掘,最为成功的一个案例来自于对新型Ⅱ型聚酮的发掘。Ⅱ型聚酮基因簇中负责聚酮骨架形成的聚酮合酶KS,链长决定因子CLF和酰基载体蛋白ACP是Ⅱ型聚酮骨架合成的核心酶。张骊駻团队[144]首先以167个经表征的Ⅱ型聚酮合酶的CLF蛋白建立了系统发育树,发现聚酮产物中骨架结构上碳原子数量与CLF的系统发育密切相关。基于此,该研究团队对NCBI上RefSeq数据库中细菌基因组展开分析,对其中所有的CLF构建系统发育树,描绘了细菌产生Ⅱ型聚酮的综合潜能。随后,研究人员选择CLF系统发育树中与已知CLF进化距离较远的条目,将对应的菌株进行发酵和产物的分离鉴定,从中发现了2类新的Ⅱ型聚酮天然产物,包括一种新的角形萘吡喃骨架结构(图12)。
trans-AT聚酮合酶虽然属于Ⅰ型模块化聚酮合酶,但与经典的cis-AT聚酮合酶采取完全不同的进化方式。相较于cis-AT聚酮合酶的每个模块都嵌合了一个能够识别延伸单元的酰基转移酶结构域(AT),trans-AT聚酮合酶基因簇只编码了一个游离于聚酮合酶之外的AT蛋白,通常负责特异性识别丙二酰辅酶A。并且,trans-AT聚酮合酶的KS结构域只专一地接受不同修饰的特定底物,因此根据KS结构域的系统进化分析可以推测出其编码的聚酮骨架[145]。Jörn Piel课题组多年来对trans-AT类的天然产物开展了较为深入和系统的研究,通过持续对KS结构域系统进化分析的补充和更新,他们从一些不常见的微生物中分离得到许多活性trans-AT天然产物,包括具有抗菌活性的Elansolid D[146]、细胞毒活性的Diaphorin[147]和Nosperin[148]等(图12)。与此同时,他们还开发了一个基于KS系统进化预测trans-AT聚酮骨架的在线网站[149](http://transator.ethz.ch/trans-AT-PKS),收录了54个trans-AT聚酮合酶的655个KS结构域,进一步推动具有潜在药用价值的聚酮化合物的基因组挖掘。
6 人工智能在基因组挖掘中的应用
随着基因组大数据时代的到来,经典的基因组挖掘工具正面临着分析处理大数据的考验,同时,历经多年的发展,基因组挖掘的目标已不仅仅是从基因组信息中识别和预测已知类别天然产物的基因簇,更需要研究者们建立新的思路,以实现对全新类别天然产物基因簇的无参考式的从头识别。近十年以来,人工智能正改变着各行各业对信息的处理方式,对于基因组挖掘而言,是值得交叉融合的重要工具。
在健康的人体肠道中,旅居着数百万种微生物,这些微生物中不乏大量的致病菌,而肠道微生物群体却能够彼此制衡,和谐共存。其中一个重要的原因是肠道中的有益菌可以产生一些具有抗菌活性的小肽,称为抗菌肽(antimicrobial peptide,AMP)[150],这些小肽或其简单的修饰物具有抑制病原微生物的能力。但是面对丰富的人体微生物测序数据资源,基因组挖掘这类小肽却面临着序列短、多样性高、相似性低的难题,传统的挖掘工具无法高效和特异性地识别这些小肽的基因。陈义华和王军研究团队[151]借用人工智能中的深度学习方法为发掘抗菌肽提供了崭新的模式,研究团队构建了5种自然语言学习的神经网络,把抗菌肽的氨基酸序列作为学习的模板,建立阳性和阴性数据集用以训练深度学习的模型,让模型可以从DNA层面直接识别抗菌肽基因[图13(a)]。最终,综合利用三种神经网络模型建立的抗菌肽预测模型,从1万多个微生物组中预测出了216种潜在的新型抗菌肽,经多肽的合成和活性评价,发现其中181种新型抗菌肽具有抗菌活性,进一步实验表明,部分抗菌肽对多重耐药革兰氏阴性菌也具有较强的抑菌能力,其中,活性最好的抗菌肽c_AMP1043通过与细胞膜和细胞壁结合,诱发了细胞膜破裂,从而杀灭微生物。
图13
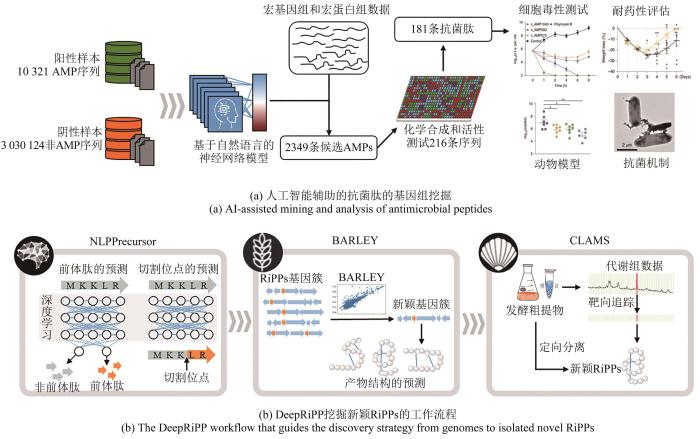
图13 人工智能在基因组挖掘中的应用
另一个利用人工智能的案例源自于针对RiPPs类天然产物的基因组挖掘,RiPPs的生物合成过程始于核糖体合成的核心肽,历经修饰加工,最终经过切割释放出最终产物[152]。而针对RiPPs的预测分析大多仍是基于已知类型中的关键修饰基因的同源性检索,这种方式很难发掘新颖类型的RiPPs。同时,在测序数据中,那些低质量的测序数据或碎片化的测序信息常被忽略,但其中很可能蕴含着大量值得分析的信息。另外,将代谢组的分析和基因水平的预测偶联为一套分析流程将能提升新颖产物的发现概率。Nathan A. Magarvey团队[47]针对此问题开发了一套由3个模块组成的RiPPs预测流程——DeepRiPP,一个集成的基因组和代谢组学的分析平台,使用机器学习来自动化选择性地发现和分离新的RiPPs[图13(b)]。第一个模块是NLPPrecursor,它使用自然语言构建了基于深度学习的预测模型,该模型分为两步,从测序数据中识别前体肽,再预测其切割位点。第二个模块BARLEY,负责对预测结果信息进行排序,以辅助研究者寻找最优价值的基因簇,而第三个模块CLAMS则可以将含有该基因簇菌株的代谢组数据与预测的产物信息进行偶联分析,自动化地从复杂的细菌提取物中识别出目标基因簇可能的产物。在研究中,研究者使用DeepRiPP对数据库中来自463个菌株的10 498个提取物展开了大规模的比较代谢组学分析,并从中发现了3种新型RiPPs,其真实结构与DeepRiPP平台预测结果完全一致,从而证明了该套分析流程的有效性和便捷性。这一方法也证明了联合使用基因组和代谢组数据,可以有效提升天然产物发掘的通量和速度。
7 总结与展望
具有巨大化学多样性的天然产物为药物开发提供了无可比拟的小分子文库,从中诞生了大量的药物或先导化合物。由于众多抗生素药物集中发现于20世纪50年代左右,这一时期被称为天然产物发现的“黄金时代”[153]。然而,自20世纪后期以来,天然产物来源的药物出现了大幅下降,这一方面是由于小分子化学合成技术的进步和高通量筛选平台的出现,更重要的是,传统的天然产物发现策略导致了大量高丰度化合物的重复出现,而低丰度或不可培养微生物中的资源却难以得到发掘[154]。随着基因组时代的到来,研究者开始逐步阐明天然产物的生物合成过程,使得天然产物的结构可以与负责编码其生物合成的基因形成对应关系,由此,通过基因组信息确定合成基因(簇)进而发现天然产物的基因组挖掘策略得以建立,该策略配合下游高效的异源表达平台,可以突破对生产者生物量的依赖,为天然产物的发现开辟了新的途径。
基因组挖掘的实施离不开高效便捷的工具软件,在基因组挖掘的前期,受限于测序成本,较小的细菌基因组增长最快,造成了大量天然产物基因(簇)预测软件应对的均是细菌基因组。与细菌相比,针对真核生物,尤其是植物的生物合成研究存在着更大的挑战。植物基因组庞大,遗传背景复杂,难以获取高质量基因组序列,并且大多数情况下天然产物生物合成基因并不成簇分布在植物基因组中。因此,针对真菌和其他高等生命体的生物合成基因预测平台发展缓慢,虽然目前已经出现了针对真菌基因组特点设计的fungiSMASH[155]和针对植物基因组特点设计的plantiSMASH[129],这些软件目前可识别的基因簇类型仍然较少,错误率较高,但随着更多真菌与植物来源天然产物的生物合成案例的阐明,和计算机性能的逐步提升,针对真菌和其他高等生命体开发的软件和工具将会更加全面和完善。
此外,基因组挖掘正在不断吸纳其他学科的最新技术和发展成果。首先是“微生物组学”带来了大量的宏基因组测序数据,如人体微生物组计划[27, 156]、海洋微生物组研究[157]等有效获取了大量不可培养微生物的遗传信息,探明了潜藏其中的天然产物合成潜能。其次,测序技术的不断迭代和成本的进一步降低,使得众多植物和动物的遗传信息得以公布,基因组、转录组、代谢组和蛋白质组学等多组学(multi-omics)联合分析与基因组挖掘的有效融合促使一些高等生命体来源的天然产物生物合成基因(簇)相继得到阐明,如通过对南方红豆杉(Taxuschinensis var.mairei)基因组进化分析和转录组分析确定参与紫杉醇生物合成的关键基因[158-161];通过基于两种益母草的比较基因组学和代谢组学等多种手段找到合成益母草碱的关键酶并阐明生物合成路径[162];通过分析石松科植物(Phlegmariurustetrastichus)不同组织的转录差异表达定位参与石松生物碱生物合成的新型类碳酸酐样酶[163];对海洋蠕虫基因组进行测序组装结合宏转录组和蛋白质组学分析找到蠕虫体内植物甾醇从头合成所需的生物合成基因[164];通过全基因组关联图谱定位决定鹦鹉羽毛颜色的关键聚酮合酶[165]以及人体内由“病毒抑制蛋白”催化产生具有抗病毒活性的小分子核糖核苷酸衍生物[166]等,这为基因组挖掘提供了物种范围更为广泛的研究素材,预计高等真核生物的基因组挖掘会在不久的未来得到迅速发展,并为天然小分子药物的发现提供更加丰富多样的先导化合物。再次,合成生物学在最近的二十年里发展迅速,一方面催生了一大批可用于基因表达的底盘和元件,为基因簇的异源表达或原位激活提供了技术保障[167-168];另一方面,DNA合成技术的成熟和成本的降低,让研究者们可以批量直接合成难以获得的基因资源。最后,人工智能在近几年的迅速崛起,已经深刻影响了合成生物学的发展,人工智能在处理大数据和发现潜在规律上的绝对优势必将深刻影响基因组挖掘的思路和方法,深度学习在抗菌肽的基因组挖掘之中已经展现了其广阔的应用前景[151]。相应地,以药物分子为导向的基因组挖掘,在发现小分子先导化合物的同时,也同步促进了大量新颖酶学机制和小分子合成通路的阐明,这为天然药物的仿生化学合成和下游的生物工程生产提供了新的酶学素材和合成路径上的设计思路[169]。这些有机小分子的发现,同样可以为产生宿主本身在生态位中的功能和互作提供了分子层面的切入点[170-172]。
总而言之,基于目前对大规模基因组数据的分析,无论是微生物还是更高等的植物和动物,基因组信息中蕴藏着无可估量的塑造天然小分子的能力,这是药物发现的分子宝库,而挖掘这一宝藏,需要研究者们进一步促进基因组挖掘与其他学科间的交叉融合,进一步提升对遗传信息的处理和分析能力,增强下游的基因簇表达通量和产物的结构预测能力,从而实现天然小分子高通量、高新颖性和高效率的发现,为开发具有自主知识产权的新药物、新化学品和新型酶催化剂服务。





)


)
--- 使用聚合函数时需要注意的点点滴滴)




环境搭建)


)

