
论文地址:Biosensors | Free Full-Text | Feature-Based Information Retrieval of Multimodal Biosignals with a Self-Similarity Matrix: Focus on Automatic Segmentation (mdpi.com)
论文源码:无
期刊:biosensors
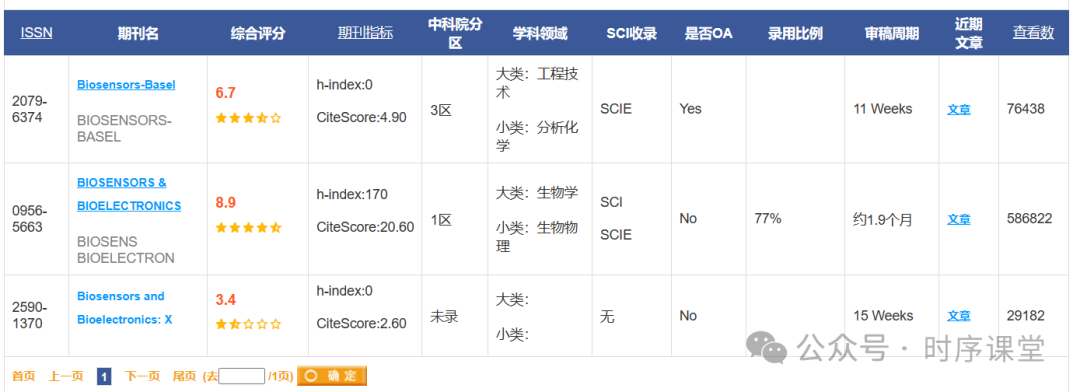

这篇论文提出了一种基于特征的信息检索方法,利用自相似矩阵(SSM)自动分割多模态生物信号数据。随着可穿戴生物传感器技术的发展,获取连续的生物信号数据变得更加容易,但这也带来了如何从大量数据中提取有用信息的挑战。为了解决这一问题,作者设计了一种算法,通过计算信号的特征表示来构建SSM,从而揭示信号内在的结构和周期性模式。
该方法主要包含三个步骤:特征提取、构建SSM和信息检索。特征提取步骤使用滑动窗口技术从时间序列中提取统计、时域和频域特征。接着,利用这些特征构建SSM,SSM中的每个元素表示时间序列中两个子序列之间的相似度。最后,通过分析SSM来检索信息,包括使用新颖性搜索确定信号的变化点、周期性搜索识别重复模式,以及通过相似性分析比较不同子序列或段落之间的相似度。
此方法的核心优势在于其自动化和通用性,能够适用于单通道或多模态生物信号数据,支持无监督的信号分析。通过公共生物信号数据集的实验验证,该方法在自动分割生物信号方面显示了优越的性能,特别是在识别周期性事件和信号变化点方面。此外,SSM的视觉支持为理解和解释生物信号提供了直观的帮助,增强了分析的可解释性。这项工作为生物信号处理和自动化分析提供了新的视角和工具,有望在健康监测、运动科学和康复辅助等领域得到应用。

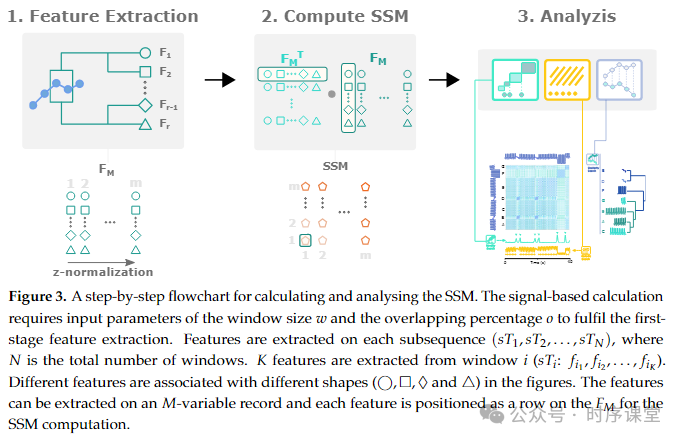
关键步骤:
1. 特征提取:使用滑动窗口技术在时间序列数据上滑动,根据窗口大小和重叠百分比,从每个窗口中提取出统计、时域和频域的特征。
2. 构建自相似矩阵(SSM):利用提取的特征构建SSM,其中矩阵中的每个元素代表时间序列中两个子序列之间的相似度。
3.信息检索:
新颖性搜索:在SSM的对角线上应用一个特定的核(如棋盘格模式的核),通过滑动这个核并计算新颖性函数来识别变化点,即时间序列行为的显著变化。
周期性搜索:通过计算SSM的每一列的总和来生成相似性函数,从中识别出周期性模式的开始点,即局部最小值。
相似性分析:分析SSM中的相似性轮廓,比较不同子序列之间的相似度,用于进一步的分析和处理。

根据案例进行说明:

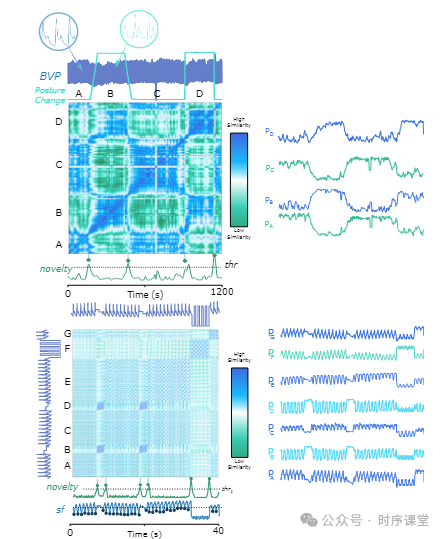

该论文提出了一种基于自相似矩阵(SSM)的多模态生物信号自动分割方法。通过滑动窗口技术提取时间序列的特征,并构建SSM来揭示信号的内在结构和周期性。该方法包括新颖性搜索以识别信号变化点,周期性搜索以发现重复模式,以及相似性分析以比较子序列间的相似度。这种方法的自动化和通用性使其在健康监测、运动科学等领域具有潜在的应用价值,为生物信号处理提供了一个有效的分析工具。
论文下载地址:
链接:https://pan.baidu.com/s/19vw0ZoVirnGEFcDSdv1Nqw
提取码:1msi
--来自百度网盘超级会员V6的分享
)

——存储策略)


)

)


)








