[医学分割大模型系列] -3- SAM-Med3D 分割大模型解析
- 1. 特点
- 2. 背景
- 3. 训练数据集
- 3.1 数据集收集
- 3.2 数据清洗
- 3.3 模型微调数据集
- 4. 模型结构
- 4.1 3D Image Encoder
- 4.2 3D Prompt Encoder
- 4.3 3D mask Decoder
- 4.4 模型权重
- 5. 评估
- 5.1 评估数据集
- 5.2 Quantitative Evaluation
- 5.3 可视化
- 6. 结论
论文地址:SAM-Med3D
开源地址:https://github.com/uni-medical/SAM-Med3D
发表日期:2023年10月
参考资料:
- 王皓宇(上海交通大学)SAM-Med3D基于SAM构建3D医学影像通用分割模型
- SAM-Med3D:三维医学图像上的通用分割模型,医疗版三维 SAM 开源了!
- SAM-Med3D (SJTU 2024)
1. 特点
- 通用分割能力:在各种3D目标上精准分割,效果明显优于SAM,SAM-Med2D(相对于切片进行2D分割)
- 更高的效率:比现有通用分割模型更快,提示需求更少(相对于切片进行2D分割)
- 迁移能力:作为预训练模型,在多个任务上效果良好
- 模型输入:要分割的图像和一个/几个提示点(提示点越多,效果越好)
- 模型输出:分割结果
- 数据集:SAM-Med3D-130K数据集,拥有 131K 3D mask和 247 个类别
- 网络结构:类SAM,将结构换成3D版本
- 分割对象:3D医学图像
2. 背景
- 3D医学图像:体素形式的3D图像和标注,以不同分布的灰度图像为主
- 任务特定模型的局限:
- 沉重的训练负担:使用U-Net,UNETR等分割网络在医学数据集上训练,使用A100也需要2-7天
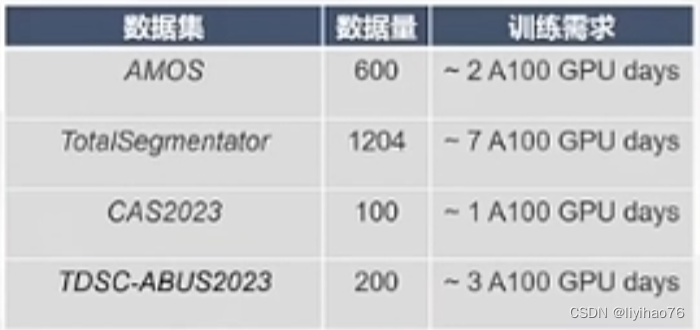
- 泛化性弱
使用特定数据集训练出来的模型(左列)在其他数据集上的表现(行)不佳
- 沉重的训练负担:使用U-Net,UNETR等分割网络在医学数据集上训练,使用A100也需要2-7天
- SAM在3D医学分割的局限:
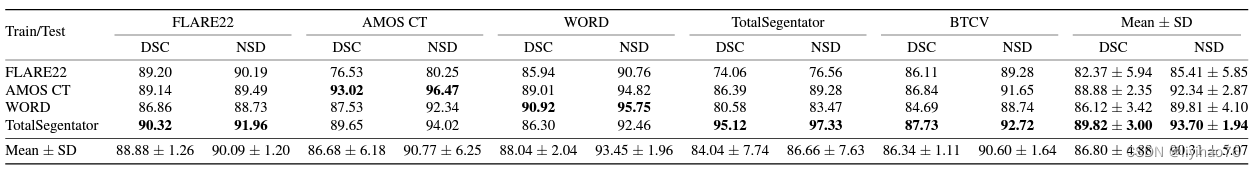
- 由于医学图像知识的严重不足,将 SAM 直接应用于医学领域的有效性有限。解决这个问题的一种直接的方法是:将医学知识融入到 SAM 中。比如,MedSAM 是一种典型示例,它通过使用110万个掩码(mask)对SAM 的解码器(Mask Decoder)进行微调,从而使 SAM 能够通过边界框(Bounding Box)作为提示来更好地分割医学影像;SAM-Med2D 则引入了适配器(Adapter)和约2000万个掩码(mask)对 SAM 进行了充分微调,从而在医学图像分割中表现出了卓越的性能。
- 然而,这些方法必须采用逐切片(slice)的方法来处理三维医学图像,也即,将三维数据从某个维度分解为二维切片,然后独立处理每个切片,最后将二维分割结果汇总为三维分割结果。这种方法忽略了切片之间的三维空间信息,因此在三维医学影像上表现不佳,这一问题可以从上图中的结果看出。SAM和SAM-Med2D都是一张张切片进行分割,每张切片都需要一个提示,所以总共需要N个提示。对于一些切片,他们的表现不佳,从而导致空间信息的不连贯。
- 除了将 SAM 直接应用于三维数据,一些研究人员希望通过引入二维到三维的适配器(Adapter)来捕捉三维空间信息。这些方法通常在保持编码器(Image Encoder)不变的同时引入了三维适配器(Adapter),以使模型能够从三维图像中学习到三维空间信息。然而,这些方法存在两个主要限制:(1)数据规模有限:这些方法的模型通常只在有限的数据规模下进行训练(通常在1K到25K个mask范围内),并且只针对有限的目标类型。这限制了模型的泛化性能和适用范围。(2)冻结的二维编码器:现有的三维 SAM-based 模型一直坚守着冻结原始二维 SAM 编码器(Image Encoder)的设计范式,这限制了模型全面建模三维空间信息的能力,大大限制了 SAM 在三维医学图像处理领域的发展潜力。
3. 训练数据集
3.1 数据集收集
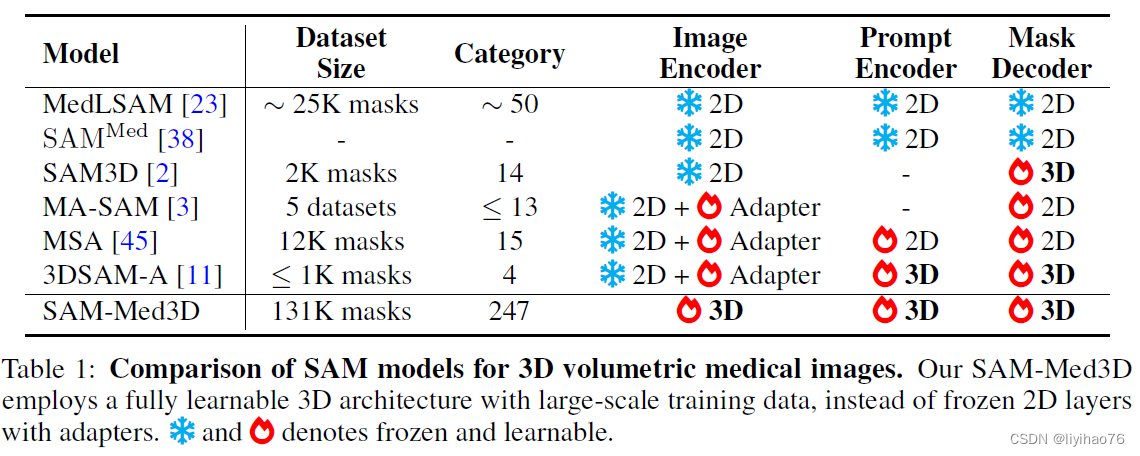
 作者进行了三维医学图像数据集的广泛收集和标准化工作,整合了116个公开和私有的三维医学图像数据集,经过4轮数据筛选和清晰,创建了迄今为止规模最大的三维医学图像分割数据集。该数据集包含了 2.1 万个三维医学图像(病人数量)和 13.1 万个三维掩码(mask)。从下表可以清晰地看出,这一数据集的规模远远超过了现有最大的三维医学图像分割数据集,如 TotalSegmentator 和 BraTS21,其规模扩大了 10 倍以上。
作者进行了三维医学图像数据集的广泛收集和标准化工作,整合了116个公开和私有的三维医学图像数据集,经过4轮数据筛选和清晰,创建了迄今为止规模最大的三维医学图像分割数据集。该数据集包含了 2.1 万个三维医学图像(病人数量)和 13.1 万个三维掩码(mask)。从下表可以清晰地看出,这一数据集的规模远远超过了现有最大的三维医学图像分割数据集,如 TotalSegmentator 和 BraTS21,其规模扩大了 10 倍以上。
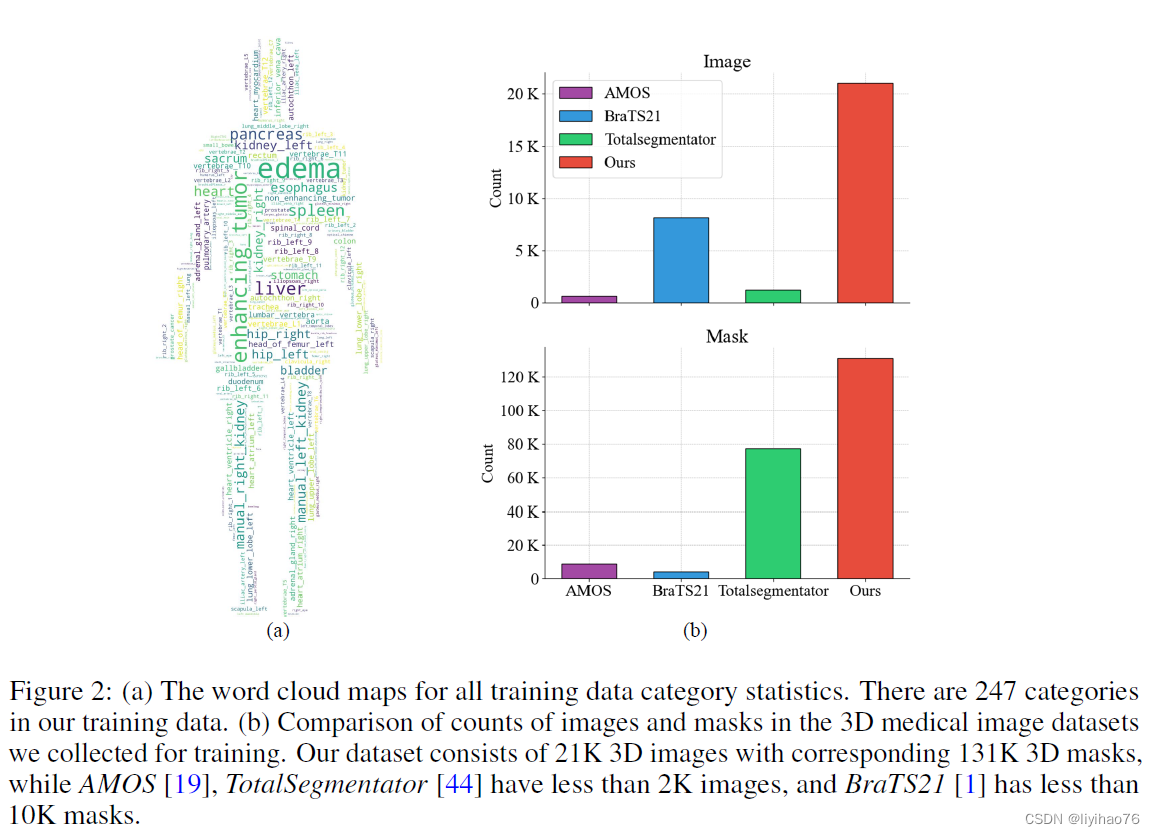
该数据集涵盖 27 种模态(CT 和 26 种MRI 序列)和 7 种解剖结构。如下图所⽰,共涵盖了 247 个不同的类别,包括器官和病变。
3.2 数据清洗
四步数据清洗:
- 基于元信息的数据清理 我们首先总结了所收集数据的元信息,包括每张医学影像的深度、宽度和高度。我们删除了所有物理尺寸小于 1 立方厘米或任何单个尺寸小于 1.5 厘米的病例,以确保目标mask的可见性。
- 基于连接域的掩码清理 在计算连通域的过程中,我们首先将原始的多类mask分割成多个类别的单击格式。然后,我们计算每个单击掩码的前 5 个最大连通域的大小和背景。根据这些掩码的汇总信息,我们会删除背景占整个体积 99% 以上的mask。
- 基于连接域的标签质量改进 对于过滤后的mask,我们设计了一个基于连接域的pipeline来提高标签质量。根据每个mask的前 5 个最大连通域的汇总信息,我们只需删除小于这 5 个连通域的任何其他域,以减少噪音。
- 基于对称性的标签质量改进 最后,我们将一些对称目标的mask拆分为不同类别的成对mask。例如,我们将 "肾 "的mask分为 "左肾 "和 “右肾”。这一步的目的是加强不同类别mask的语义一致性,防止模型分不清是分割整个结构还是只分割单个的左右部分。为了解决这个问题,SAM 为每个提示生成多个预测,并采用额外的头部生成分数,以方便选择最合适的预测。鉴于医学图像的mask通常不那么模糊,我们选择直接处理数据来消除这种模糊性,从而增强mask类别之间的语义一致性,降低网络训练的复杂性。
3.3 模型微调数据集
目前SAM-Med3D-turbo是现已发布经过微调的 SAM-Med3D 的最新版本checkpoint。在SAM-Med3D的基础上又在 44 个数据集 ( 以下list )上对其进行了微调以提高性能。
AMOS2022
ATM2022
AbdomenCT1K
BTCV_Cervix
BraTS2020
BraTS2021
BrainTumour
Brain_PTM
CAUSE07
CHAOS_Task_4
COSMOS2022
COVID19CTscans
CTPelvic1k
CT_ORG
FLARE21
FLARE22
Heart_Seg_MRI
ISLES_SISS
ISLES_SPES
KiPA22
KiTS
KiTS2021
LAScarQS22_task1
LAScarQS22_task2
LITS
MMWHS
MSD_Colon
MSD_HepaticVessel
MSD_Liver
MSD_Pancreas
MSD_Prostate
MSD_Spleen
PROMISE12
Parse22
Promise09
Prostate_MRI_Segmentation_Dataset
SLIVER07
STACOM_SLAWT
SegThor
Totalsegmentator_dataset
VESSEL2012
VerSe19
VerSe20
WORD
4. 模型结构
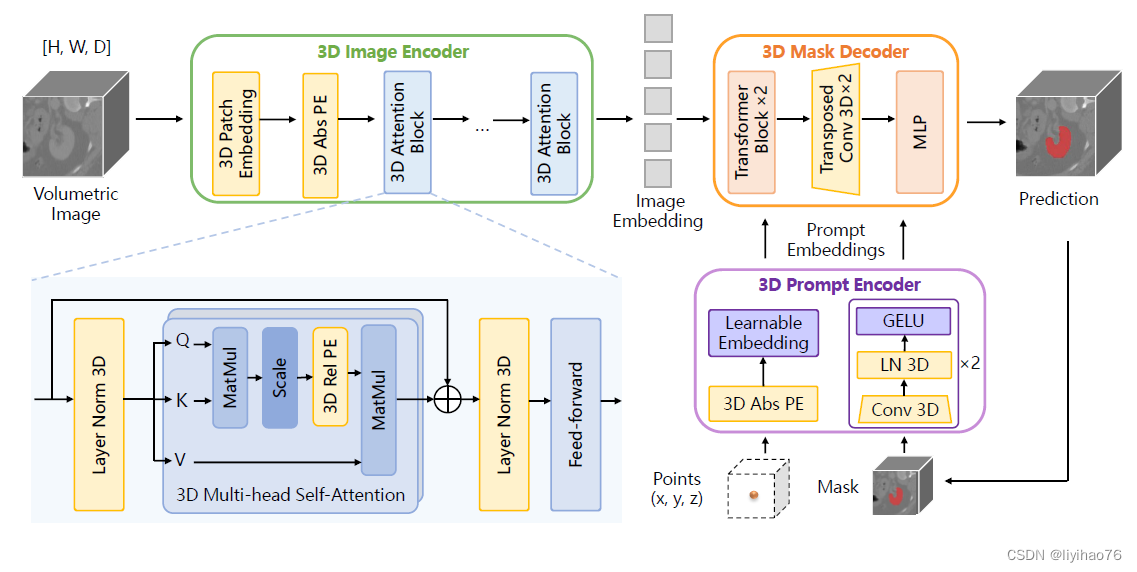
基于SAM修改后SAM-Med3D 的 3D 架构。 原始2D组件被转换为3D对应组件,包括3D Image Encoder、3D Prompt Encoder 和3D mask Decoder。采用3D卷积、3D位置编码(PE)和3D layer norm来构建3D模型。
4.1 3D Image Encoder
在 3D 图像编码器中,首先使用内核大小为 (16, 16, 16) 的 3D 卷积嵌入块生成embedding,并与可学习的 3D 绝对位置编码 absolute Positional Encoding (PE) 配对。 这种编码是通过自然地将附加维度扩展到 SAM 的 2D PE 来获得的。 然后将补丁的嵌入输入到 3D 注意力块中。 对于 3D 注意力模块,我们将 3D 相关 PE 合并到 SAM 的多头自注意力(MHSA)模块中,使其能够直接捕获空间细节。
class PatchEmbed3D(nn.Module):"""Image to Patch Embedding."""def __init__(self,kernel_size: Tuple[int, int] = (16, 16, 16),stride: Tuple[int, int] = (16, 16, 16),padding: Tuple[int, int] = (0, 0, 0),in_chans: int = 1,embed_dim: int = 768,) -> None:"""Args:kernel_size (Tuple): kernel size of the projection layer.stride (Tuple): stride of the projection layer.padding (Tuple): padding size of the projection layer.in_chans (int): Number of input image channels.embed_dim (int): Patch embedding dimension."""super().__init__()self.proj = nn.Conv3d(in_chans, embed_dim, kernel_size=kernel_size, stride=stride, padding=padding)def forward(self, x: torch.Tensor) -> torch.Tensor:x = self.proj(x)# B C X Y Z -> B X Y Z Cx = x.permute(0, 2, 3, 4, 1)return x
class Attention(nn.Module):"""Multi-head Attention block with relative position embeddings."""def __init__(self,dim: int,num_heads: int = 8,qkv_bias: bool = True,use_rel_pos: bool = False,rel_pos_zero_init: bool = True,input_size: Optional[Tuple[int, int, int]] = None,) -> None:"""Args:dim (int): Number of input channels.num_heads (int): Number of attention heads.qkv_bias (bool): If True, add a learnable bias to query, key, value.rel_pos (bool): If True, add relative positional embeddings to the attention map.rel_pos_zero_init (bool): If True, zero initialize relative positional parameters.input_size (tuple(int, int) or None): Input resolution for calculating the relativepositional parameter size."""super().__init__()self.num_heads = num_headshead_dim = dim // num_headsself.scale = head_dim**-0.5self.qkv = nn.Linear(dim, dim * 3, bias=qkv_bias)self.proj = nn.Linear(dim, dim)self.use_rel_pos = use_rel_posif self.use_rel_pos:assert (input_size is not None), "Input size must be provided if using relative positional encoding."# initialize relative positional embeddingsself.rel_pos_d = nn.Parameter(torch.zeros(2 * input_size[0] - 1, head_dim))self.rel_pos_h = nn.Parameter(torch.zeros(2 * input_size[1] - 1, head_dim))self.rel_pos_w = nn.Parameter(torch.zeros(2 * input_size[2] - 1, head_dim))def forward(self, x: torch.Tensor) -> torch.Tensor:B, D, H, W, _ = x.shape# qkv with shape (3, B, nHead, H * W, C)qkv = self.qkv(x).reshape(B, D * H * W, 3, self.num_heads, -1).permute(2, 0, 3, 1, 4)# q, k, v with shape (B * nHead, H * W, C)q, k, v = qkv.reshape(3, B * self.num_heads, D * H * W, -1).unbind(0)attn = (q * self.scale) @ k.transpose(-2, -1)if self.use_rel_pos:attn = add_decomposed_rel_pos(attn, q, self.rel_pos_d, self.rel_pos_h, self.rel_pos_w, (D, H, W), (D, H, W))attn = attn.softmax(dim=-1)x = (attn @ v).view(B, self.num_heads, D, H, W, -1).permute(0, 2, 3, 4, 1, 5).reshape(B, D, H, W, -1)x = self.proj(x)return x
4.2 3D Prompt Encoder
在提示编码器中,稀疏提示利用 3D 位置编码来表示 3D 空间细微差别,而密集提示则通过 3D 卷积进行处理。
class PromptEncoder3D(nn.Module):def __init__(self,embed_dim: int,image_embedding_size: Tuple[int, int, int],input_image_size: Tuple[int, int, int],mask_in_chans: int,activation: Type[nn.Module] = nn.GELU,) -> None:"""Encodes prompts for input to SAM's mask decoder.Arguments:embed_dim (int): The prompts' embedding dimensionimage_embedding_size (tuple(int, int)): The spatial size of theimage embedding, as (H, W).input_image_size (int): The padded size of the image as inputto the image encoder, as (H, W).mask_in_chans (int): The number of hidden channels used forencoding input masks.activation (nn.Module): The activation to use when encodinginput masks."""super().__init__()self.embed_dim = embed_dimself.input_image_size = input_image_sizeself.image_embedding_size = image_embedding_sizeself.pe_layer = PositionEmbeddingRandom3D(embed_dim // 3)self.num_point_embeddings: int = 2 # pos/neg pointpoint_embeddings = [nn.Embedding(1, embed_dim) for i in range(self.num_point_embeddings)]self.point_embeddings = nn.ModuleList(point_embeddings)self.not_a_point_embed = nn.Embedding(1, embed_dim)self.mask_input_size = (image_embedding_size[0], image_embedding_size[1], image_embedding_size[2])self.mask_downscaling = nn.Sequential(nn.Conv3d(1, mask_in_chans // 4, kernel_size=2, stride=2),LayerNorm3d(mask_in_chans // 4),activation(),nn.Conv3d(mask_in_chans // 4, mask_in_chans, kernel_size=2, stride=2),LayerNorm3d(mask_in_chans),activation(),nn.Conv3d(mask_in_chans, embed_dim, kernel_size=1),)self.no_mask_embed = nn.Embedding(1, embed_dim)def get_dense_pe(self) -> torch.Tensor:"""Returns the positional encoding used to encode point prompts,applied to a dense set of points the shape of the image encoding.Returns:torch.Tensor: Positional encoding with shape1x(embed_dim)x(embedding_h)x(embedding_w)"""return self.pe_layer(self.image_embedding_size).unsqueeze(0) # 1xXxYxZdef _embed_points(self,points: torch.Tensor,labels: torch.Tensor,pad: bool,) -> torch.Tensor:"""Embeds point prompts."""points = points + 0.5 # Shift to center of pixelif pad:padding_point = torch.zeros((points.shape[0], 1, 3), device=points.device)padding_label = -torch.ones((labels.shape[0], 1), device=labels.device)points = torch.cat([points, padding_point], dim=1)labels = torch.cat([labels, padding_label], dim=1)point_embedding = self.pe_layer.forward_with_coords(points, self.input_image_size)point_embedding[labels == -1] = 0.0point_embedding[labels == -1] += self.not_a_point_embed.weightpoint_embedding[labels == 0] += self.point_embeddings[0].weightpoint_embedding[labels == 1] += self.point_embeddings[1].weightreturn point_embeddingdef _embed_boxes(self, boxes: torch.Tensor) -> torch.Tensor:"""Embeds box prompts."""boxes = boxes + 0.5 # Shift to center of pixelcoords = boxes.reshape(-1, 2, 2)corner_embedding = self.pe_layer.forward_with_coords(coords, self.input_image_size)corner_embedding[:, 0, :] += self.point_embeddings[2].weightcorner_embedding[:, 1, :] += self.point_embeddings[3].weightreturn corner_embeddingdef _embed_masks(self, masks: torch.Tensor) -> torch.Tensor:"""Embeds mask inputs."""mask_embedding = self.mask_downscaling(masks)return mask_embeddingdef _get_batch_size(self,points: Optional[Tuple[torch.Tensor, torch.Tensor]],boxes: Optional[torch.Tensor],masks: Optional[torch.Tensor],) -> int:"""Gets the batch size of the output given the batch size of the input prompts."""if points is not None:return points[0].shape[0]elif boxes is not None:return boxes.shape[0]elif masks is not None:return masks.shape[0]else:return 1def _get_device(self) -> torch.device:return self.point_embeddings[0].weight.devicedef forward(self,points: Optional[Tuple[torch.Tensor, torch.Tensor]],boxes: Optional[torch.Tensor],masks: Optional[torch.Tensor],) -> Tuple[torch.Tensor, torch.Tensor]:"""Embeds different types of prompts, returning both sparse and denseembeddings.Arguments:points (tuple(torch.Tensor, torch.Tensor) or none): point coordinatesand labels to embed.boxes (torch.Tensor or none): boxes to embedmasks (torch.Tensor or none): masks to embedReturns:torch.Tensor: sparse embeddings for the points and boxes, with shapeBxNx(embed_dim), where N is determined by the number of input pointsand boxes.torch.Tensor: dense embeddings for the masks, in the shapeBx(embed_dim)x(embed_H)x(embed_W)"""bs = self._get_batch_size(points, boxes, masks)sparse_embeddings = torch.empty((bs, 0, self.embed_dim), device=self._get_device())if points is not None:coords, labels = pointspoint_embeddings = self._embed_points(coords, labels, pad=(boxes is None))sparse_embeddings = torch.cat([sparse_embeddings, point_embeddings], dim=1)if boxes is not None:box_embeddings = self._embed_boxes(boxes)sparse_embeddings = torch.cat([sparse_embeddings, box_embeddings], dim=1)if masks is not None:dense_embeddings = self._embed_masks(masks)else:dense_embeddings = self.no_mask_embed.weight.reshape(1, -1, 1, 1, 1).expand(bs, -1, self.image_embedding_size[0], self.image_embedding_size[1], self.image_embedding_size[2])return sparse_embeddings, dense_embeddings
4.3 3D mask Decoder
3D mask Decoder与 3D 上采样集成,采用 3D 转置卷积。
class TwoWayAttentionBlock3D(nn.Module):def __init__(self,embedding_dim: int,num_heads: int,mlp_dim: int = 2048,activation: Type[nn.Module] = nn.ReLU,attention_downsample_rate: int = 2,skip_first_layer_pe: bool = False,) -> None:"""A transformer block with four layers: (1) self-attention of sparseinputs, (2) cross attention of sparse inputs to dense inputs, (3) mlpblock on sparse inputs, and (4) cross attention of dense inputs to sparseinputs.Arguments:embedding_dim (int): the channel dimension of the embeddingsnum_heads (int): the number of heads in the attention layersmlp_dim (int): the hidden dimension of the mlp blockactivation (nn.Module): the activation of the mlp blockskip_first_layer_pe (bool): skip the PE on the first layer"""super().__init__()self.self_attn = Attention(embedding_dim, num_heads)self.norm1 = nn.LayerNorm(embedding_dim)self.cross_attn_token_to_image = Attention(embedding_dim, num_heads, downsample_rate=attention_downsample_rate)self.norm2 = nn.LayerNorm(embedding_dim)self.mlp = MLPBlock3D(embedding_dim, mlp_dim, activation)self.norm3 = nn.LayerNorm(embedding_dim)self.norm4 = nn.LayerNorm(embedding_dim)self.cross_attn_image_to_token = Attention(embedding_dim, num_heads, downsample_rate=attention_downsample_rate)self.skip_first_layer_pe = skip_first_layer_pedef forward(self, queries: Tensor, keys: Tensor, query_pe: Tensor, key_pe: Tensor) -> Tuple[Tensor, Tensor]:# Self attention blockif self.skip_first_layer_pe:queries = self.self_attn(q=queries, k=queries, v=queries)else:q = queries + query_peattn_out = self.self_attn(q=q, k=q, v=queries)queries = queries + attn_outqueries = self.norm1(queries)# Cross attention block, tokens attending to image embeddingq = queries + query_pek = keys + key_peattn_out = self.cross_attn_token_to_image(q=q, k=k, v=keys)queries = queries + attn_outqueries = self.norm2(queries)# MLP blockmlp_out = self.mlp(queries)queries = queries + mlp_outqueries = self.norm3(queries)# Cross attention block, image embedding attending to tokensq = queries + query_pek = keys + key_peattn_out = self.cross_attn_image_to_token(q=k, k=q, v=queries)keys = keys + attn_outkeys = self.norm4(keys)return queries, keys4.4 模型权重
测试了三种训练策略,结果表明从头训练效果最好
- 沿用2d sam,加上3d adapter进行改造。
- 将2d sam的权重改造成 3d 结构可以使用的权重(对3d层采用权重复制策略)。 以卷积为例,我们将二维卷积的核复制D次并将它们堆叠起来形成三维卷积,其中D表示第三维中核的大小。
- 使用3d数据从头训练。

5. 评估
对于2D切片分割和3D体积分割,我们从前景中随机采样一个点作为第一个提示,并从误差区域中随机选择以下点。 值得注意的是,2D SAM 方法(SAM、SAM-Med2D)是逐片推断的,而我们的 SAM-Med3D 使用基于补丁的推断方法进行操作。 这与 nnUNet 等最先进的医学图像分割方法一致,赋予 SAM-Med3D 在推理时间方面的优势。 此外,2D方法在推断3D医学图像时对每个切片进行独立交互,而3D方法仅在体积上进行全局交互。 这意味着2D执行的交互次数实际上是3D的N倍(N表示包含对象的切片数量,通常范围为10到200)。 尽管 2D 方法采用了更多的提示点,但其固有的片间交互缺乏造成了明显的性能上限,特别是在相对复杂的 3D 结构上。
5.1 评估数据集
在评估阶段,我们选择了 13 个公共基准数据集来审查各种临床场景,并纳入了 MICCAI2023 挑战赛中的 2 个额外数据集来验证不同模型的性能。 该验证集包含七个重要的解剖结构,例如胸部和腹部器官、大脑结构、骨骼等。 它还包括医学领域非常感兴趣的五种病变类型,以及一系列体积测量模式,包括 CT、US(超声)和八个 MRI 序列。 此外,它还包含具有挑战性的、以前未见过的目标,最终形成了不同类别的 153 个不同目标。 验证集有三部分:

5.2 Quantitative Evaluation
- 整体表现
SAM-Med3D在使用更少点击次数的情况下,获得了更好的性能。N表示待分割目标包含的切片(slice)数目,通常10 ≤ N ≤ 200。 T i n f T_{inf} Tinf为N =100时所需的推理时间 (Inference time) 。
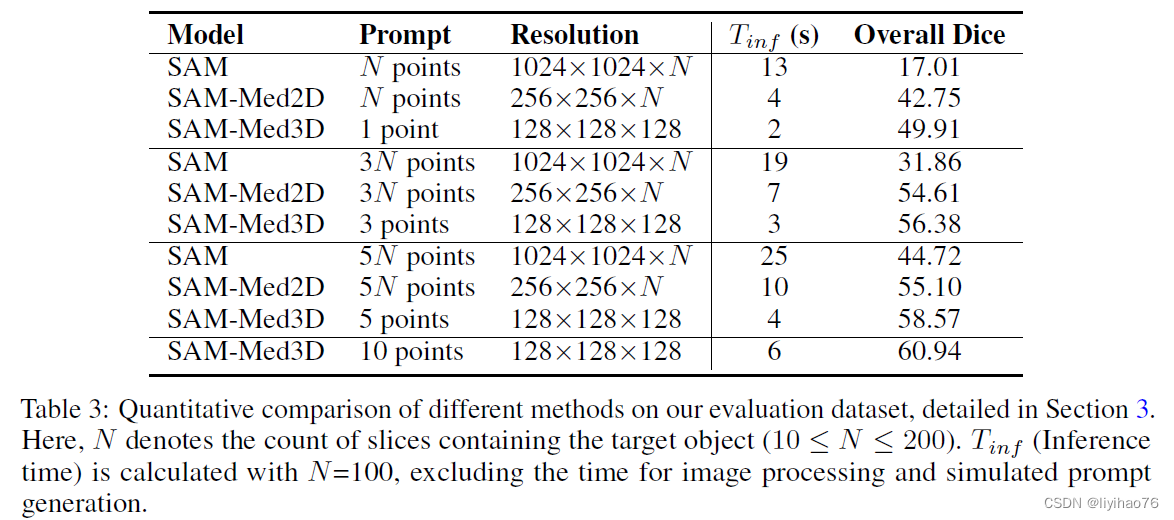
- 从解剖结构和病变角度进行评估
A&T 表示腹部和胸部。SAM-Med3D 只需10个提示点(最后一行)即可取得比 SAM 和 SAM-Med2D 更好的性能,而后两者往往需要上百个提示点。在评估中,我们考虑了各种⽅法中可见和不可见(zero-shot)的病变。对于不可见的病变,当提示有限时,表现次优。
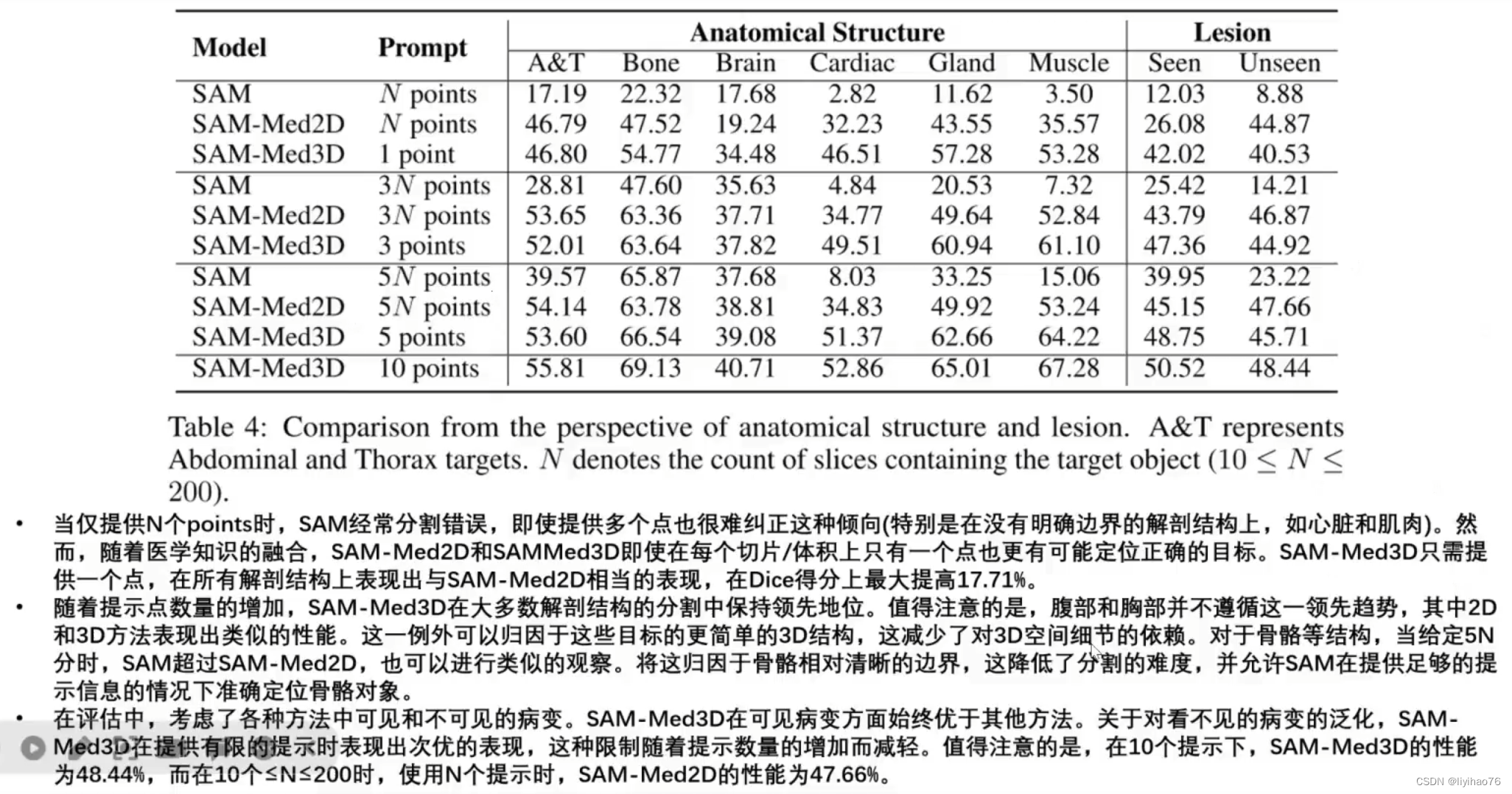
左侧三张图展示了不同模型在不同模态下的性能对比,其中SAM-Med3D在所有模态下均展现出优异性能。即使SAM-Med3D没有使用超声(US)图像训练,其性能仍与 SAM-Med2D相当。
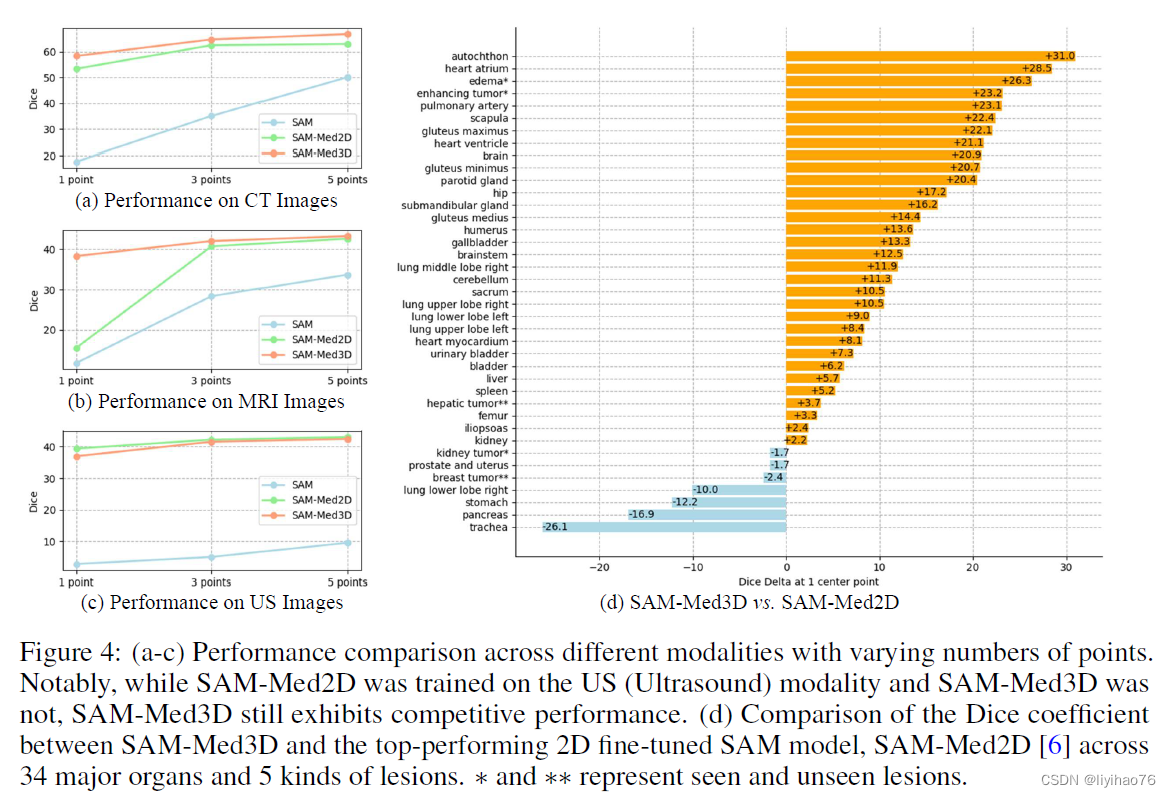
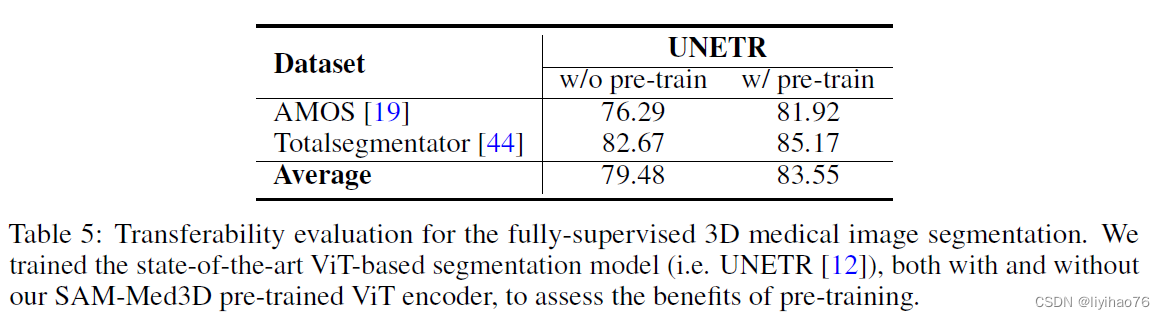
- 迁移性评估
作者将 SAM-Med3D 预训练的 ViT 图像编码器迁移到 UNETR 中进行使用,发现能够获得效果上的提升,证明了作者提出的 SAM-Med3D 具有迁移能力,这将能够对三维医学图像领域的发展提供帮助。据我们所知,SAM-Med3D 可能被定位为第一个基于 ViT 的 3D 医学图像基础模型。
5.3 可视化
图五:在不同的解剖结构中,针对不同数量的点,对SAM、SAM-Med2D和SAM-Med3D进行可视化。作者同时展示了轴切片和冠状切片/矢状切片来全面说明三维结果。
图六:在各种模态下,针对不同的点数,对SAM、SAM-Med2D和SAM-Med3D进行可视化。作者同时展示了轴切片和冠状/矢状切片来全面说明三维结果。
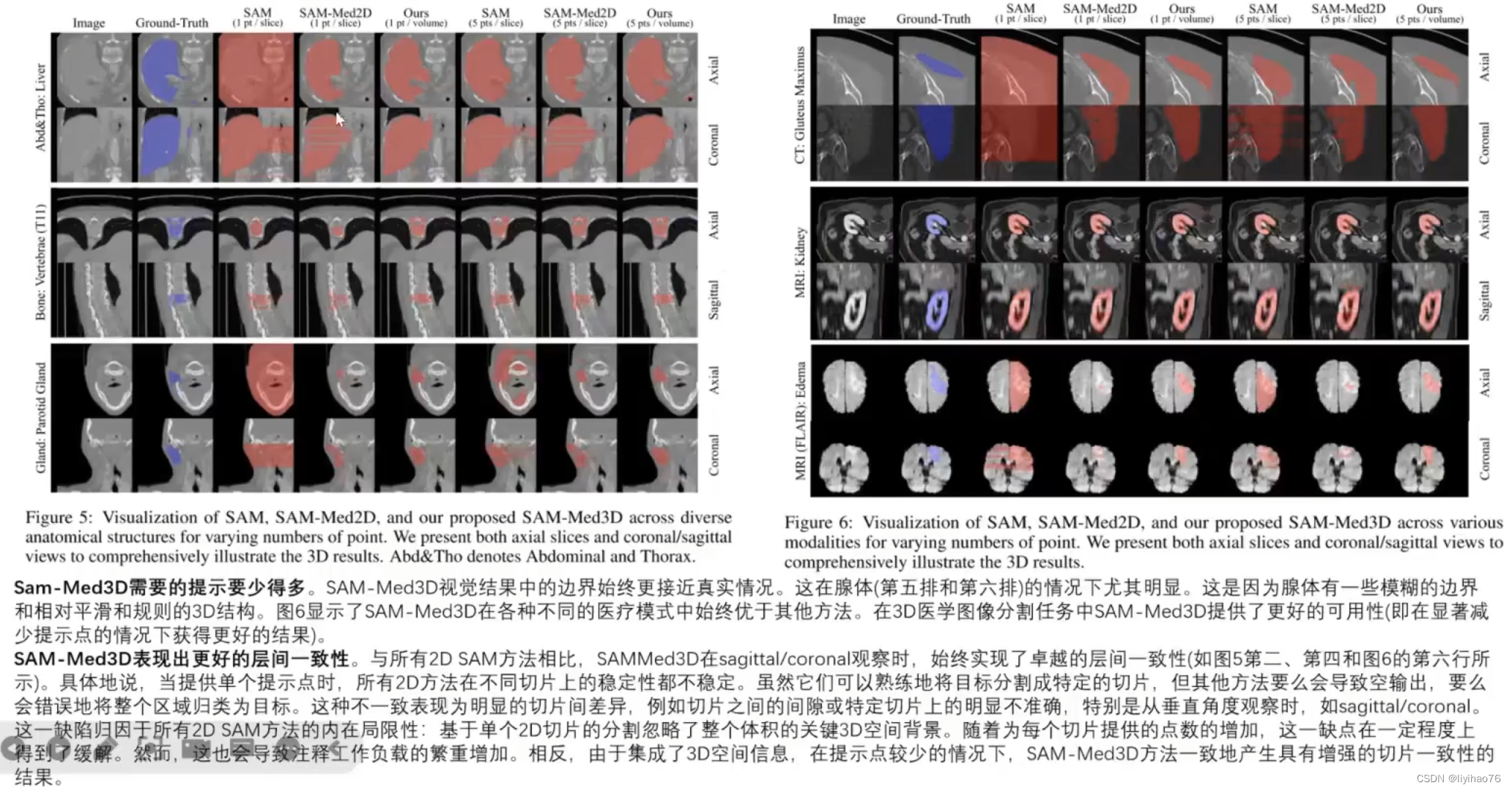
6. 结论
- 在这项研究中,作者提出了 SAM-Med3D,这是一种专门用于3D体素医学图像分割的三维 SAM 模型。SAM-Med3D 在大规模的三维医学图像数据集上从头训练,其在不同组件中都采用了三维位置编码,直接整合三维空间信息,这使得它在体素医学图像分割任务中表现出卓越的性能。具体而言,SAM-Med3D 在提供仅一个提示点的情况下,相较于 SAM 在每个切片上提供一个提示点来说,性能提高了32.90%。这表明它能够在更少的提示点的情况下,在体素医学图像分割任务中取得更好的结果,这证明了它出色的可用性。
- 此外,作者还从多个角度广泛评估了 SAM-Med3D 的能力。对于不同的解剖结构,如骨骼、心脏和肌肉,在提供有限提示点的情况下,SAM-Med3D 明显优于其他方法。在不同的图像模态下,特别是核磁共振图像,通常需要比CT图像更多的提示点才能达到相同的性能,但 SAM-Med3D 在各种模态(包括核磁共振图像)、器官和病变下始终表现出色。此外,SAM-Med3D 的可迁移性也在不同的基准任务上经过了验证,该模型表现出了很强的潜力,因此 SAM-Med3D 有望成为一种强大的三维医学图像 Transformer 的预训练模型。
- 需要强调的是,不仅仅在数值结果方面,在可视化的结果中,SAM-Med3D 模型也表现出了更好的切片间的一致性和可用性。然而,三维模型在体积图像中的提示点变得更加稀疏,这增加了训练的难度。因此,如何更好地训练三维SAM仍然是需要进一步探索的领域,但这项研究为这一领域的未来发展提供了有力的方向和工具。



)
——编译期的处理)







)




![[蓝桥杯 2023 省 A] 颜色平衡树:从零开始理解树上莫队 一颗颜色平衡树引发的惨案](http://pic.xiahunao.cn/[蓝桥杯 2023 省 A] 颜色平衡树:从零开始理解树上莫队 一颗颜色平衡树引发的惨案)

