2.prodigal教程
2.1 介绍
Prodigal 由橡树岭国家实验室和田纳西大学诺克斯维尔分校于2007年在能源部联合基因组研究所的主持下联合开发,是一种用于细菌和古细菌基因组的蛋白质编码基因预测软件工具,Prodigal 已成为世界上最受欢迎的微生物基因预测算法之一。首字母缩略词代表 PROkaryotic DYnamic Programming Genefinding ALgorithm。Dictionary.com 提供了“Prodigal”一词的几种定义。作者希望援引的是:
**3.**丰富多彩;丰富:大自然的Prodigal资源。
而不是更常见的含义(例如浪费,或“Prodigal回头”)。
2.1.1 Prodigal是做什么的?
- 预测蛋白质编码基因:Prodigal 以 GFF3、Genbank 或 Sequin 表格格式提供快速、准确的蛋白质编码基因预测。
- 处理基因组草图和宏基因组:Prodigal 在完成的基因组、草图基因组和宏基因组上平稳运行。
- 快速运行:Prodigal 在现代 MacBook Pro 上 10 秒内分析大肠杆菌 K-12 基因组。
- 无监督运行:Prodigal 是一种无监督机器学习算法。它不需要提供任何训练数据,而是自动从序列本身学习基因组的属性,包括遗传密码、RBS 基序使用、起始密码子使用和编码统计。
- 处理间隙、支架和部分基因:用户可以指定 Prodigal 应如何处理间隙,并有许多选项来允许或禁止基因进入或跨越间隙。
- 识别翻译起始位点:Prodigal 预测大多数基因的正确翻译起始位点,并可以输出有关基因组中每个潜在起始位点的信息,包括置信度评分、RBS 基序等。
- 输出每个基因组的详细汇总统计数据:Prodigal 为每个基因组提供许多统计数据,包括重叠群长度、基因长度、GC 含量、GC 偏度、使用的 RBS 基序以及开始和停止密码子的使用。
2.1.2 Prodigal不能做什么?
- 预测 RNA 基因:目前,Prodigal 不预测 RNA 基因,尽管我们不排除在未来版本中添加此功能。
- 处理带有内含子的基因:带有内含子的基因非常罕见,以至于 Prodigal 不会费心去寻找它们。
- 功能注释基因:Prodigal 不为其预测的基因提供功能注释。
- 处理frameshift:Prodigal 不包含任何处理插入或删除的逻辑。这些类型的测序错误将对Prodigal的基因预测产生有害影响。
- 病毒基因预测:作者尚未对Prodigal进行病毒测试,尽管匿名模式可能在这种情况下有效;然而,Prodigal不包含处理病毒基因组的特殊规则或例程。
2.1.3 cheatsheet
长和短参数选项都支持
Prodigal 这四个命令都是等效的:
$ prodigal -i sequence.fna -o genes.gff -f gff
$ prodigal -isequence.fna -ogenes.gff -fgff
$ prodigal --input_file sequence.fna --output_file genes.gff --output_format gff
$ prodigal --input_file=sequence.fna --output_file=genes.gff --output_format=gff
Prodigal 2.x: Older versions of Prodigal only support short options, and there must be a space between each option and its parameter.
帮助输出
Usage: prodigal [-a protein_file] [-c] [-d mrna_file][-e gap_mode] [-f out_format] [-g trans_table][-h] [-i input_file] [-n] [-o output_file][-p mode] [-q] [-s start_file] [-t train_file][-v] [-w summ_file]Gene Modeling Parameters-p, --mode: Specify mode (normal, train, or anon).normal: Single genome, any number ofsequences. (Default)train: Do only training. Inputshould be multiple FASTA ofone or more closelyrelated genomes.anon: Anonymous sequences, analyzeusing preset training files,ideal for metagenomic dataor single short sequences.meta: (Deprecated) Same as anon.single: (Deprecated) Same as normal.-g, --trans_table: Specify a translation table to use.auto: Tries 11 then 4 (Default)11: Standard Bacteria/Archaea4: Mycoplasma/Spiroplasma#: Other genetic codes 1-25-e, --gap_mode: Specify gap-handling behavior.0: Partial genes run into gaps.(Default)1: Genes cannot run into gaps.2: Do not treat N's as gaps.-c, --closed: Closed ends. Do not allow partial genesat edges of sequence.-n, --force_nonsd: Do not use the Shine-Dalgarno RBS finderand instead force Prodigal to scan formotifs.-t, --training_file: Specify the training file location. Intrain mode, writes the training file.In normal mode, reads the training file.-m: (Deprecated) Similar to '-e 1'.Input/Output Parameters-i, --input_file: Specify input file (default stdin).-o, --output_file: Specify output file (default stdout).-a, --protein_file: Specify protein translations file.-d, --mrna_file: Specify nucleotide sequences file.-s, --start_file: Specify complete starts file.-w, --summ_file: Specify summary statistics file.-f, --output_format: Specify output format.gbk: Genbank-like format (Default)gff: GFF formatsqn: Sequin feature table formatsco: Simple coordinate output-q, --quiet: Run quietly (suppress logging output).Other Parameters-h, --help: Print help menu and exit.-v, --version: Print version number and exit
2.2 *nux系统下的安装
现在一般的电脑都是win10,自带WSL,也是linux啦,mac也是类似的安装步骤。
git clone https://github.com/hyattpd/Prodigal.git
cd Prodigal
make #如果只用一两次,到这里就可以以绝对路径使用啦
# 如果需要经常使用
sudo make install
2.3 基因预测模式
Prodigal 支持三种基因预测模式。
- 普通模式,在这种模式下,Prodigal采用您提供的序列,对其进行研究,了解其特性,然后根据这些特性预测基因。
- 匿名模式,在这种模式下,Prodigal 将预先计算的训练文件应用于提供的输入序列,并根据最佳结果预测基因。
- 训练模式,其工作方式与普通模式类似,但 Prodigal 会保存训练文件以备将来使用。
哪种方法适合您的序列取决于您正在分析的数据集类型。只要你有足够的数据供Prodigal训练,就应该使用正常模式(100kb+ 用于好的 3’ 预测,500kb+ 用于好的 5’ 预测,是安全的数字,尽管你可以用更少的数据来度过难关)。匿名模式应用于宏基因组数据集,或用于太短而无法提供良好训练数据的序列。
正常模式应用于成品基因组、质量合理的基因组草图和大病毒。匿名模式应用于宏基因组、低质量基因组草图、小病毒和小质粒。训练模式是第三个选项,其工作方式与普通模式类似,但会输出一个训练文件,可用于以后的分析。这主要适用于您希望在与要分析的序列不同的序列上进行训练时。
Prodigal 帮助描述中的摘要:
-p, --mode: Specify mode (normal, train, or anon).normal: Single genome, any number ofsequences. (Default)train: Do only training. Inputshould be multiple FASTA ofone or more closelyrelated genomes.anon: Anonymous sequences, analyzeusing preset training files,ideal for metagenomic dataor single short sequences.meta: (Deprecated) Same as anon.single: (Deprecated) Same as normal.
下面将更详细地解释这些模式。
2.3.1 正常模式
要在单个或多个 FASTA 输入序列上以正常模式运行 Prodigal,请执行以下操作:
$ prodigal -i my.genome.fna -o gene.coords.gbk -a protein.translations.faa
-i 选项指定输入文件,可以是单个/多个 FASTA、Genbank 或 EMBL 格式。然而,Genbank 和 EMBL 解析器并不复杂,也没有经过彻底的测试,因此我们建议尽可能使用 FASTA。
-o 选项指定输出文件(基因坐标),-a 选项指定写入蛋白质翻译的位置。蛋白质翻译是可选的,但大多数用户默认需要此信息。
在未指定输入或输出文件的情况下,Prodigal 从 stdin 读取并写入 stdout。因此,以下工作也有效:
$ prodigal < my.genome.fna > gene.coords.gbk
以下选项指定 Prodigal 的输入和输出文件:
Input/Output Parameters-i, --input_file: Specify input file (default stdin).-o, --output_file: Specify output file (default stdout).-a, --protein_file: Specify protein translations file.-d, --mrna_file: Specify nucleotide sequences file.-s, --start_file: Specify complete starts file.-w, --summ_file: Specify summary statistics file.-f, --output_format: Specify output format.gbk: Genbank-like format (Default)gff: GFF formatsqn: Sequin feature table formatsco: Simple coordinate output-q, --quiet: Run quietly (suppress logging output).
了解Prodigal输出中更详细地介绍了各种输出选项和格式。
2.3.2 匿名模式
要在匿名模式下运行 Prodigal,只需在命令行中添加一个“-p anon”选项,即
$ prodigal -i metagenome.fna -o coords.gbk -a proteins.faa -p anon
Prodigal 2.x:在旧版本的 Prodigal 中,此模式称为宏基因组模式,并使用 -p meta 选项调用。Prodigal 3.x 将继续支持此语法,尽管 -p meta 被认为已弃用。我们选择将此模式重命名为匿名模式,因为这更准确地描述了其功能,不仅限于宏基因组序列。
2.3.3 训练模式
Prodigal 还有一个训练模式,可以写一个训练数据文件供以后使用。使用此模式的主要原因是,如果您希望在一个输入序列上进行训练,然后在另一个输入序列上进行分析。例如,要在基因组 1 上训练并在基因组 2 上运行,可以做到:
$ prodigal -i genome1.fna -p train -t genome1.trn
这会将训练数据文件写入 genome1.trn,如 -t 选项所指定。
-t, --training_file: Specify the training file location. Intrain mode, writes the training file.In normal mode, reads the training file.
2.4 按输入类型提供建议
2.4.1 完成的基因组
我们将完成的基因组定义为每个染色体或质粒都在一个重叠群中,并且没有 N(间隙)的基因组。
完成的基因组应在正常模式下运行。
对于确定序列的第一个和最后一个碱基不属于基因的基因组,您应该考虑 -c 选项。
-c, --closed: Closed ends. Do not allow partial genesat edges of sequence.
如果基因组由多条染色体组成,您可以一起或单独分析它们。只有当 (1) 每条染色体至少为 500kb,并且 (2) 您有理由相信染色体在 GC 含量、RBS 基序使用和其他参数方面有很大不同时,才应分离染色体。
质粒更棘手,目前尚不清楚最好的方法是什么。它们可以与染色体一起包含在染色体上(在这种情况下,Prodigal 将一起训练染色体和质粒),或者您可以单独分析它们,如下所述。您的决定应再次以质粒是否与基因组的其余部分相似或不同为指导。
2.4.2 基因组草图
在大多数情况下,应在正常模式下分析基因组草图。即使平均重叠群长度很小(3000+bp),Prodigal也应该做得很好。或者,即使存在一个长重叠群,通常也足以提供良好的训练数据。
如果 Prodigal 在构建一个好的训练集时遇到问题(由于序列在太多重叠群中),它将输出如下所示的警告:
Warning: Training sequence is highly fragmented.
You may get better results with the '-p anon' option.
默认情况下,Prodigal 的参数非常适合支架和/或具有许多重叠群的多个 FASTA。部分基因被允许进入 N 的间隙,这意味着您应该得到相同的结果,分析一个文件中的 1000 个重叠群,或者分析一个支架,其中 1000 个重叠群通过 N 的运行连接在一起。此外,基因被允许从边缘运行。
Prodigal 可以使用 -e 选项以多种方式处理间隙(定义为两个或多个完全模棱两可的字符的连续密码子):
-e, --gap_mode: Specify gap-handling behavior.0: Partial genes run into gaps.(Default)1: Genes cannot run into gaps.2: Do not treat N's as gaps.
在极少数情况下,如果你正在分析一个scaffold,并且你确定你的所有间隙中都有完全正确的N数量(为了保持阅读框架),你可以选择*-e 2*选项,这将允许Prodigal建立跨越间隙的基因模型。如果您的重叠群很大,但您的序列质量较低,并且包含许多不应被视为间隙的短运行 N,您也可以使用此选项。
Prodigal 2.x:旧版本的 Prodigal 不包含间隙处理(-m 选项除外,>它的作用类似于上面的 -e 1,但需要运行 50 N 才能将其视为间隙)。
如果你觉得你的基因组草图在太多的重叠群中,无法获得好的结果(或者如果你看到上面显示的警告),另一种选择是找到一个已经完成的密切相关的基因组,对其进行训练,然后使用该训练文件来分析你高度碎片化的基因组草图。此过程在训练模式一节中进行了介绍。
如果您的基因组处于低质量草图中,并且您没有高质量的密切相关基因组可供训练,则应在匿名模式下分析序列。
提示:在许多重叠群中,您应该小心地将 -c 选项与基因组草图一起使用,因为这将阻止 Prodigal 预测部分基因。同样,如果你有一个支架,里面有很多间隙,你应该小心使用 -e 选项,因为你也可能丢失许多部分基因。
2.4.3 宏基因组
宏基因组最简单的方法是将所有序列放在一个 FASTA 文件中,并在匿名模式下对其进行分析。这将产生合理的结果(大约95%的效果,就像Prodigal在实际基因组上接受过训练一样)。它还具有易于并行化的优点,因为文件中的每个序列都可以独立于文件中的任何其他序列进行处理。
如果可能,更理想的解决方案是从样本中组装尽可能多的基因组,将每个基因组放入 FASTA 文件中,然后使用正常模式分析每个基因组。然后,您可以使用匿名模式分析剩菜。
同样,您可以使用分箱或分类程序对序列进行分箱(这些程序通常依赖于 GC 内容、BLAST 搜索、k-mer 搜索或其他信息)。然后,您可以从每个 bin 制作多个 FASTA 文件,并使用正常模式对其进行分析。
提示:切勿使用正常模式分析包含来自多个基因组序列的多个 FASTA 文件。这条规则的唯一例外是基因组密切相关(同一物种的菌株)。
上述两种解决方案都应该比匿名模式产生更好的结果,因为当 Prodigal 可以在序列本身而不是依赖预设的训练文件时,它总是做得更好。但是,这些方法涉及大量的预处理工作,并且无法轻松并行运行。最快的解决方案是使用匿名模式。
测序错误曾经在宏基因组样本中很常见,但随着测序技术的改进,问题正在逐渐减少。我们选择不花时间在Prodigal中解决这个问题。尽管如此,如果您的读段包含许多插入/删除,您最好使用像 FragGeneScan 这样的程序来分析您的样品,该程序专门用于处理测序错误。
2.4.4 替代遗传密码
Prodigal 支持 NCBI 定义的所有遗传密码。大多数细菌和古细菌使用遗传密码 11,它使用三个终止密码子(TAA、TGA 和 TAG)。有些细菌不使用TGA作为终止密码子。支原体、螺旋体和解脲支原体将 UGA 翻译成色氨酸 (W)(遗传密码 4),而使用遗传密码 25 的细菌将 UGA 翻译成甘氨酸 (G)。
默认情况下,Prodigal 会尝试遗传密码 11。如果平均基因长度太低,它会尝试遗传密码 4。如果平均基因长度仍然太低,它会恢复到遗传密码 11 并输出警告。这如下所示:
Building training set using genetic code 11...done!
Checking average training gene length...459.7, too low.
Trying genetic code 4...still bad, reverting to genetic code 11.
Redoing genome with genetic code 11...done.Warning: Average training gene length is low (459.7).
Double check translation table or check for pseudogenes/gene decay.Examining upstream regions and training starts...done.
Prodigal不能自动区分遗传密码 4 和遗传密码 25。在这种情况下,它可能会选择遗传密码 4,并且您需要使用遗传密码 25 手动重新运行。
自动检测是高度可靠的,不应该犯任何错误(我们在20,000个基因组上进行了测试,它在遗传密码确定中没有犯任何错误)。但是,用户也可以使用 -g 选项显式指定遗传密码。
-g, --trans_table: Specify a translation table to use.auto: Tries 11 then 4 (Default)11: Standard Bacteria/Archaea4: Mycoplasma/Spiroplasma#: Other genetic codes 1-25
对于任何使用遗传密码 25 的生物体来说,这将是必要的,并且可能有一些无法被自动检测识别的 4。如果您知道基因组的遗传密码,则不妨覆盖自动检测并使用此选项显式指定它。
Prodigal 2.x:以前版本的 Prodigal 默认只执行遗传密码 11,不会自动检测 11 和 4。要分析具有遗传密码 4 的基因组,必须使用 -g 4 选项显式指定此翻译表。
2.4.5 具有基因衰变的生物体
如果您在正常模式下运行 Prodigal,并且您会看到以下警告:
Warning: Average training gene length is low (459.7).
Double check translation table or check for pseudogenes/gene decay.
那么最终的基因预测可能有些“错误”。当 Prodigal 训练集中的平均基因长度小于 600bp 时,会给出上述警告。有些生物体的基因可能比平均水平小。如果是这种情况,您可以忽略警告并照常继续
Prodigal 2.x:以前版本的 Prodigal 不会输出有关潜在基因衰变的警告。
然而,该生物体可能已经经历了广泛的基因衰变(例如麻风分枝杆菌),Prodigal 预测的许多基因可能是基因片段、假基因或简单的无稽之谈(假阳性)。在这种情况下,您可能需要过滤掉很多这些预测。
一种可能有帮助的替代方法是使用训练模式对密切相关的基因组进行训练。例如,如果 Prodigal 接受结核分枝杆菌训练,并将该训练文件应用于麻风分枝杆菌,则 Prodigal 预测的基因比直接在麻风分枝杆菌上以正常模式运行时少得多。为了使这个过程起作用,训练基因组必须质量良好(并且本身没有经历过衰变),并且与要分析的基因组不要太远。
2.4.6 质粒、噬菌体、病毒和其他短序列
分离的短序列(<100kbp),如质粒、噬菌体和病毒,通常应使用匿名模式进行分析。对于质粒,最好将质粒与染色体相结合,并在正常模式下运行所有内容。但是,如果您认为质粒与基因组的其余部分完全不同,那么匿名模式是最佳选择。如果序列足够长,则应改用正常模式。
我们从未广泛研究过Prodigal到底需要多少序列才能产生好的结果。我们普遍认为,20kbp-100kbp太短了,无法收集足够的统计数据来很好地预测基因,但我们没有实际数据来支持这一说法。如果您觉得在正常模式下运行 50kbp 序列很舒服,并且您对获得的结果感到满意,那么请务必继续这样做。尤其是起始站点,需要大量数据(100kbp 可能只有训练集中的 ~80 个左右的起始站点,因为 Prodigal 只在较长的 ORF 开始时进行训练)。我们建议 500kbp+ 以获得理想的 5’ 预测,尽管这可能是保守的。如果您的序列处于这个模糊的范围内(3’ 预测为 20kbp 至 100kbp,5’ 预测为 100kbp 至 500kbp),您可以尝试运行正常和匿名模式,并手动检查结果是否存在任何差异。
Prodigal 不包含处理病毒的特殊例程。因此,它无法处理病毒中有时发生的某些现象,例如frameshift。病毒通常应按上述方式进行分析,短基因组以匿名模式分析,较长的基因组以正常模式分析。
2.5 了解Prodigal输出
默认情况下,Prodigal 会生成一个输出文件,该文件由基因坐标和与每个基因关联的一些元数据组成。但是,该程序可以根据用户的要求生成另外四个输出文件:蛋白质翻译(使用 -a 选项)、核苷酸序列(使用 -d 选项)、所有开始/终止对的完整列表以及分数信息(使用 -s 选项)以及有关基因组或宏基因组的统计信息摘要(使用 -w 选项)。下面,我们将解释每种类型的文件的输出。
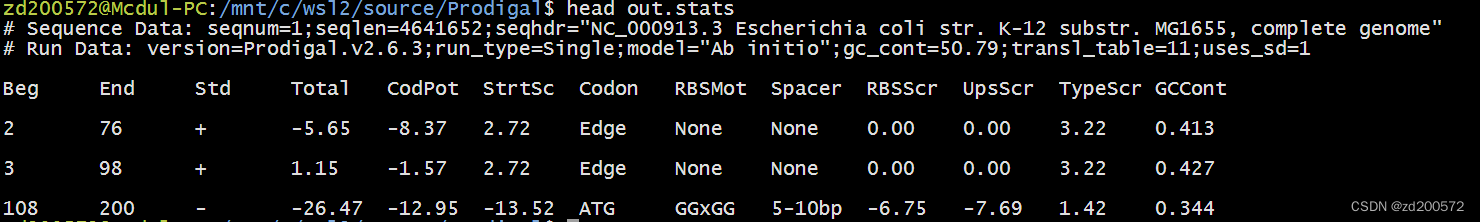
2.5.1 基因坐标
基因坐标文件列出了每个基因的位置以及一些额外的评分信息。默认情况下,Prodigal 会生成一个类似 Genbank 的特征表;但是,用户可以通过 -f 选项指定一些其他输出类型:
-f, --output_format: Specify output format.gbk: Genbank-like format (Default)gff: GFF formatsqn: Sequin feature table formatsco: Simple coordinate output
gff 参数生成GFF3 输出。sqn 参数生成SFT,但用户仍必须填写信息才能获得“可供提交”的文件。sco 参数生成SCO输出,适用于用户只需要基因坐标而不需要其他任何信息的情况。
对于 FASTA 输入文件中的每个单独序列,Prodigal 都会生成一个标头,其中包含一个以分号分隔的字符串,其中包含有关该序列及其分析方式的信息(以名称 = 值对的形式)。在Genbank格式中,它被放置在“DEFINITION”行上。在 GFF3 中,此信息分为两行注释。
Genbank 标头示例:
DEFINITION seqnum=1;seqlen=4639675;seqhdr="NC_000913 # Escherichia coli str. K-12 substr. MG1655, completegenome.";version=Prodigal.v2.60;run_type=Single;model="Ab initio";gc_cont=50.79;transl_table=11;uses_sd=1
GFF3 接头示例:
##gff-version 3
# Sequence Data: seqnum=1;seqlen=4639675;seqhdr="NC_000913 # Escherichia coli str. K-12 substr. MG1655, com
plete genome."
# Model Data: version=Prodigal.v3.0.0-devel.1.0;run_type=Normal;model="Ab initio";gc_cont=50.79;transl_tabl
e=11;uses_sd=1
此标头中的字段如下所示:
- seqnum:此序列的序号 ID,从 1 开始。
- seqlen:序列中的碱基数。
- seqhdr:整个 FASTA 标头行。
- version:用于分析此序列的 Prodigal 版本。
- run_type:“Ab initio”表示正常模式,“Anonymous”表示匿名模式。
- model*(仅限匿名模式):*用于分析序列的预设训练文件的信息。
- gc_cont:序列的 % GC 含量。
- transl_table:用于分析序列的遗传密码。
- uses_sd:如果 Prodigal 使用其默认的 RBS 取景器,则设置为 1,如果扫描其他图案,则设置为 0。
除了链(由 GFF 中的“+/-”标签指定,或由 Genbank 格式中表示反向链的“互补”关键字指定)和基因边界之外,Prodigal 还生成一个以分号分隔的名称-值对字符串(以 name=value 的形式),其中包含有关每个基因的评分和统计信息。
在Genbank格式中,它被放置在“注释”行上,如下所示:
CDS 2655..2882/note="ID=1_3;partial=00;start_type=ATG;stop_type=TAA;rbs_motif=AGxAGG/AGGxGG;rbs_spac
er=5-10bp;gc_cont=0.241;conf=100.00;score=44.71;cscore=32.81;sscore=11.90;rscore=9.40;uscore=-1.05;tscore=3
.55;"
在 GFF3 格式中,字符串被放置在最后一个字段中,因此基因只占据一行而不是两行,如下所示:
NC_000913 Prodigal_v3.0.0-devel.1.0 CDS 337 2799 338.7 + 0 ID=1_2;part
ial=00;start_type=ATG;stop_type=TGA;rbs_motif=GGAG/GAGG;rbs_spacer=5-10bp;gc_cont=0.531;conf=99.99;score=33
8.70;cscore=322.16;sscore=16.54;rscore=11.24;uscore=1.35;tscore=3.95;
GFF3 格式要求在第一个字段中使用“id”。Prodigal 从 FASTA 标头中提取第一个单词,并将其用作其 ID。此 ID 不保证是唯一的(文件中各种标头的第一个单词可能相同),因此我们建议用户改用分号分隔的字符串中的“ID”字段。
以分号分隔的字符串中的字段如下所示:
- ID:每个基因的唯一标识符,由序列的序号 ID 和序列中该基因的序号 ID 组成(用下划线分隔)。例如,“4_1023”表示文件中第 4 个序列中的第 1023 个基因。
- partial:基因是否从序列边缘或间隙中运行的指标。“0”表示基因具有真正的边界(开始或停止),而“1”表示基因在该边缘“未完成”(即部分基因)。例如,“01”表示基因在右边界处是部分的,“11”表示两个边缘都是不完整的,“00”表示具有起始密码子和终止密码子的完整基因。
- start_type:起始密码子的序列(通常为 ATG、GTG 或 TTG)。如果基因没有起始密码子,则此字段将被标记为“Edge”。
- stop_type:终止密码子的序列(通常为 TAA、TGA 或 TAG)。如果基因没有终止密码子,则此字段将被标记为“Edge”。
- rbs_motif:浪子发现的RBS图案(如“AGGA”或“GGA”等)
- rbs_spacer:起始密码子和观察到的基序之间的碱基数。
- gc_cont:基因序列的GC含量。
- gc_skew:基因序列的GC偏斜。
- conf:该基因的置信度分数,代表该基因是真实的概率,即 78.3% 表示浪子认为该基因在 78.3% 的时间内是真实的,在 21.7% 的时间内是假阳性。
- score:该基因的总分。
- cscore:分数的六聚体编码部分,即该基因看起来像真蛋白质的程度。
- sscore:该基因翻译起始位点的分数;它是以下三个字段的总和。
- rscore:该基因的 RBS 基序的分数。
- uscore:围绕起始密码子的序列的分数。
- tscore:起始密码子类型的分数(ATG vs. GTG vs. TTG vs. Nonstandard)。
- mscore:剩余信号的分数(终止密码子类型和前导/滞后链信息)。
Prodigal 2.x:旧版本的 Prodigal 没有 stop_type、gc_skew 或 mscore 字段。此外,uscore 字段仅引用起始密码子上游序列的分数。
2.5.2 蛋白质翻译
蛋白质翻译文件由来自多个 FASTA 格式的所有序列中的所有蛋白质组成。FASTA 标头以文本 ID 开头,该文本 ID 由原始 FASTA 序列标头的第一个单词组成,后跟下划线,后跟蛋白质的序数 ID。此文本 ID 不保证是唯一的(这取决于用户提供的 FASTA 标头),这就是为什么我们建议在最后一个以分号分隔的字符串中使用“ID”字段。
大肠杆菌基因组中第 4 个蛋白质的 id NC_000913 的示例标头:
>NC_000913_4 # 3734 # 5020 # 1 # ID=1_4;partial=00;start_type=ATG;rbs_motif=GGA/GAG/AGG;rbs_spacer=5-10bp;g
c_cont=0.528
标题中接下来的三个字段(用“#”号分隔)是基因组中最左边的坐标、最右边的坐标和链(1 表示正向链基因,-1 表示反向链基因)。坐标信息后面是一个以分号分隔的字符串,与基因坐标文件中描述的字符串相同(有关字段定义,请参阅此处的列表),仅使用以下字段:ID、partial、start_type、stop_type、rbs_motif、rbs_spacer、gc_cont 和 gc_skew 以及 conf。除 conf 字段外,标头不包含有关该基因的任何评分信息。
2.5.3 核苷酸序列
核苷酸序列文件按照蛋白质翻译部分所述的相同规则和约定生成多个 FASTA 输出。唯一值得注意的一点是,Prodigal 使用 DNA 字母表来生成这些序列,而不是 mRNA(因此您会在输出中看到“T”而不是“U”)。
其他文件还有启动文件和汇总统计。
2.6 软件实战
./prodigal -v
Prodigal V2.6.3: February, 2016
./prodigal -i GCF_000005845.2_ASM584v2_genomic.fna -o my.genes.gff -a my.proteins.faa -f gff -s out.stats
注:这是基于软件2.6.3版本的实践




函数详细介绍)








获取某一接口所有实现类,应用于策略模式)






)
