在RNA-seq数据分析中,差异表达基因分析是一项基本的技能,其中热图又是一种特别常见的用来展示差异表达基因分析结果的方式,今天分享一个非常好用的绘制热图的R函数:heatmap.2。该函数来自gplots包。
安装
install.packages("gplots")library(gplots)使用
1. 读取数据
# 毫无疑问,绘制热图需要一个数值矩阵作为输入# 我准备好了一个基因表达数据集,存放在名为data的文件中,该数据集含有11个样本,50个基因# 每列是一个样本,每行是一个基因# commanddata sep = "\t", header = T,row.names = 1, stringsAsFactors = F))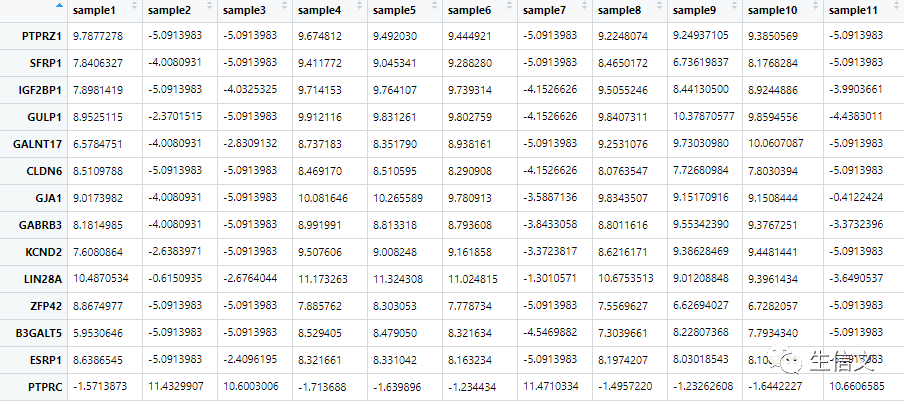
数据集data的一部分
2. 最基本使用方法
# 有了数值矩阵就可以绘制热图了,# commandheatmap.2(data,trace="none")# 参数: # 1. data: 刚才读入的数值矩阵 # 2. trace: 稍后再讲# 结果说明: # 1. 上述命令可以说是使用heatmap.2最简单的方式 # 2. 在这种情况下,除 trace这个参数之外,其余的参数均使用默认值 # 3. 得到的结果如下所示 # 4. 在默认情况下,生成的热图有如下要点: # a. 函数默认计算各行和各列数据之间的相关性,并根据它们的相似程度对行和列进行聚类,并且对行和列都绘制了系统发生树 # b. 左上角的标度中有一条线,这条线表示处于各个颜色区段内的值的个数 # c. 函数默认以输入矩阵的行名和列名为热图中行和列的lable,在这个例子中,行的lable数目不等于矩阵中的行数,是因为图比较小,不足以显示所有的lable,R自动选择显示了一部分 # d. 热图的配色使用默认配色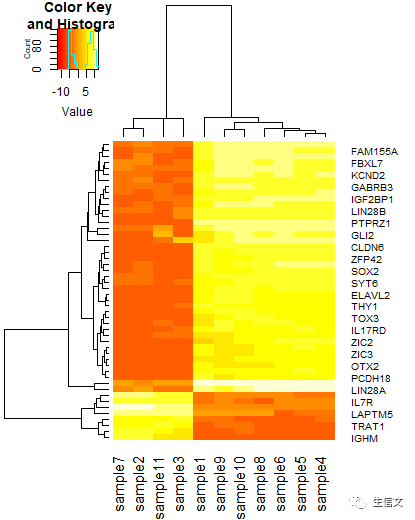
3. 更多参数
# commandheatmap.2(data,trace="none", Rowv = F) # commandheatmap.2(data,trace="none", Colv = F)# 新参数: # 1. Rowv: 该参数接受一个逻辑值 TRUE(T)或FALSE(F),表示是否根据各行数据之间的相似程度对它们进行聚类 ,默认值为TRUE # 2. Colv:与Rowv类似,表示是否根据各列数据之间相似程度对它们进行聚类,默认值为TRUE # 结果说明: # 1. 新结果如下方所示 # 2. 当Rowv被设置为FALSE时,可以看到函数不再根据各行数据之间的相似性对其聚类,自然也就不会绘制系统发生树 # 3. 当Colv被设置为FALSE时,不再对列进行聚类,也没有系统发生树的绘制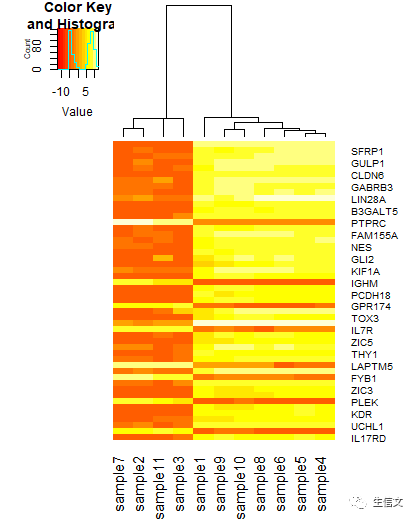
将RowV设置为FALSE
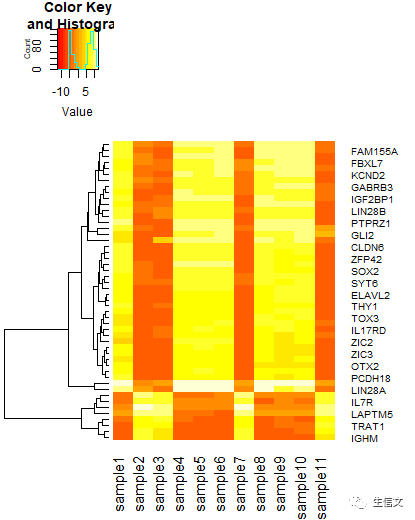
将ColV设置为FALSE
# commandheatmap.2(data,trace="none", dendrogram = "row")# 新参数 # 1. dendrogram:该参数接受以下三个值中的一个: # a. "row":表示只对行绘制系统发生树 # b. "column":表示只对列绘制系统发生树 # c. "both:表示对行和列都绘制系统发生树,这也是该参数的默认值 # 注意: # 1. 该参数只是指定是否系统发生树,不会干扰对行和列进行聚类 # 2. 在参数Rowv和Colv为TRUE的情况下,函数仍然会进行行和列的聚类 # 结果说明: # 1. 新结果如下方所示 # 2. 可以看到该参数并没有影响行和列的聚类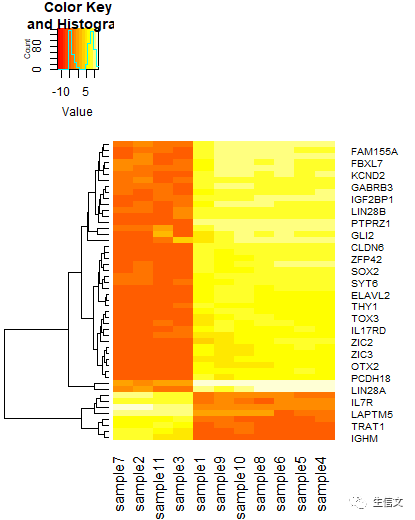
dendrogram = "row"
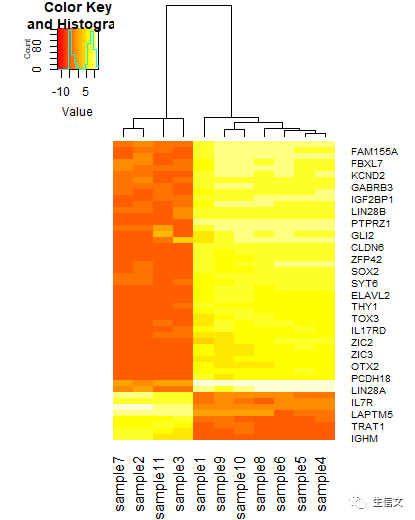
dendrogram = "column"
# commandheatmap.2(data,trace="none", dendrogram = "column", scale = "row")# 新参数 # 1. scale:该参数接受以下三个值中的一个: # a. "row":表示将每行数据进行转化,使每行数据的平均数为0,其实就将每行数据转为其 Z score # b. "column": 表示将每列数据转换为对应的Z score # c. "none":行和列的数据都不做转换,该值是该参数的默认值 # 结果说明: # 1. 新结果如下所示 # 2. 与上边的图相比,对数据进行转换使得不同类别间的差别更明显,图片也更好看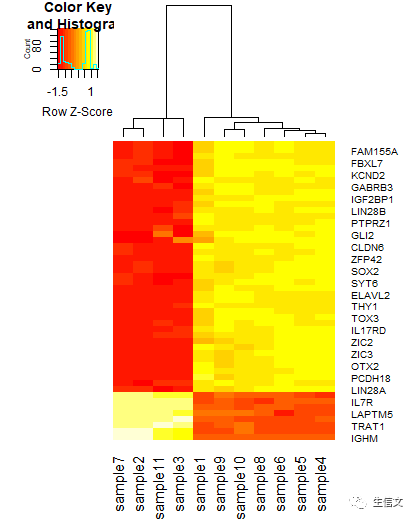
# commandheatmap.2(data,trace="none", dendrogram = "column", scale = "row", colsep = 4, rowsep = 42, sepcolor = "blue") # 新参数 # 1. colsep:指定一个正整数n,然后函数将会在热图中的第n列与第n+1列中间留出一段间隔 # 2. rowsep:与colsep功能类似,指定一个正整数n,然后函数将会在热图中的第n行与第n+1行中间留出一段间隔 # 3. sepcolor:间隔的颜色,如果不指定该参数,则默认值为白色# 结果说明: # 1. 新结果如下方所示 # 2. 可以看到,与上图相比,热图中多出了两个蓝色的间隔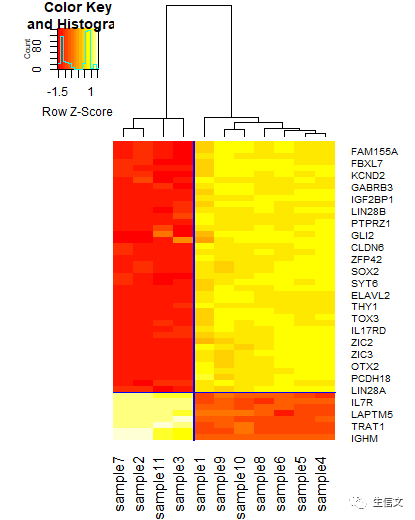
# commandheatmap.2(data,trace="none", dendrogram = "column", scale = "row", colsep = 4, rowsep = 42, cellnote = data, notecex = 1.2, notecol = "red") # 新参数: # 1. cellnote:指定一个矩阵,将该矩阵中的值添加到热图上的每个色块中,该矩阵应该与绘制热图的矩阵有相同的行数和列数 # 2. notecex:与cellnote对应,表示将填充到色块上的值放大到默认大小的多少倍,大于1表示放大,小于1表示缩小 # 3. notecol:填充到色块中的值的字体颜色 # 说明: # 1. 将值添加到色块上适合色块比较少的热图,该例子中色块较多,添加了值之后一片模糊,因此在这里就不展示了# commandheatmap.2(data, dendrogram = "column", scale = "row", colsep = 4, rowsep = 42, trace = "column") # 参数: # 1. trace:该参数接受如下4个值中的一个 # a. "row":表示对每行数据绘制一条曲线,曲线高低起伏表示色块对应的值的大小 # b. "column":表示对每列数据绘制一条曲线,曲线高低起伏表示色块对应的值的大小,该值为该参数的默认值 # c. "both":表达对行和列都绘制曲线 # d. "none":表示对行和列都不绘制曲线 # 结果说明: # 1. 新结果如下方所示 # 2. 一般热图不需要绘制该曲线,该曲线让热图看起来杂乱 # 3. 因为该参数的默认值为 column,因此在之前的例子中,该参数被专门设置为none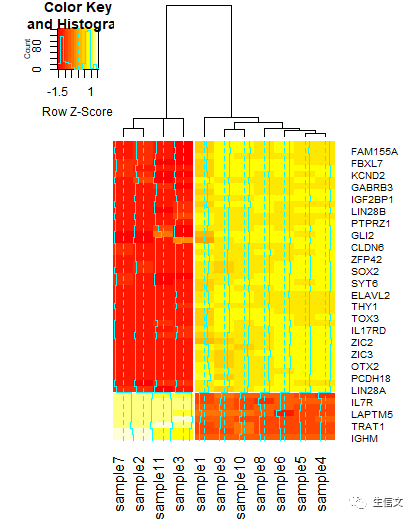
# commandheatmap.2(data,trace = "none", dendrogram = "column", scale = "row", colsep = 4, rowsep = 42, cexRow = 1.5, cexCol = 1.5) # 新参数: # 1. cexRow:表示将行的lable变为默认值的多少倍,大于1表示放大,小于1表示缩小 # 2. cexCol:与cexRow类似# 结果说明: # 1.可以看到行和列的lable都比之前大了一些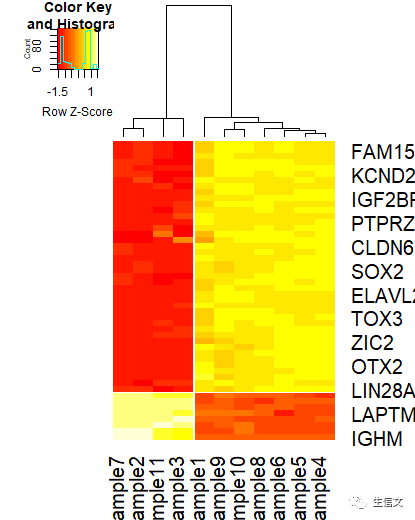
# commandheatmap.2(data,trace = "none", dendrogram = "column", scale = "row", colsep = 4, rowsep = 42, labRow = 1:50, labCol = 1:11, colRow = c("red","red"), colCol = c("blue","blue")) # 新参数 # 1. labRow:指定一个向量作为行的lable,指定为NA则不显示lable # 2. labCol:指定一个向量作为列的lable,指定为NA则不显示lable # 3. colRow:指定行lable的颜色 # 4. colCol:指定列lable的颜色 # 注意: # 1. colRow 和 colCol的值不会循环补齐,也就说,指定几个颜色,就给几个lable着色,其它lable仍然使用默认的黑色 # 结果说明: # 1. 新结果如下方所示 # 2. 可以看到,我们分别只指定了两个行和列lable的颜色,因此也就分别只有两个lable被着色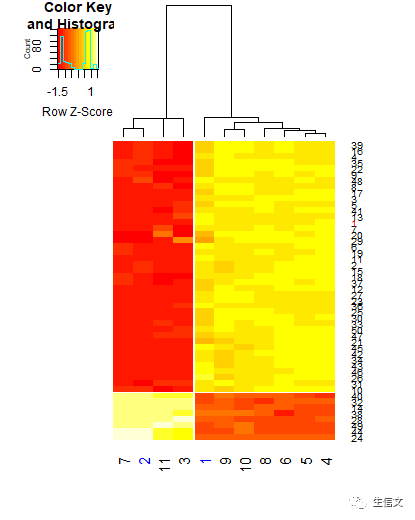
# commandheatmap.2(data,trace = "none", dendrogram = "column", scale = "row", colsep = 4, rowsep = 42, key = TRUE, density.info = "none", key.title = "GENE", key.xlab = "Z Score", key.ylab = NA)# 新参数: # 1. key:接受一个逻辑值TRUE或FALSE,表示是否绘制左上角的颜色标度条,默认为TRUE # 2. density.info:接受一下3个值中的一个: # a. "none":表示在颜色标度条中不绘制密度曲线或直方图 # b. "histogram":表示在颜色标度中绘制直方图 # c. "density",表示在颜色标度中绘制密度曲线 # 3. key.title:标度条的名称,绘制在标度条的上方 # 4. key.xlab:标度条的 x 轴的名称,绘制在标度条的下方,设置为NA则不显示 # 5. key.ylab:标度条的 y 轴的名称,绘制在标度条的左侧,设置为NA则不显示 # 结果说明: # 1. density.info 被设为"none",因此没有了标度条中的线条 # 2. 标度条的 x,y轴名称也发生了相应的变化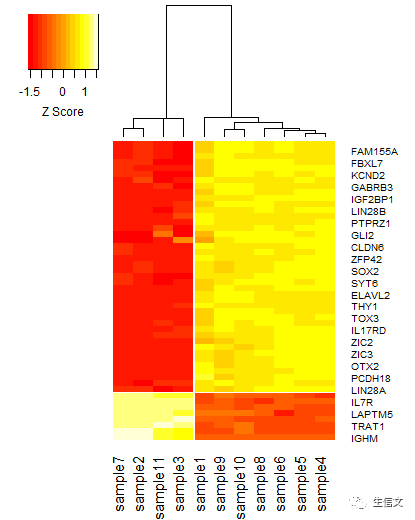
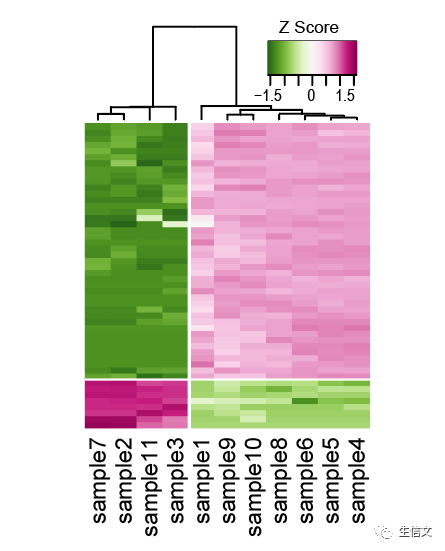
# commandlibrary(RColorBrewer)mypalette "PiYG")morecols heatmap.2(data, dendrogram = "column", scale="row", col=rev(morecols(150)), trace="none", colsep = 4, rowsep = 42, labRow = NA, density.info = "none", key.title = NA, key.xlab = "Z Score", key.ylab = NA) # 新参数 # 1. col:指定用来绘制热图的颜色# 说明: # 1. 这里产生用来绘制热图的渐变色用到了一个RColorBrewer包和colorRampPalette函数,这里不细讲,感兴趣的可以自己学习 # 2. 新结果如下方所示 # 3. 最后用AI稍微修一修,比如调整一下标度条的位置,就可以了