UNet 系列
- UNet
- 下采样和上采样
- 跳跃连接
- UNet++:多层级和多尺度的密集链接
- nnUNet
- 集成模型
- 预处理
- 训练过程
- 推理
- 后处理
- 4行命令使用 nnUNet 训练自己的医学图像分割模型
UNet
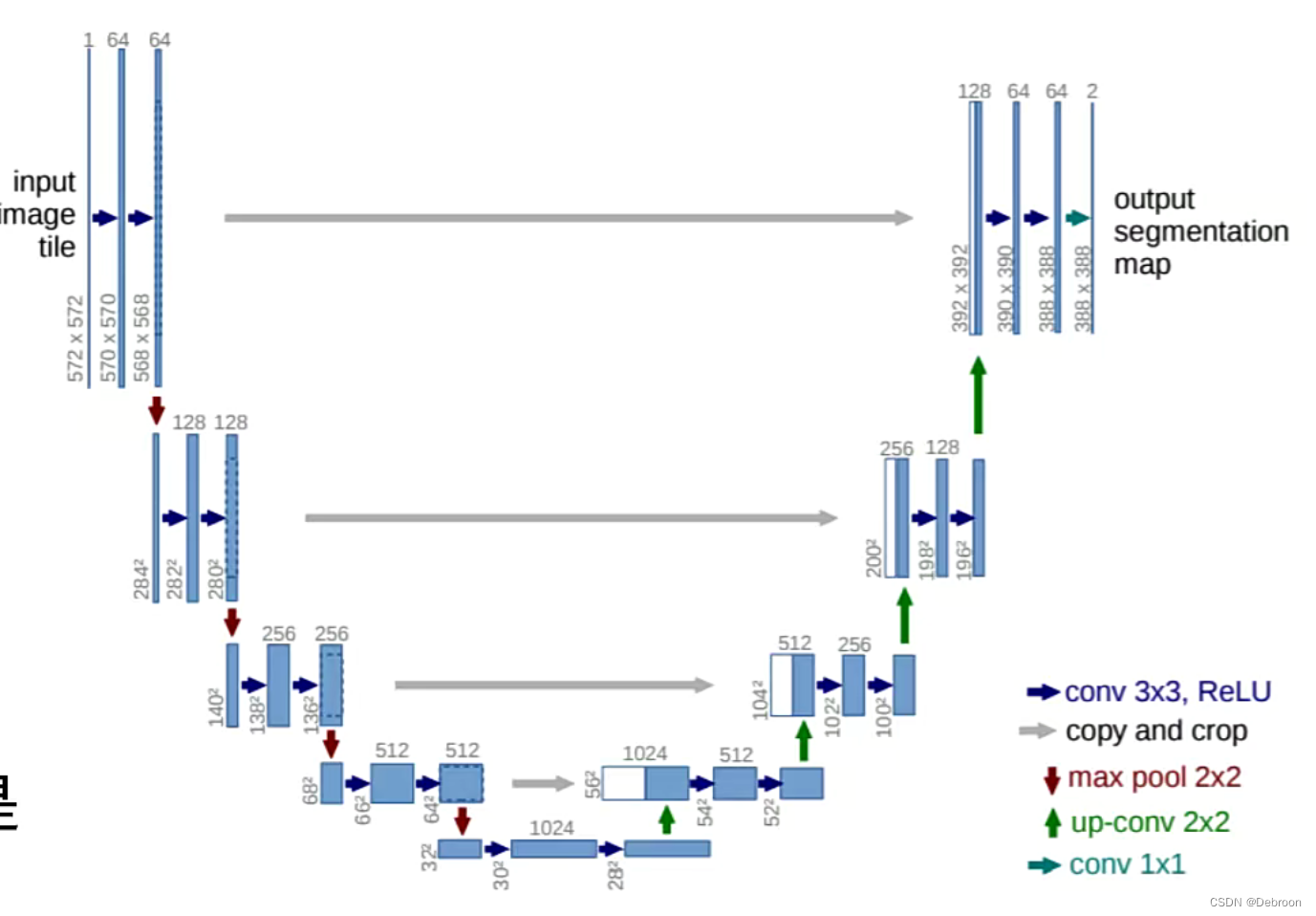
经典的卷积神经网络都很深,越深的卷积层越适合处理大目标的东西,而医学病症区域往往极小,UNet结构简单适合处理小目标。
医学图像边界模糊,梯度复杂,病症区域可能极其微小,需要较多的高分辨信息。
同时人体内部结构相对固定,分割目标在人体图像中的分布很有规律,语义简单明确,低分辨率信息就可以提供。
UNet使用低分辨率信息做粗调找到病症大概位置,再使用高分辨率精确分割。
假设我们有一张来自太空望远镜的高分辨率图片需要让人来识别上面的行星。
高分辨率图片可以提供丰富的细节,如行星的形状、颜色甚至是表面的模样。所以它有助于我们准确地识别和区分行星。
但如果图片太大,你可能要花很久的时间去一点一点地找。
在这个时候,如果我们还有一张低分辨率的图片,虽然它的细节不够,但可以快速看到图上大概有哪些行星,那么小学生就可以用低分辨率的图片找到大概的位置,然后用高分辨率的图片去精确地找到行星和识别行星。
在UNet中,高分辨率信息可以提供丰富的细节,有助于精确定位、识别、分割,而低分辨率信息可以扩大视野,帮助网络找到有用信息的粗略位置。
下采样和上采样
下采样(卷积步长为2)和上采样()其实很好理解,就像是在看一本有非常多内容,但是文字非常小的书,所以我们需要用放大镜来帮助我们看清楚每一个字,这个过程就像是上采样。
反过来,如果我们需要快速的浏览这本书,而不需要关注每一个具体的字,那么我们就可能会缩小书页,让每一页的内容都能够一目了然,这就像是下采样。
在UNet这类神经网络中,上采样和下采样也有类似的作用。
下采样可以帮助网络抽象和理解图片的总体结构和规律,就像是快速浏览书籍,而上采样则可以帮助网络恢复图片的细节,就像是放大字来看清楚每个字。
跳跃连接
跳跃连接的作用?
跳跃连接的作用就像是在搭积木的时候,直接从一堆积木中找到你需要的积木,而不用一块块的从低到高去查找。
这样就可以更快的找到你需要的积木,并能很好的保存积木的原始信息,而不是在查找的过程中可能会改变积木的形状。
再你做家庭作业的时候,假设你正在解决一个很难的数学问题,可能需要很多步骤。
当你在后面的步骤中遇到困难的时候,往往需要回头去查看前面的步骤,这时候如果你能直接回到具体的步骤,看到原始的解题思路,那你会更快的找出问题,并完成作业。
这里,“回到具体的步骤,看到原始的解题思路”就像是跳跃连接,直接找到你需要补充的信息,而不是从头开始再回顾一遍。
这在神经网络中也很有用,特别是在一个深层神经网络中。
因为在网络的深层中,网络可能已经失去了原始的信息,这时候如果有跳跃连接直接跳回到浅层获取原始的信息,网络就能更好的综合考虑全局和局部的信息,做出更准确的预测。
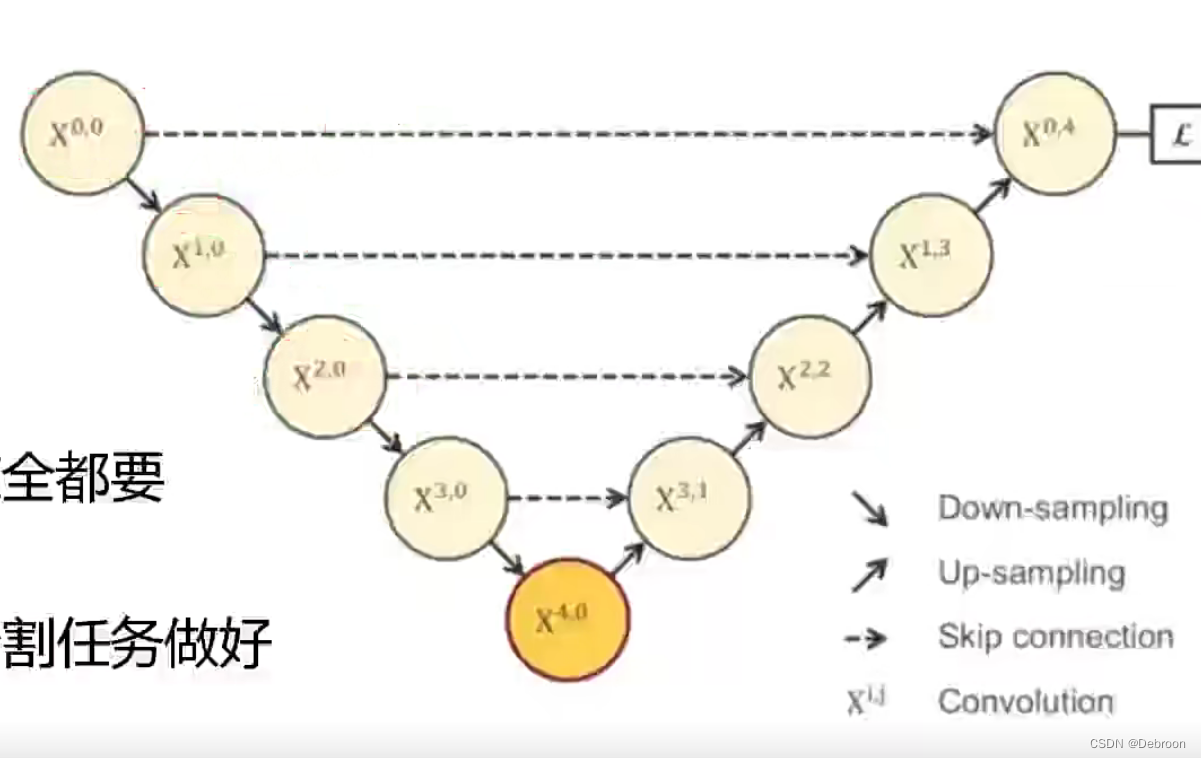
UNet++:多层级和多尺度的密集链接
UNet:
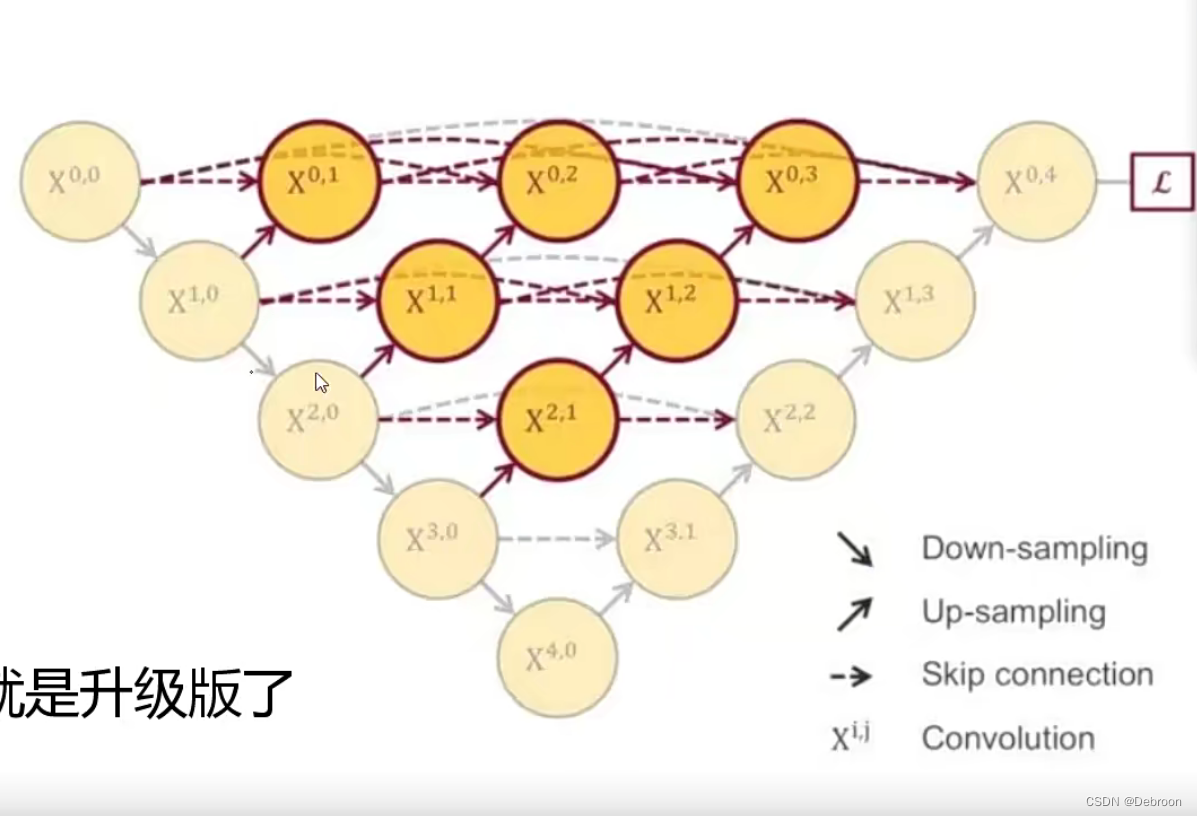
UNet++:
UNet和UNet++ 类似 飞机和飞机升级版。
都是用来飞行,能带我们去不同的地方。但升级版飞机注重在对原来飞机的改良上,帮助它更好地完成任务。
UNet就像第一代飞机,它的构造很简单,包括下降路径(encoder)和上升路径(decoder),它们尽可能地提取图像信息和预测图像类别。
但是这第一代飞机有局限性。比如说,它只能够做一种类型的飞行任务,不能多任务并行,而且飞行路径不能及时地进行优化调整。
然后,人们发明了UNet++,就像升级版的飞机。它不仅有下降路径和上升路径,还增加了跳跃链接和深度监控。跳跃链接就像是告诉飞行员的不同时期的飞行状况,以便更好地调整飞机路径;深度监控则是增加了飞行途中的多个监控点,可以及时地根据飞行状况进行路径调整。
也就是说,UNet++ 创新性地将解码器(decoder)的不同阶段和编码器(encoder)的各个阶段相互连起来,改进了信息流动的方式,让飞机飞行更加灵活,可控且高效。
再比如,原来每个人只能收到前面一个人的信息,那最前面和最后面距离很远,类似年轻人和老年人之间有代沟。
这样的结构可以有效地缓解两个主要问题:一是语义分割中的大对象和小对象的偏好问题;二是深度监督所带来的深层网络训练难的问题。
nnUNet
集成模型
通常的分割模型都是针对某一特定任务(如心脏分割)进行研究的,需要特定的网络架构设计以及训练方法的设定,它只能解决特定问题而无法解决一系列问题。
而 nnUNet 是一个适应多种分割任务的算法。
一个自适应任何新数据集的医学影像分割框架,该框架能根据给定数据集的属性自动调整所有超参数,整个过程无需人工干预。
具体实现方式:
nnUNet 由 2DUNet、3DUNet、U-Net Cascade 组成。
- 2D UNet:处理 2D 图像
- 3DUNet:适合处理3D小尺寸物体的分割。
实际3D训练,因为GPU内存量的限制,只能在切成图像块(patches)训练。
小结构(例如脑肿瘤,海马和前列腺)适合切块,但是像肝这种大结构,切了之后就会损失很多上下文信息。
- U-Net Cascade:解决3D U-Net在具有大图像尺寸的数据集上的实际缺陷。
nnUNet 对 UNet网络架构的两个小改动,修改了激活函数和归一化的方法。
- 激活函数:用 leaky ReLU 而不是 ReLU
- 归一化方法:使用 instance normalization 代替了更流行的 batch normalization
-
ReLUs:当输入的信号小于0时,ReLU函数的输出就是0;当输入的信号大于0时,ReLU函数的输出就直接是输入的信号。比如,如果我们给ReLU函数输入-5,那么输出就是0;如果我们输入5,那么输出就是5。
-
Leaky ReLU: 它是ReLU的一个改进版本。在输入信号小于0时,Leaky ReLU函数的输出不再是0,而是输入的信号乘以一个很小的常数,比如0.01。比如,如果我们给Leaky ReLU函数输入-5,那么输出就是-5乘以0.01等于-0.05;如果我们输入5,那么输出就是5。
-
ReLUs和Leaky ReLU的区别:想象你有一个可以调亮调暗的手电筒,你可以把电筒调暗到最低,但是它仍然会发出一点点微弱的光。这就像Leaky ReLU,即使输入的信号是负的,输出也不会完全是0。而ReLUs就像一个开关,当你关闭它时,它就完全不会发出光。当你打开它时,它会发出与你给它的电力相等的光。
-
不完全为0的好处:因为其将所有的输入负数变为0,在训练中可能很脆弱,很容易导致神经元失活,使其不会在任何数据点上再次激活。对于ReLu中(x<0)的激活,此时梯度为0,因此在下降过程中权重不会被调整。
-
Batch Normalization (BN):假设我们正在烘焙一批饼干,我们要求所有饼干的大小和烘焙时间都一样。为了达到这个目标,我们需要测量一批饼干的平均大小和烘焙时间,然后根据这个标准来制作每一个饼干。这样我们就可以得到一批相似的饼干。这就像BN,它基于一批样本的平均值和标准差来对每一个样本进行调整。
-
Instance Normalization (IN):但是,如果我们想要每一块饼干都有其特有的风味和烹饪方式,那么我们每做一块饼干,都会根据这一块饼干的特性来调整我们的烘焙方式。这就是像IN,它不是参考其他的样本,而是只基于每个单独样本的特性来进行调整。
因此,BN和IN的主要区别在于,BN是对所有样本进行整体标准化,而IN则是对每个样本单独进行标准化。
除了对自适应预处理,还有训练方案和推理的全面设计。
预处理
下面介绍的步骤无需任何用户干预即可执行。
-
裁剪:这就像是把所有多余的空白部分都剪掉,只留下有图像的部分。这样做对大部分数据集影响不大。
-
采样:这就像是把不同大小的图像都变成一样的大小,这样我们的模型可以更好地理解图像的形状和结构。
-
局部扩展:如果一个对象足够大,那么我们可以放大它,让它的一部分占满整个视野,然后分别观察每一部分。
-
归一化:这就像是把所有的分数都变成0到100之间的数,这样我们就可以更好地比较它们。
对于那些裁剪后变小了很多的图像,我们只看图像的部分,其他的都不看。
训练过程
我们在做一些实验,就是让电脑自己学会看医学影像并且能自己找到问题。这个过程就像小朋友学习认识事物一样,需要反复练习并及时纠正错误。我们用到了一种叫做U-Net的学习方法,给电脑看了很多医学影像的例子,让电脑自己逐渐明白怎么样能找到问题。
但是,例子如果太少,电脑就有可能学到的不够好。所以我们用了一种叫做“数据增强”的方法,就像是用一个镜子把电脑看过的医学影像做一些拉长、缩短、旋转等变换,制造出更多的例子给电脑看。这样电脑就能更好地学习并且自己找出问题。
为了让电脑学得更好,我们还会让电脑针对每个影像分块去学习,这样电脑就可以更专注于每个小部分,提高学习效果。
patch 采样:同时,我们还要求每次学习的时候,必须有三分之一以上的影像是有问题的,这样电脑才能更好地认识和理解问题。
推理
在我们训练电脑的时候,我们是把整个医学影像分成很多小块进行学习的。所以,当电脑用学到的知识去检测新的影像的时候,也是分块进行的。我们发现,电脑检查每块影像边缘的部分时,错误的可能性比较大。就好像我们看书一样,边边角角的字可能看得不太清楚。所以,我们会让电脑更重视每块影像中心的部分。
为了让电脑检测得更准确,我们会把医学影像做一些变换,好像用镜子照出另一半影像来,让电脑多看几遍。这个方法就叫做"测试时的数据增强"。
最后,我们实际让网络去检测影像的时候,不止用一个网络,而是用五个网络共同完成,就像几个小朋友一起做一个题目,这样可以提高正确率。
后处理
我们在电脑学习的时候,会让它去看看数据里的各种标签(比如肿瘤,血管等等的位置信息)。如果一个标签(比如肿瘤)总是被包在一个区域里,我们就认为这是一个规律,电脑要学习这个规律。
然后当电脑自己去检测医学影像的时候,也会照着这个规律去做。如果电脑发现了一些标签,但是这些标签并没有被包在一个区域里,那么电脑就会认为这个地方可能判断错了,就会去掉这些标签。
所以,我们这个方法就是让电脑学习一种规律,这种规律可以帮助电脑判断哪些地方可能是错判断的,然后去掉这些错判断,从而让结果更准确。
4行命令使用 nnUNet 训练自己的医学图像分割模型
nnU-Net 需要结构化格式的数据集。
并将原始数据存放在文件夹:nnUNet_raw_data_base/nnUNet_raw_data/
nnUNet_raw_data_base 是安装 nnU-Net 时指定了该文件夹的地址。
每个数据集都存储为单独的“Task”。
统一命名为:Task ID_任务名称。
比如 Task005_Prostate 以’Prostate’作为任务名称任务 ID 为 5。
并将原始数据存放在文件夹:nnUNet_raw_data_base/nnUNet_raw_data/TaskXXX_Prostate

在每一个Task文件夹里,又包含以下文件夹:
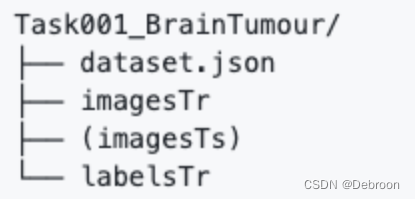
再把你的数据变成 nnU-Net 支持的数据格式,是你使用 nnU-Net 唯一需要做的事情,具体搜索一下教程。
数据预处理:nnUNet_plan_and_preprocess -t XXX --verify_dataset_integrity
- nnUNet_plan_and_preprocess:数据预处理命令
- XXX:TaskXXX_MYTASK(如005, 就写5)可以一次传递多个任务 ID
- verify_dataset_integrity:对数据集执行一些检查以确保它与 nnU-Net 兼容
数据训练:nnUNet_train CONFIGURATION TRAINER_CLASS_NAME TASK_NAME_OR_ID FOLD (additional options)
- nnUNet_train:训练命令
- CONFIGURATION:模型架构,三种Unet: 2D U-Net, 3D U-Net and a U-Net Cascade (U-Net级联)
- TRAINER_CLASS_NAME:使用的 model trainer. 默认为 nnUNetTrainerV2 就可以
- TASK_NAME_OR_ID:任务全名TaskXXX_MYTASK或者是ID号
- FOLD:第几折交叉验证,可选 [0, 1, 2, 3, 4],一共五折。
举例:2D U-Net 训练第三折
- nnUNet_train 2d nnUNetTrainerV2 Task100_adrenal 3
举例:3D full resolution U-Net 训练第二折
- nnUNet_train 3d_fullres nnUNetTrainerV2 Task100_adrenal 2
举例三:3D U-Net cascade 训练第二折
分为两步:
step1 : 3D low resolution U-Net 训练
- nnUNet_train 3d_lowres nnUNetTrainerV2 TaskXXX_MYTASK FOLD
step2: 3D full resolution U-Net 训练
-
nnUNet_train 3d_cascade_fullres nnUNetTrainerV2CascadeFullRes TaskXXX_MYTASK FOLD
3D full resolution U-Net 训练需要使用nnUNetTrainerV2CascadeFullRes
训练完成后,我们当然要看看在测试集上的表现啦。
使用命令 nnUNet_predict。
nnUNet_predict -i INPUT_FOLDER -o OUTPUT_FOLDER -t TASK_NAME_OR_ID -m CONFIGURATION
- INPUT_FOLDER: 测试数据地址
- OUTPUT_FOLDER:分割数据存放地址
- TASK_NAME_OR_ID:任务全名TaskXXX_MYTASK或者是ID号
- CONFIGURATION:使用的什么架构,2d or 3d_fullres or 3d_cascade_fullres
如:
- nnUNet_predict -i /home/…/nnunet_file/nnUNet_raw/nnUNet_raw_data/Task100_adrenal/imagesTs -o /home/…/nnunet_file/output -t 100 -m 2d -f 2



数据可视化概述)






)






 E题题解)

