原仓库地址如下,github有时候无法访问,等一段时间再试就行:
YongxinLiu/EasyMetagenome: Easy Metagenome Pipeline (github.com)
相关文章,看文章更清晰这个可干啥:
EasyAmplicon: An easy‐to‐use, open‐source, reproducible, and community‐based pipeline for amplicon data analysis in microbiome research - Liu - 2023 - iMeta - Wiley Online Librarypractical guide to amplicon and metagenomic analysis of microbiome data | Protein & Cell | Oxford Academic (oup.com)
其他相关资源供参考:
EasyMicrobiome-易扩增子、易宏基因组等分析流程依赖常用软件、脚本文件和数据库注释文件-CSDN博客
EasyAmplicon (易扩增子)-扩增子高通量序列分析软件流程及脚本-详细使用方法——来自刘永鑫团队的秘籍-CSDN博客
相关资源为照顾国内伙伴已经给了国内资源链接:
ftp://download.nmdc.cn/tools/
百度网盘 请输入提取码
1、环境准备及软件安装
看看仓库代码就知道已经很用心了,安装的主要配置脚本都在0Install.sh;当然这里面有些软件和数据库大家可以边安装,边搜索网上的最新版本,尤其是数据库,大部分都有更新了,软件不一定要最新版本,但在合适的时候最新版还是能带来一些好处的。
当然,这里还需要给大家说一下的这些软件如果要配置好所有的数据库是需要占用较大的空间的,另外,这些数据库最好是放在固态硬盘上,在多server节点同时共享使用的时候读取大数据库会成为分析过程中的瓶颈
这里面有些环境配置大家自己了解就好不一定完全参考其脚本,重点在于自己知道自己需要使用什么样的环境和软件,配置哪些数据库。
易宏基因组软件和数据库 EasyMetagenome software & database
# 版本Version: 1.20, 2023/11/23
# 操作系统Operation System: Linux Ubuntu 22.04+ / CentOS 8+
# 主页Homepage: https://github.com/YongxinLiu/EasyMetagenome
所有软件和数据库可从官网下载,All software and databases can be downloaded from the official website, 备选 optional:
中科院微生物所Institute of Microbiology, Chinese Academy of Sciences:ftp://download.nmdc.cn/tools/ (FileZilla访问)
百度网盘Baidu Netdisk:https://pan.baidu.com/s/1Ikd_47HHODOqC3Rcx6eJ6Q?pwd=0315
一、数据预处理 Data preprocessing
初始化:每次开始安装必须运行下面代码 Initialization: The following code must be run every time when installation starts
安装前准备:软件和数据库位置 Before Installation: Software and Database Locations
数据库安装位置Database Locations,默认~/db目录(无需管理权限),管理员可选/db
db=~/db
mkdir -p ${db} && cd ${db}
# 软件安装位置Software installation location,默认为~/miniconda3,测试服务器为/anaconda3
soft=~/miniconda3
经常使用的服务器环境,可把全文${db}和${soft}替换为绝对路径,将不再需要每次读取以上环境变量
# In the frequently used server environment, you can replace the variable ${db} and ${soft} with absolute paths, and you will no longer need to run the above environment variables every time
可选:初始化环境变量,可能提高软件安装成功率
# Optional: Initialize environment variables, which may improve the success rate of software installation
PATH=${soft}/bin:${soft}/condabin:/usr/local/sbin:/usr/local/bin:/usr/sbin:/usr/bin:/sbin:/bin:${db}/EasyMicrobiome/linux:${db}/EasyMicrobiome/script
echo $PATH
EasyMetagenome流程Pipeline
EasyMetagenome流程,包括流程安装、使用和可视化脚本,以及流程测试数据和结果正对照,网址:https://github.com/YongxinLiu/EasyMetagenome
三种下载方法:依赖尝试/任选其一至成功即可
# 方法1. 网页 https://github.com/YongxinLiu/EasyMetagenome 中Code - Download ZIP下载压缩包,上传至服务器
# 解压,Command 'unzip' not found使用sudo apt install unzip安装
unzip EasyMetagenome-master.zip
# 改名
mv EasyMetagenome-master EasyMetagenome# 方法2. 微生物所备用链接,可能不是最新版
wget -c ftp://download.nmdc.cn/tools/soft/EasyMetagenome.tar.gz
tar xvzf EasyMetagenome.tar.gz# 方法3. git下载,需安装git
git clone https://github.com/YongxinLiu/EasyMetagenome
# 旧版更新
cd EasyMetagenome && git pull && cd ../EasyMicrobiome软件和数据库合集dependencies
EasyMetagenome依赖EasyMicrobiome,其包括众多脚本、软件和数据库的集合,网址:https://github.com/YongxinLiu/EasyMicrobiome
# 方法1. 网页中下载
# https://github.com/YongxinLiu/EasyMicrobiome 中Code Download ZIP下载压缩包,上传至服务器,并解压
unzip EasyMicrobiome-master.zip
mv EasyMicrobiome-master EasyMicrobiome# 方法2. 备用链接下载
wget -c ftp://download.nmdc.cn/tools/soft/EasyMicrobiome.tar.gz
tar -xvzf EasyMicrobiome.tar.gz# 方法3. git下载,需安装git
git clone https://github.com/YongxinLiu/EasyMicrobiome
# 旧版更新
cd EasyMicrobiome && git pull && cd ../软件安装
# 添加linux命令可执行权限
chmod +x `pwd`/EasyMicrobiome/linux/* `pwd`/EasyMicrobiome/script/*
# 添加环境变量
echo "export PATH=\"\$PATH:`pwd`/EasyMicrobiome/linux:`pwd`/EasyMicrobiome/script\"" >> ~/.bashrc
source ~/.bashrc
echo $PATH软件管理器Conda
# 下载最新版miniconda3,70M
wget -c https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
# 安装,-b批量,-f无提示,-p目录,许可协议打yes
bash Miniconda3-latest-Linux-x86_64.sh -b -f -p ${soft}
# 激活,然后关闭终端重开,提示符前出现(base)即成功
${soft}/condabin/conda init
source ~/.bashrc
# 查看版本,conda 23.7.3, python 3.11.4
conda -V
python --version
# 添加常用频道
conda config --add channels bioconda # 生物软件
conda config --add channels conda-forge # Highest priority# conda默认配置文件为 ~/.condarc 查看配置文件位置
conda install mamba -c conda-forge -y
mamba install pandas -c conda-forge -y
mamba install conda-pack -c conda-forge -y
conda config --set channel_priority strict
conda config --show-sources
# 查看虚拟环境列表
conda env list更多conda中文安装使用教程参考:[Nature Method:Bioconda解决生物软件安装的烦恼](https://mp.weixin.qq.com/s/SzJswztVB9rHVh3Ak7jpfA)
[一文掌握Conda软件安装:虚拟环境、软件通道、加速solving、跨服务器迁移](https://mp.weixin.qq.com/s/tKAU09_w7Cu7khA9M2EGEQ)
质控Quality control: kneaddata/fstqc/multiqc/fastp
**注:直接安装、下载解压安装,二选一。一种方法不成功,尝试另一种。**
BioConda: https://bioconda.github.io/recipes/kneaddata/README.html
方法1.kneaddata直接安装
# 新建kneaddata环境
conda create -y -n kneaddata
conda activate kneaddatafastqc质量评估,multiqc评估报告汇总,kneaddata质量控制流程,fastp质控工具
mamba install kneaddata fastqc multiqc fastp r-reshape2 -y 方法2.kneaddata下载解压安装
# 指定conda文件名
s=kneaddata
# 下载,可选NMDC、百度云等
wget -c ftp://download.nmdc.cn/tools/conda/${s}.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/${s}
tar -xvzf ${s}.tar.gz -C ${soft}/envs/${s}
# 启动环境
conda activate ${s}
# 初始化环境
conda unpackkneaddata安装测试
fastqc -v # v0.12.1
kneaddata --version # 0.12.0
trimmomatic -version # 0.39
bowtie2 --version # 2.5.1
multiqc --version # 1.15# (可选)安装软件打包,f覆盖输出文件,ignore跳过修改检测
n=kneaddata
conda pack -f --ignore-missing-files -n ${n} -o ${n}.tar.gzkneaddata数据库下载
# 查看可用数据库
kneaddata_database
# 包括人基因组bowtie2/bmtagger、人类转录组、核糖体RNA和小鼠基因组# 下载人基因组bowtie2索引 3.44 GB
mkdir -p ${db}/kneaddata/human
kneaddata_database --download human_genome bowtie2 ${db}/kneaddata/human# 备用链接下载人类基因组至上述目录,并解压
cd ${db}/kneaddata/human
wget -c ftp://download.nmdc.cn/tools/meta/kneaddata/human_genome/Homo_sapiens_hg37_and_human_contamination_Bowtie2_v0.1.tar.gz
tar xvzf Homo_sapiens_hg37_and_human_contamination_Bowtie2_v0.1.tar.gz# 下载小鼠基因组bowtie2索引 2.83 GB
mkdir -p ${db}/kneaddata/mouse
kneaddata_database --download mouse_C57BL bowtie2 ${db}/kneaddata/mouse
# 备用手动下载
cd ${db}/kneaddata/mouse
wget -c http://huttenhower.sph.harvard.edu/kneadData_databases/mouse_C57BL_6NJ_Bowtie2_v0.1.tar.gz
tar xvzf mouse_C57BL_6NJ_Bowtie2_v0.1.tar.gzkneaddata自定义参考基因组索引
**(任何构建好的 bowtie2 索引都可,放置位置不限;如果有多个宿主,可以把多个宿主参考基因组序列合并后构建索引)**
自定义基因组构建索引,大多数基因组可在ensembl genome下载。此处以拟南芥为例,访问 http://plants.ensembl.org/index.html ,选择Arabidopsis thaliana —— Download DNA sequence (FASTA),选择toplevel右键复制链接,填入下面链接处
# 创建子目录
mkdir -p ${db}/kneaddata/ath
cd ${db}/kneaddata/ath
# 下载
wget -c http://ftp.ensemblgenomes.org/pub/plants/release-51/fasta/arabidopsis_thaliana/dna/Arabidopsis_thaliana.TAIR10.dna.toplevel.fa.gz
mv Arabidopsis_thaliana.TAIR10.dna.toplevel.fa.gz tair10.fa.gz
# wget -c ftp://download.nmdc.cn/tools/meta/kneaddata/tair10.fa.gz
# 解压
gunzip tair10.fa.gz
# bowtiew建索引,输入文件,输出文件前缀,4线程2分
bowtie2-build -f tair10.fa tair10 --threads 4二、基于读长分析 Read-based (HUMAnN3/Kraken2)
宏基因组基于读长的分析 HUMAnN3/MetaPhlAn4/GraPhlAn
HUMAnN3+MetaPhlAn4为目前最新版,目前最广泛使用的HUMAnN2安装见附录
HUMAnN3直接安装
# 安装HUMAnN3.7+MetaPhlAn4
conda create -n humann3
conda activate humann3
conda install humann=3.7 -c bioconda -c conda-forge
# 打包(可选)
conda pack -f -n humann3 -o humann3.tar.gzHUMAnN3解包安装
# 下载
wget -c ftp://download.nmdc.cn/tools/conda/humann3.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/humann3
tar -xvzf humann3.tar.gz -C ${soft}/envs/humann3
# 启动环境
conda activate humann3
# 初始化环境
conda unpackHUMAnN3安装测试
# 记录核心软件版本
humann --version # v3.7
metaphlan -v # 4.0.6 (1 Mar 2023)
diamond help | head -n 1 # v2.1.8.162
# 测试
humann_testHUMAnN3物种和功能数据库
# 显示可用分类、泛基因组和功能数据库
humann_databases# 安装数据库
cd ${db}
mkdir -p ${db}/humann3 # 建立下载目录
# 微生物泛基因组 16 GB
humann_databases --download chocophlan full ${db}/humann3
# 功能基因diamond索引 20 GB
humann_databases --download uniref uniref90_diamond ${db}/humann3
# 输助比对数据库 2.6 GB
humann_databases --download utility_mapping full ${db}/humann3# humann3数据库无法自动下载,备用链接下载安装
wget -c ftp://download.nmdc.cn/tools/meta/humann3/full_chocophlan.v201901_v31.tar.gz
wget -c ftp://download.nmdc.cn/tools/meta/humann3/uniref90_annotated_v201901b_full.tar.gz
wget -c ftp://download.nmdc.cn/tools/meta/humann3/full_mapping_v201901b.tar.gz
# 安装、解压
mkdir -p ${db}/humann3/chocophlan
tar xvzf full_chocophlan.v201901_v31.tar.gz -C ${db}/humann3/chocophlan
mkdir -p ${db}/humann3/uniref
tar xvzf uniref90_annotated_v201901b_full.tar.gz -C ${db}/humann3/uniref
mkdir -p ${db}/humann3/utility_mapping
tar xvzf full_mapping_v201901b.tar.gz -C ${db}/humann3/utility_mapping# 设置数据库位置
# 显示参数
humann_config --print
# 如修改线程数,推荐3-8,根据实际情况调整
humann_config --update run_modes threads 8
# 设置核酸、蛋白和注释库位置
humann_config --update database_folders nucleotide ${db}/humann3/chocophlan
humann_config --update database_folders protein ${db}/humann3/uniref
humann_config --update database_folders utility_mapping ${db}/humann3/utility_mapping
# 核对设置结果
humann_config --printMetaPhlAn4物种数据库
个人整理了一个详细使用流程:
202310-宏基组学物种分析工具-MetaPhlAn4安装和使用方法-Anaconda3- centos9 stream-CSDN博客
# MetaPhlAn4数据库下载2022数据和索引3G+20G
mkdir -p ${db}/metaphlan4
cd ${db}/metaphlan4# 官网下载
wget -c http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/mpa_vOct22_CHOCOPhlAnSGB_202212.tar
wget -c http://cmprod1.cibio.unitn.it/biobakery4/metaphlan_databases/bowtie2_indexes/mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar
tar xvf mpa_vOct22_CHOCOPhlAnSGB_202212.tar
tar xvf mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar# 官方没有压缩体积大下载慢,备用国内百度链接:https://pan.baidu.com/s/1Ikd_47HHODOqC3Rcx6eJ6Q?pwd=0315 或 微生物所FTP ftp://download.nmdc.cn/tools/meta 下载压缩包
wget -c ftp://download.nmdc.cn/tools/meta/metaphlan4/mpa_vOct22_CHOCOPhlAnSGB_202212.tar.gz
wget -c ftp://download.nmdc.cn/tools/meta/metaphlan4/mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar.gz
tar xvzf mpa_vOct22_CHOCOPhlAnSGB_202212.tar.gz
tar xvzf mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar.gz
# 可选(制作下载文件和md5值)
gunzip mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar.gz
md5sum mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.tar > mpa_vOct22_CHOCOPhlAnSGB_202212_bt2.md5生物标记鉴定和可视化LEfSe
方法1. 在线ImageGP http://www.bic.ac.cn/ImageGP/ 选择左侧LEfSe方法2. 下载conda预安装包解压n=lefse
# 下载
wget -c ftp://download.nmdc.cn/tools/conda/${n}.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/${n}
tar -xvzf ${n}.tar.gz -C ${soft}/envs/${n}
# 启动环境
conda activate ${n}
# 初始化环境
conda unpack方法3. 直接安装:经常无法安装,安装的代码也有可能存在问题mamba create -n lefse lefse -c bioconda -y物种注释Kraken2/bracken/krakentools/krona
kraken2 基于LCA算法的物种注释 https://ccb.jhu.edu/software/kraken/Kraken2解包安装# 下载
wget -c ftp://download.nmdc.cn/tools/conda/kraken2.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/kraken2
tar -xvzf kraken2.tar.gz -C ${soft}/envs/kraken2
# 启动环境
conda activate kraken2
# 初始化环境
conda unpackKraken2直接安装,居然只安了2.0.7mamba create -n kraken2 -y -c bioconda kraken2 bracken krakentools krona r-optparse记录软件版本kraken2 --version # 2.1.2Kraken2数据库安装
下载数据库(NCBI每2周更新一次),记录下载日期和大小。需根据服务器内存、使用目的选择合适方案。--standard标准模式下只下载5种**标准数据库:古菌archaea、细菌bacteria、人类human、载体UniVec_Core、病毒viral**。也可选直接下载作者构建的索引,还包括bracken的索引。链接:https://benlangmead.github.io/aws-indexes/k2 (10/9/2023版)。 注:中科院网络下载较快,家里和农科院较慢,有时新版会有错误,可以退回旧版
方案1. 下载标准+原生动物+真菌 16GB (PlusPF-16) v=k2_pluspf_16gb_20231009
mkdir -p ~/db/kraken2/pluspf16g
cd ~/db/kraken2
wget -c https://genome-idx.s3.amazonaws.com/kraken/${v}.tar.gz
# 备用链接
wget -c ftp://download.nmdc.cn/tools/meta/kraken2/${v}.tar.gz
tar xvzf ~/db/kraken2/${v}.tar.gz -C ~/db/kraken2/pluspf16g方案2. 下载标准+原生动物+真菌 69GB (PlusPF) v=k2_pluspf_20231009
mkdir -p ~/db/kraken2/pluspf
cd ~/db/kraken2
wget -c https://genome-idx.s3.amazonaws.com/kraken/${v}.tar.gz
# 备用链接
wget -c ftp://download.nmdc.cn/tools/meta/kraken2/${v}.tar.gz
tar xvzf ~/db/kraken2/${v}.tar.gz -C ~/db/kraken2/pluspf方案3. 下载标准+原生动物+真菌+植物完整库 144G (PlusPFP) 指定解压目录,包括时间和类型v=k2_pluspfp_20231009
mkdir -p ~/db/kraken2/pluspfp
cd ~/db/kraken2
wget -c https://genome-idx.s3.amazonaws.com/kraken/${v}.tar.gz
tar xvzf ${db}/kraken2/${v}.tar.gz -C pluspfp
三、组装 Assemble-based
组装、注释和定量 megahit/spades/quast/cd-hit/emboss/salmon/prodigal
megahit解包安装
# 下载
wget -c ftp://download.nmdc.cn/tools//conda/megahit.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/megahit
tar -xvzf megahit.tar.gz -C ${soft}/envs/megahit
# 启动环境
conda activate megahit
# 初始化环境
conda unpackmegahit直接安装
mamba create -y -n megahit megahit spades quast cd-hit emboss salmon prodigal
conda activate megahit megahit安装后测试
megahit -v # MEGAHIT v1.2.9
metaspades.py -v # metaSPAdes v3.15.4
metaquast.py -v # MetaQUAST v5.0.2
cd-hit -v | grep version # CD-HIT v4.8.1
embossversion # EMBOSS v6.6
salmon -v # salmon v1.8蛋白同源综合注释eggNOG
eggNOG http://eggnogdb.embl.de
eggNOG解包安装
# 下载
wget -c ftp://download.nmdc.cn/tools/conda/eggnog.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/eggnog
tar -xvzf eggnog.tar.gz -C ${soft}/envs/eggnog
# 启动环境
conda activate eggnog
# 初始化环境
conda unpackeggNOG直接安装
# 新建环境并进入
conda create -n eggnog -y
conda activate eggnog
# 安装eggnog比对工具emapper
conda install eggnog-mapper -y -c bioconda -c conda-forgeeggNOG安装测试
emapper.py --version # 2.1.12
# Expected eggNOG DB version: 5.0.2 / Installed eggNOG DB version: 5.0.2 /
# Diamond version found: diamond version 2.0.15 / MMseqs2 version found: 13.45111eggNOG数据库安装
# 下载常用数据库,注意设置下载位置
mkdir -p ${db}/eggnog && cd ${db}/eggnog
# -y默认同意,-f强制下载,eggnog.db.gz 6.3G+4.9G,解压后48G
download_eggnog_data.py -y -f --data_dir ${db}/eggnog# 百度或微生物所备用链接下载eggnog.tar.gz
wget -c ftp://download.nmdc.cn/tools/meta/eggnog/eggnog.tar.gz
tar xvzf eggnog.tar.gz
# 查看版本时会显示默认数据位置
emapper.py --version # 2.1.12
# 链接至默认目录,注意按实际情况修改
ln -sf ${db}/eggnog ${soft}/envs/eggnog/lib/python3.9/site-packages/data/
# 复制数据至内存中加速比对
# cp eggnog.* /dev/shm碳水化合物CAZy
dbCAN3 http://bcb.unl.edu/dbCAN2
# 创建数据库存放目录并进入
mkdir -p ${db}/dbcan3 && cd ${db}/dbcan3
# 下载序列和描述
wget -c https://bcb.unl.edu/dbCAN2/download/Databases/V12/CAZyDB.07262023.fa
wget -c https://bcb.unl.edu/dbCAN2/download/Databases/V12/CAZyDB.08062022.fam-activities.txt
# 提取基因家簇对应注释
grep -v '#' CAZyDB.08062022.fam-activities.txt | sed 's/ //'| \sed '1 i CAZy\tDescription' > CAZy_description.txt
# 打包压缩
tar -cvzf CAZyDB.tar.gz CAZyDB.07262023.fa CAZyDB.08062022.fam-activities.txt CAZy_description.txt# 备用数据库下载并解压(待上传)
wget -c ftp://download.nmdc.cn/tools/meta/dbcan3/CAZyDB.tar.gz
tar xvzf CAZyDB.tar.gz# diamond建索引,1G,11s
diamond --version # 2.1.8
time diamond makedb --in CAZyDB.07262023.fa --db CAZyDB抗生素抗性基CARD/rgi
CARD官网:https://card.mcmaster.ca
RGI Github: https://github.com/arpcard/rgi
rgi解包安装
# 下载
wget -c ftp://download.nmdc.cn/tools/conda/rgi.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/rgi
tar -xvzf rgi.tar.gz -C ${soft}/envs/rgi
# 启动环境
conda activate rgi
# 初始化环境
conda unpackrgi直接安装
mamba create -y -n rgi6 rgi=6.0.3
conda activate rgi6
# (可选)打包,待上传
n=rgi6
conda pack -f --ignore-missing-files -n ${n} -o ${n}.tar.gzrgi版本和数据库部署
# 查看版本 6.0.3
rgi main -v# 数据库部署
mkdir -p ${db}/card && cd ${db}/card
# 下载最新版数据库,3.8M (2023-10-2, 3.2.8)
wget -c https://card.mcmaster.ca/latest/data
# 解压后35M
tar -xvf data ./card.json
# 加载数据库
rgi load --card_json card.json
# 宏基因组分析扩展数据库和加载
rgi card_annotation -i card.json
mv card_database_v3.2.8_all.fasta card.fasta
rgi load -i card.json --card_annotation card.fasta四、分箱挖掘单菌基因组Binning
metawrap分箱binning
软件主页:https://github.com/bxlab/metaWRAP
metawrap下载安装
# 下载
wget -c ftp://download.nmdc.cn/tools/conda/metawrap.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/metawrap
tar -xvzf metawrap.tar.gz -C ${soft}/envs/metawrap
# 启动环境
conda activate metawrap
# 初始化环境
conda unpackmetawrap conda安装
mamba create -y --name metawrap --channel ursky metawrap-mg=1.3.2
conda activate metawrapmetawrap相关数据库
cd ${db} CheckM用于Bin完整和污染估计和物种注释mkdir -p checkm && cd checkm
# 下载文件275 MB,解压后1.4 GB
wget -c https://data.ace.uq.edu.au/public/CheckM_databases/checkm_data_2015_01_16.tar.gz
tar -xvf *.tar.gz
# 设置数据库位置,直接2次回车默认为当前位置
checkm data setRootNCBI核酸和物种信息(可选)
# 核酸
mkdir -p ${db}/NCBI/nt
(cd ${db}/NCBI/nt; wget -c ftp://ftp.ncbi.nlm.nih.gov/blast/db/nt.*.tar.gz)
(cd ${db}/NCBI/nt; for i in *.tar.gz; do tar xzf $i; done)
# 可能会出现个别库下载不完整的情况,删了重下,不要续传
# 物种信息,压缩文件45M,解压后351Mmkdir -p ${db}/NCBI/tax
(cd ${db}/NCBI/tax; wget -c ftp://ftp.ncbi.nlm.nih.gov/pub/taxonomy/taxdump.tar.gz)
(cd ${db}/NCBI/tax; tar -xvzf taxdump.tar.gz)数据库位置设置
which config-metawrap
# 配置文件通常为~/miniconda3/envs/metawrap/bin/config-metawrap
# 使用Rstudio/vim等文本编辑器来修改数据库的位置
# config-metawrap文件中内容如下# Paths to metaWRAP scripts (dont have to modify)
mw_path=$(which metawrap)
bin_path=${mw_path%/*}
SOFT=${bin_path}/metawrap-scripts
PIPES=${bin_path}/metawrap-modules# CONFIGURABLE PATHS FOR DATABASES (see 'Databases' section of metaWRAP README for details)
# path to kraken standard database
KRAKEN_DB=~/KRAKEN_DB
KRAKEN2_DB=~/db/kraken2/pluspf/# path to indexed human (or other host) genome (see metaWRAP website for guide). This includes .bitmask and .srprism files
BMTAGGER_DB=~/BMTAGGER_DB# paths to BLAST databases
BLASTDB=~/db/NCBI/nt
TAXDUMP=~/db/NCBI/taxdrep基因组去冗余
挑单菌测序的基因组存在大量冗余。metawrap混合分箱的结果中冗余度非常低,甚至无冗余。而单样本、分批次分箱的结果中存在大量冗余,需要采用drep获得非冗余的基因组。
GitHub: https://github.com/MrOlm/drep
Conda: https://bioconda.github.io/recipes/drep/README.html
drep 基因组去冗余解包安装
# 下载dRep v3.2.3无法安装依赖chechm,仍用旧版2.6.2(500M),这个压缩包没有checkm且版本为3.4.2
wget -c ftp://download.nmdc.cn/tools/conda/drep.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/drep
tar -xvzf drep.tar.gz -C ${soft}/envs/drep
# 启动环境
conda activate drep
# 初始化环境
conda unpack
dRep -hdrep 基因组去冗余直接安装
# 2023/9/12尝试,仍无法安装checkm
mamba create -y -n drep drep=3.4.3
conda activate drep
# 不满足依赖关系
mamba install checkm-genome -y
dRep -hdrep 数据库构建
CheckM用于Bin完整和污染估计和物种注释,安装过metawrap已经下载完成mkdir -p ${db}/checkm && cd ${db}/checkm
# 下载文件275 MB,解压后1.4 GB
wget -c https://data.ace.uq.edu.au/public/CheckM_databases/checkm_data_2015_01_16.tar.gz
tar -xvf *.tar.gz
# 设置数据库位置,直接2次回车默认为当前位置
checkm data setRoot `pwd`coverm基因组定量
conda安装
conda create -n coverm -y
conda activate coverm
conda install coverm -c bioconda -y
# conda安装后打包(可选)
conda pack -f --ignore-missing-files -n coverm -o coverm.tar.gz压缩包安装
# 指定conda文件名
s=coverm
# 下载,可选NMDC、百度云等
# wget -c ftp://download.nmdc.cn/tools/conda/${s}.tar.gz
# 指定安装目录
mkdir -p ~/miniconda3/envs/${s}
tar -xvzf ${s}.tar.gz -C ~/miniconda3/envs/${s}
# 启动环境
conda activate ${s}
# 初始化环境
conda unpackGTDB细菌基因组注释和进化分析
Github: https://github.com/Ecogenomics/GTDBTk
GTDB-Tk是一个软件工具包,用于根据基因组数据库分类法GTDB为细菌和古细菌基因组分配客观的分类法。它旨在与最近的进展一起使用,从而可以直接对环境样本中获得数百或数千个由宏基因组组装的基因组(MAG)进行物种分类注释。它也可以用于分离和单细胞的基因组物种注释。
本次测试版本为 gtdbtk-2.2.6,Release 07-RS207v2 (11th May 2022)。
硬件要求:内存200Gb,硬盘66Gb,64核1小时可分析1000个细菌基因组
GTDB-Tk直接安装
# gtdbtk-2.3.2, 2023-7-8
n=gtdbtk2.3
mamba create -y -n ${n} -c conda-forge -c bioconda gtdbtk=2.3.2
# 检查版本
gtdbtk -v # 2.3.2# conda pack软件打包一次
# --exclude gtdbtk-2.3.2 指定排除数据库
conda pack -n ${n} -o ${n}.tar.gz --exclude gtdbtk-2.3.2 --ignore-editable-packages --ignore-missing-files
chmod 755 *GTDB-Tk解包安装
soft=~/miniconda3
# 下载,目前为2.1,需更新为2.3
wget -c ftp://download.nmdc.cn/tools/conda/gtdbtk.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/gtdbtk
tar -xvzf gtdbtk.tar.gz -C ${soft}/envs/gtdbtk
# 启动环境
conda activate gtdbtk
# 初始化环境
conda unpackGTDB-Tks数据库安装
download-db.sh自动下载数据库,将下载至conda中的envs/gtdbtk/share/gtdbtk-2.3.2/db/,我们修改为~/db/gtdb中
conda activate gtdbtk2.3
# download-db.sh中,修改数据库下载位置,的 wget 建议改成wget -c 防止覆盖
sed -i 's#miniconda3/envs/gtdbtk2.3/share/gtdbtk-2.3.2/db#db/gtdb2.3#;s/wget /wget -c /' ${soft}/envs/gtdbtk2.3/bin/download-db.sh
# 下载数据,78G
download-db.sh(备选)下面无法下载时手动下载和配置GTDB数据库mkdir -p ${db}/gtdb2.3 && cd ${db}/gtdb2.3
# 下载解压
wget -c https://data.gtdb.ecogenomic.org/releases/release214/214.0/auxillary_files/gtdbtk_r214_data.tar.gz
# 再运行, gtdb配置数据库
download-db.sh# 备用链接和手工解压,指定安装完整路径
wget -c ftp://download.nmdc.cn/tools/meta/gtdb/gtdbtk_r214_data.tar.gz
tar xvzf gtdbtk_r207_v2_data.tar.gz -C ./ --strip 1
conda env config vars set GTDBTK_DATA_PATH="/data/meta/db/gtdb/"五、单菌基因组、病毒组等其他软件
CheckM2
Conda主页:https://bioconda.github.io/recipes/checkm2/README.html
软件主页:https://github.com/chklovski/CheckM2
# 软件安装
mamba create --name checkm2 checkm2
conda activate checkm2
checkm2 -h # CheckM2 v1.0.1
# 数据库安装
mkdir ~/db/checkm2
checkm2 database --download --path ~/db/checkm2
# 报错:checkm2.zenodo_backpack.ZenodoConnectionException: Connection error: HTTPSConnectionPool(host='zenodo.org', port=443): Max retries exceeded with url: /record/5571251 (Caused by NewConnectionError('<urllib3.connection.HTTPSConnection object at 0x7f335c64abe0>: Failed to establish a new connection: [Errno 111] Connection refused'))
# 在github中搜索和查找issues无解答,提新issue https://github.com/chklovski/CheckM2/issues
# 数据库 https://zenodo.org/records/5571251 下载,需要VPNexport CHECKM2DB="path/to/database"
# 测试
checkm2 testrun# 运行,输入目录或文件列表
checkm2 predict --threads 30 --input <folder_with_bins> --output-directory <output_folder>
checkm2 predict --threads 30 --input ../bin1.fa ../../bin2.fna /some/other/directory/bin3.fasta --output-directory <output_folder> 常见问题
软件和数据库国内备份
国家微生物科学数据中心 —— 数据下载
http://nmdc.cn/datadownload,可以使用Filezilla直接连接 ftp://download.nmdc.cn/tools
本资源由宏基因组平台发起,微生物所提供服务器,宏基因组团队负责维护的常用软件、扩增子和宏基因组数据库的国内下载链接,解决常用数据库下载慢、或无法下载的问题。同时提供定制的软件、数据库索引,节约大家下载时间,节省数据库编制索引的计算资源消耗。
# humann3为例
mkdir -p ~/db/humann3 && cd ~/db/humann3
site=ftp://download.nmdc.cn/tools
wget -c ${site}/humann3/full_chocophlan.v296_201901.tar.gz
wget -c ${site}/humann3/uniref90_annotated_v201901.tar.gz
wget -c ${site}/humann3/full_mapping_v201901.tar.gz
mkdir -p chocophlan uniref utility_mapping
tar xvzf full_chocophlan.v296_201901.tar.gz -C chocophlan/
tar xvzf uniref90_annotated_v201901.tar.gz -C uniref/
tar xvzf full_mapping_v201901.tar.gz -C utility_mapping/### 百度云备份链接
https://pan.baidu.com/s/1Ikd_47HHODOqC3Rcx6eJ6Q?pwd=0315
下载的tar.gz压缩包,可放置于指定目录,使用`tar -xvzf *.tar.gz`解压
# 大文件的分卷压缩和解压 以kraken2为例
cd ~/db/kraken2
# https://www.cnblogs.com/wang--lei/p/9046643.html
# 文件夹kraken2/打包压缩,1h
tar -zcvf kraken2.tar.gz kraken2/
# b分割为指定大小文件G/M/K,-d数字,a序列长度,输入和输出前缀
split -b 13G -d -a 1 kraken2.tar.gz kraken2.tar.gz.
# 一行命令打包并分割
tar -zcvf kraken2.tar.gz kraken2idx/ | split -b 19G -d -a 1 - kraken2.tar.gz.
# 分割后合并及解压缩
cat kraken2.tar.gz.* | tar -zxvkneaddata常见问题
kneaddata运行提示java版本不支持
# 解决思路,新建虚拟环境,安装kneaddata,再安装对应java版本
# 务必指定2.7,软件依赖2.7的python,但conda会自动安装3.6,运行报错
conda create -n kneaddata python=2.7
conda activate kneaddata
conda install openjdk=8.0.152
conda install kneaddata=0.6.1解压失败-重新下载再安装
tar -xvzf kneaddata.tar.gz -C ~/miniconda3/envs/kneaddata解压文件提示如下错误gzip: stdin: invalid compressed data--format violated
tar: Unexpected EOF in archive
tar: Unexpected EOF in archive
tar: Error is not recoverable: exiting now检查md5值确认文件是否不同md5sum kneaddata.tar.gz当前为d26125bee1def1faa99d03a9715bf392
原文件为9fa47a364096b2c33be52a91850b2cde删除当前文件并重新下载即可rm kneaddata.tar.gz
wget ftp://download.nmdc.cn/tools//conda/kneaddata.tar.gzLefse在Rstudio中运行命令调用R版本问题的解决
# 在Rstudio中默认调用Rstudio的R,具体写在/etc/rstudio/rserver.conf
# 或在R中用Sys.getenv()["R_HOME"],在rpy2中print(robjects.r)可以查看其调用的r版本
# 指定lefse调用的R版本,需根据conda实际目录修改
sed -i "2 i os.environ['R_HOME'] = '~/miniconda3/envs/meta/lib/R/'" \~/miniconda3/envs/meta/share/lefse-1.0.8.post1-1/lefse.pyKraken2
### 定制数据库
官方教程详见 https://github.com/DerrickWood/kraken2/blob/master/docs/MANUAL.markdown
本地构建最完整索引,自定义微生物数据库,如标准+真菌+原生动物+质粒+植物
mkdir -p ${db}/kraken2/kraken2_self
conda activate kraken2
# 显示帮助
kraken2-build -h
# 下载物种注释
kraken2-build --download-taxonomy --threads 24 --db ${db}/kraken2/kraken2_self
# 下载数据库,需要12-24小时
for i in archaea bacteria UniVec_Core viral human fungi plasmid protozoa plant; dokraken2-build --download-library $i --threads 24 --db ${db}/kraken2/kraken2_self
done
# 确定的库建索引,4p,4h
time kraken2-build --build --threads 48 --db ${db}/kraken2/kraken2_self
# bracken索引,长度推荐100/150, 24p, 1h;
time bracken-build -d ./ -t 24 -k 35 -l 100
time bracken-build -d ./ -t 24 -k 35 -l 150Perl版本不对
常见问题:Perl版本不对,人工指定perl版本如下
PERL5LIB=~/miniconda3/envs/kraken2/lib/site_perl/5.26.2/x86_64-linux-thread-multi:~/miniconda3/envs/kraken2/lib/site_perl/5.26.2:~/miniconda3/envs/kraken2/lib/5.26.2/x86_64-linux-thread-multi:~/miniconda3/envs/kraken2/lib/5.26.2salmon手动安装和使用
# 如不可用,尝试下载二进制和添加环境变量
wget https://github.com/COMBINE-lab/salmon/releases/download/v0.14.0/salmon-0.14.0_linux_x86_64.tar.gz
tar xvzf salmon-0.14.0_linux_x86_64.tar.gz
cp -rf salmon-latest_linux_x86_64/ ${soft}/envs/metagenome_env/share/salmon
# 或者直接使用软件全路径
${soft}/envs/metagenome_env/share/salmon/bin/salmon -v # 0.14.0MetaWRAP分箱
shorten_contig_names.py报错
更新 ${soft}/envs/metawrap/bin/metawrap-scripts/shorten_contig_names.py 脚本
#!/usr/bin/env python2.7
import sys
shorten=False
for line in open(sys.argv[1]):if line[0]!=">":print line.rstrip()else:if shorten==True:#print "_".join(line.rstrip().split("_")[:4])lineL = line.rstrip().split("_")new_line = '_'.join([lineL[0], lineL[1], lineL[3]])print new_line[:20]elif len(line)>20 and len(line.split("_"))>5:lineL = line.rstrip().split("_")new_line = '_'.join([lineL[0], lineL[1], lineL[3]])#print "_".join(line.rstrip().split("_")[:4])print new_line[:20]shorten=Trueelse:print line.rstrip()绘图plot_binning_results.py报错
更新 ${soft}/envs/metawrap/bin/metawrap-scripts/plot_binning_results.py 脚本
# 原脚本存在嵌套错误,会输出报错,修改部分如下
# Traceback (most recent call last):
# File "/anaconda3/envs/metawrap-env/bin/metawrap-scripts/plot_binning_results.py", line 119, in <module>
# plt.text(x_pos, y_pos, bin_set, fontsize=18, color=c)
# NameError: name 'x_pos' is not defined# add bin set label to plotfor x_pos,y in enumerate(data[bin_set]):if y>y_pos:breakplt.text(x_pos, y_pos, bin_set, fontsize=18, color=c)y_pos+=y_increment# add plot and axis titles and adjust the edges
plt.title("Bin contamination ranking", fontsize=26)
plt.xlabel("Acending contamination rank", fontsize=16)
plt.ylabel("Estimated bin contamination (log scale)", fontsize=16)
plt.gcf().subplots_adjust(right=0.9)# save figure
print "Saving figures binning_results.eps and binning_results.png ..."
plt.tight_layout(w_pad=10)
plt.subplots_adjust(top=0.92, right=0.90, left=0.08)
plt.savefig("binning_results.png",format='png', dpi=300)
plt.savefig("binning_results.eps",format='eps')
#plt.show()
EOFblast版本不兼容
更新 metawrap 中的 blast 版本,直接到https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/,下载最新版本blastn,再到conda的metawrap环境bin目录下,替换掉旧版本的blastn
# 如果出现这个错误,BLAST Database error: Error: Not a valid version 4 database.
# 是metawrap 中 blast 版本太老了,需要更新下
wget -c https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.13.0+-x64-linux.tar.gz
tar xvzf ncbi-blast-2.13.0+-x64-linux.tar.gz
mv ncbi-blast-2.13.0+/bin/* ${soft}/envs/metawrap-env/bin/附录
Conda安装小工具
以下小工具已经整合至EasyMicrobiome项目中的linux文件夹,以下代码提供学习多种自主安装的参考方法,用于积累conda使用
并行计算管理rush/paprllel
# conda安装rush,无依赖关系更好用的并行工具
conda install rush -c bioconda
# Ubuntu下安装方法 apt install parallel
# conda安装parallel,版本有点老
conda install parallel -c bioconda
parallel --version # GNU parallel 20170422表格统计工具csvtk和序列处理seqkit(可选中)
# 方法1. conda安装,可能有点旧
conda install csvtk -c bioconda
conda install seqkit -c bioconda# 方法2. 直接下载最新版 https://github.com/shenwei356,如以csvtk为例手动安装
wget -c https://github.com/shenwei356/csvtk/releases/download/v0.22.0/csvtk_linux_amd64.tar.gz
tar xvzf csvtk_linux_amd64.tar.gz
cp csvtk ~/miniconda3/bin/宿主参考基因组下载
- EnsembleGenomes http://ensemblgenomes.org/
- 包括动物、植物、原生生物、真菌、细菌等,此外植物还 Phytozome https://phytozome-next.jgi.doe.gov/ ,以及单个物种和专用数据库
以Ensemble中拟南芥为例:Arabidopsis thaliana -- Genome assembly -- Download DNA sequence (无反应),点TAIR链接跳转ENA,下载All Seq FASTA
wget https://www.ebi.ac.uk/ena/browser/api/fasta/GCA_000001735.1?download=true&gzip=true
mv GCA_000001735.1\?download\=true TAIR10.fa
以Ensemble中水稻为例:Oryza sativa Japonica —— IRGSP-1.0
wget https://www.ebi.ac.uk/ena/browser/api/fasta/GCA_001433935.1?download=true&gzip=true
mv GCA_001433935.1\?download\=true IRGSP1.0.fa
KEGG层级注释整理
己整合至EasyMicrobiome中,自己更新请访问 https://www.kegg.jp/kegg-bin/show_brite?ko00001.keg 下载htext
# 转换ABCD为列表
kegg_ko00001_htext2tsv.pl -i ko00001.keg -o ko00001.tsv
# 统计行数,2021.1月版55761行,整理后为55103个条目
wc -l ko00001.*
# 统计各级数量, /54/527/23917
for i in `seq 1 2 8`;docut -f ${i} ko00001.tsv|sort|uniq|wc -l ; done
# 生成KO编号和注释列表
cut -f 7,8 ko00001.tsv|sort|uniq|sed '1 i KO\tDescription' \> KO_description.txt
# KO与通路(Pathway)对应表,用于合并D级为C级
awk 'BEGIN{FS=OFS="\t"} {print $7,$6}' ko00001.tsv | sed '1 i KO\tpathway' \> KO_path.list毒力因子数据库VFDB
官网:http://www.mgc.ac.cn/VFs/ 数据每周更新
mkdir -p ${db}/vfdb && cd ${db}/vfdb
# 毒力因子描述文件
wget -c http://www.mgc.ac.cn/VFs/Down/VFs.xls.gz
# 核心数据库(1117K)
wget -c http://www.mgc.ac.cn/VFs/Down/VFDB_setA_pro.fas.gz
# 完整数据库(4.99M)
wget -c http://www.mgc.ac.cn/VFs/Down/VFDB_setB_pro.fas.gz
# 解压
gunzip *.gz宏基因组基于读长的分析 HUMAnN2/Metaphlan2/graphlan
HUMAnN2解包安装
# 下载
wget -c ftp://download.nmdc.cn/tools/conda/humann2.tar.gz
# 指定安装目录
mkdir -p ${soft}/envs/humann2
tar -xvzf humann2.tar.gz -C ${soft}/envs/humann2
# 启动环境
conda activate humann2
# 初始化环境
conda unpackHUMAnN2直接安装
# mamba 是快速版本的 conda
mamba create -n humann2 humann2 graphlan export2graphlan -c bioconda -yHUMAnN2安装测试
conda activate humann2
# 记录核心软件版本
humann2 --version # v2.8.1
metaphlan2.py -v # 2.7.5 (6 February 2018)
diamond help | head -n 1 # v0.8.36.98
graphlan.py --version # 1.1.3 (5 June 2018)
export2graphlan.py -h # 0.22 of 05 May# 测试流程是否可用
humann2_testHUMAnN2物种和功能数据库
# 显示可用分类、泛基因组和功能数据库
humann2_databases# 安装数据库(注:数据库下载慢或失败,附录有国内备份链接)
cd ${db}
mkdir -p ${db}/humann2 # 建立下载目录
# 输助比对数据库 593MB
humann2_databases --download utility_mapping full ${db}/humann2
# 微生物泛基因组 5.37 GB
humann2_databases --download chocophlan full ${db}/humann2
# 功能基因diamond索引 10.3 GB
humann2_databases --download uniref uniref90_diamond ${db}/humann2# humann2数据库无法下载:附录备用链接下载后手动配置
mkdir -p ${db}/humann2/chocophlan && cd ${db}/humann2/chocophlan
tar xvzf full_chocophlan_plus_viral.v0.1.1.tar.gz
mkdir -p ${db}/humann2/uniref && cd ${db}/humann2/uniref
tar xvzf uniref90_annotated_1_1.tar.gz
mkdir -p ${db}/humann2/utility_mapping && cd ${db}/humann2/utility_mapping
tar xvzf full_mapping_1_1.tar.gz# 设置数据库位置
# 显示参数
humann2_config --print
# 如修改线程数,推荐3-8,根据实际情况调整
humann2_config --update run_modes threads 4
humann2_config --update database_folders utility_mapping ${db}/humann2/utility_mapping
humann2_config --update database_folders nucleotide ${db}/humann2/chocophlan
humann2_config --update database_folders protein ${db}/humann2/uniref
humann2_config --print## metaphlan2数据库下载和配置
mkdir -p ${db}/humann2 && cd ${db}/humann2
wget -c ftp://download.nmdc.cn/tools/humann2/metaphlan2.tar.gz
tar xvzf metaphlan2.tar.gz
# 链接到软件安装目录
mkdir -p ${soft}/envs/humann2/bin/databases
ln -s ${db}/humann2/metaphlan2/* ${soft}/envs/humann2/bin/databases/n=kneaddata
conda pack -f --ignore-missing-files -n ${n} -o ${n}.tar.gz2、序列处理分析流程
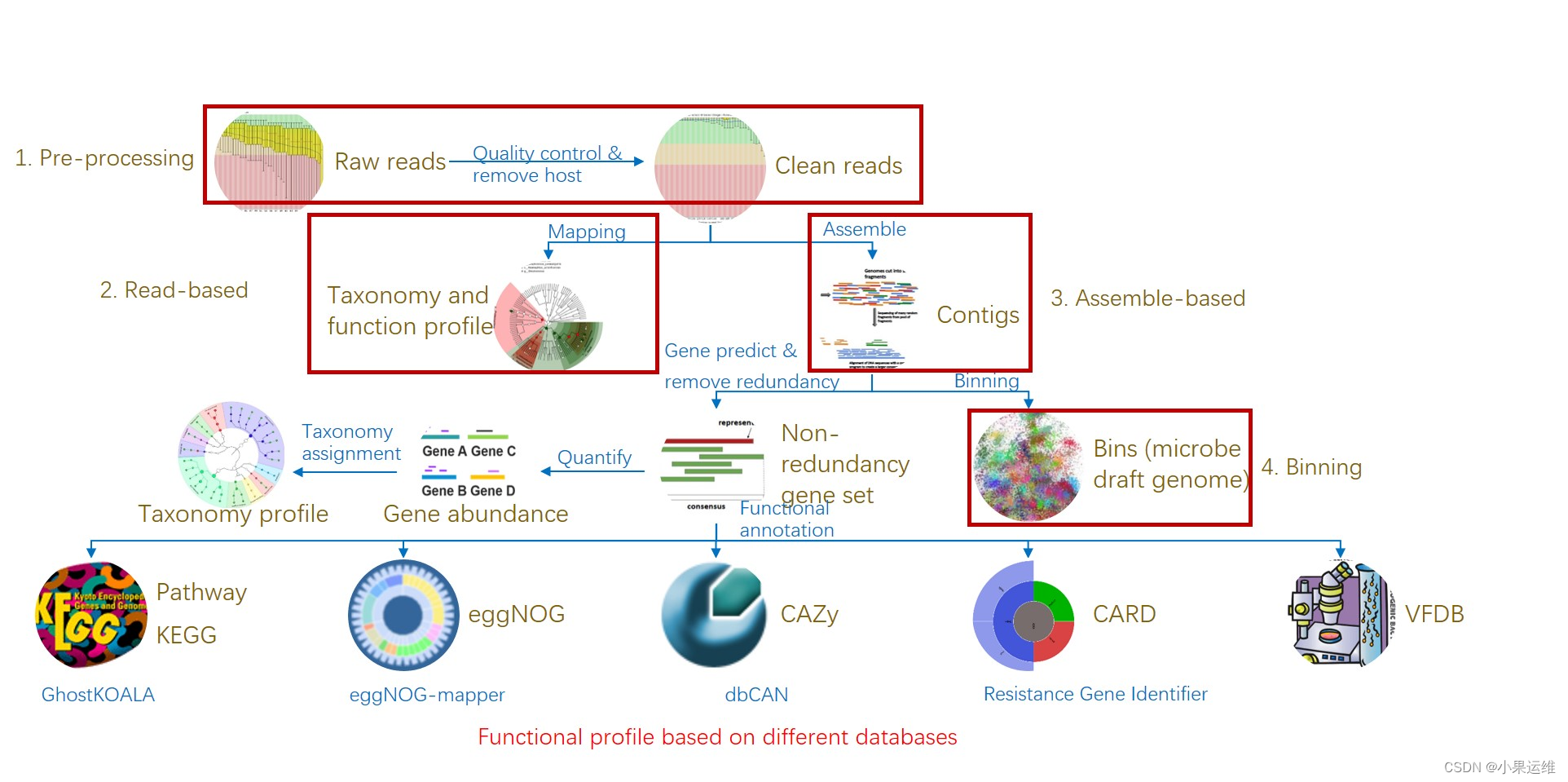
# 易宏基因组流程 EasyMetagenome Pipeline
# 版本Version: 1.20, 2023/11/23
# 操作系统Operation System: Linux Ubuntu 22.04+ / CentOS 7.7+
一、数据预处理 Data preprocessing
1.1 准备工作 Preparing
1. 首次使用请参照`0Install.sh`脚本,安装软件和数据库(大约1-3天,仅一次)
2. 易宏基因组(EasyMetagenome)流程`1Pipeline.sh`复制到项目文件夹,如本次为meta
3. 项目文件夹准备测序数据(seq/*.fq.gz)和样本元数据(result/metadata.txt)
**环境变量设置 Environment variable settings**
**分析前必须运行,设置数据库、软件和工作目录**
# Conda软件安装目录,`conda env list`查看,如/anaconda3
soft=~/miniconda3
# 数据库database(db)位置,如管理员/db,个人~/db
db=~/db
# 设置工作目录work directory(wd),如meta
wd=~/meta
# 创建并进入工作目录
mkdir -p $wd && cd $wd
# 创建3个常用子目录:序列,临时文件和结果
mkdir -p seq temp result
# 添加分析所需的软件、脚本至环境变量,添加至~/.bashrc中自动加载
PATH=$soft/bin:$soft/condabin:/usr/local/sbin:/usr/local/bin:/usr/sbin:/usr/bin:/sbin:/bin:$db/EasyMicrobiome/linux:$db/EasyMicrobiome/script
echo $PATH**元数据和序列文件 Metadata and Sequence Files**
元数据
# 上传元数据metadata.txt至result目录,此处下载并重命名
wget http://www.imeta.science/github/EasyMetagenome/result/metadata.txt
mv metadata.txt result/metadata.txt
# 检查文件格式,^I为制表符,$为Linux换行,^M$为Windows回车,^M为Mac换行符
cat -A result/metadata.txt# 根据样本文件生成元数据,可筛选子集,如EB开头
ls seq/EB*|grep '_1'|cut -f1 -d '_'|cut -f 2 -d '/'|sed'1 i SampleID'>result/metadataEB.txt
cp result/metadataEB.txt result/metadata.txt# 元数据细节优化
# 转换Windows回车为Linux换行
sed -i 's/\r//' result/metadata.txt
# 去除数据中的一个多余空格
sed -i 's/Male /Male/' result/metadata.txt
cat -A result/metadata.txt序列文件
# 用户使用filezilla上传测序文件至seq目录,本次从网络下载
# seq 目录下已经有测试文件,下载跳过
cd seq/
awk '{system("wget -c http://www.imeta.science/github/EasyMetagenome/seq/"$1"_1.fq.gz")}' <(tail -n+2 ../result/metadata.txt)
awk '{system("wget -c http://www.imeta.science/github/EasyMetagenome/seq/"$1"_2.fq.gz")}' <(tail -n+2 ../result/metadata.txt)
cd ..
# ls查看文件大小,-l 列出详细信息 (l: list),-sh 显示人类可读方式文件大小 (s: size; h: human readable)
ls -lsh seq/*.fq.gz序列文件格式检查
zless/zcat查看可压缩文件,检查序列质量格式(质量值大写字母为标准Phred33格式,小写字母为Phred64,需参考附录:质量值转换);检查双端序列ID是否重名,如重名需要改名。参考**附录 —— 质控kneaddata,去宿主后双端不匹配;序列改名**。
# 设置某个样本名为变量i,以后再无需修改
i=C1
# zless查看压缩文件,空格翻页,q退出; head指定显示行数
zless seq/${i}_1.fq.gz | head -n4**工作目录和文件结构总结**# ├── pipeline.sh
# ├── result
# │ └── metadata.txt
# ├── seq
# │ ├── C1_1.fq.gz
# │ ├── ...
# │ └── N1_2.fq.gz
# └── temp* 1pipeline.sh是分析流程代码;
* seq目录中有2个样本Illumina双端测序,4个序列文件;
* temp是临时文件夹,存储分析中间文件,结束可全部删除节约空间
* result是重要节点文件和整理化的分析结果图表,
* 实验设计metadata.txt也在此
1.2 Fastp质量控制 Quality Control
# 创建目录,记录软件版本和引文
mkdir -p temp/qc result/qc
fastp# 单样本质控
i=C1
fastp -i seq/${i}_1.fq.gz -I seq/${i}_2.fq.gz \-o temp/qc/${i}_1.fastq -O temp/qc/${i}_2.fastq# 多样本并行
# -j 2: 表示同时处理2个样本
time tail -n+2 result/metadata.txt|cut -f1|rush -j 2 \"fastp -i seq/{1}_1.fq.gz -I seq/{1}_2.fq.gz \-j temp/qc/{1}_fastp.json -h temp/qc/{1}_fastp.html \-o temp/qc/{1}_1.fastq -O temp/qc/{1}_2.fastq \> temp/qc/{1}.log 2>&1 "# 质控后结果汇总
echo -e "SampleID\tRaw\tClean" > temp/fastp
for i in `tail -n+2 result/metadata.txt|cut -f1`;doecho -e -n "$i\t" >> temp/fastpgrep 'total reads' temp/qc/${i}.log|uniq|cut -f2 -d ':'|tr '\n' '\t' >> temp/fastpecho "" >> temp/fastp
done
sed -i 's/ //g;s/\t$//' temp/fastp
# 按metadata排序
awk 'BEGIN{FS=OFS="\t"}NR==FNR{a[$1]=$0}NR>FNR{print a[$1]}' temp/fastp result/metadata.txt \> result/qc/fastp.txt
cat result/qc/fastp.txt1.3 KneadData去宿主 Host removal
kneaddata是流程主要依赖bowtie2比对宿主,然后筛选非宿主序列用于下游分析。
# 创建目录、启动环境、记录版本
mkdir -p temp/hr
conda activate kneaddata
kneaddata --version # 0.12.0多样品并行去宿主,16p 4h
time tail -n+2 result/metadata.txt|cut -f1|rush -j 2 \"sed '1~4 s/ 1:/.1:/;1~4 s/$/\/1/' temp/qc/{}_1.fastq > /tmp/{}_1.fastq; \sed '1~4 s/ 2:/.1:/;1~4 s/$/\/2/' temp/qc/{}_2.fastq > /tmp/{}_2.fastq; \kneaddata -i1 /tmp/{1}_1.fastq -i2 /tmp/{1}_2.fastq \-o temp/hr --output-prefix {1} \--bypass-trim --bypass-trf --reorder \--bowtie2-options '--very-sensitive --dovetail' \-db ${db}/kneaddata/human/hg37dec_v0.1 \--remove-intermediate-output -v -t 3; \rm /tmp/{}_1.fastq /tmp/{}_2.fastq"# * 匹配任意多个字符,? 匹配任意一个字符
ls -shtr temp/hr/*_paired_?.fastq简化改名
# Ubuntu系统改名
rename 's/paired_//' temp/hr/*.fastq
# CentOS系统改名
rename 'paired_' '' temp/hr/*.fastq大文件清理,高宿主含量样本可节约>90%空间
# 使用命令的绝对路径确保使用无参数的命令,管理员用alias自定义命令含参数,影响操作结果
/bin/rm -rf temp/hr/*contam* temp/hr/*unmatched* temp/hr/reformatted* temp/hr/_temp*
ls -l temp/hr/质控结果汇总
kneaddata_read_count_table --input temp/hr \--output temp/kneaddata.txt
# 筛选重点结果列
cut -f 1,2,5,6 temp/kneaddata.txt | sed 's/_1_kneaddata//' > result/qc/sum.txt
# 对齐方式查看表格
csvtk -t pretty result/qc/sum.txt二、基于读长分析 Read-based (HUMAnN3+MetaPhlAn4+Kraken2)
2.1 准备HUMAnN输入文件
HUMAnN要求双端序列合并的文件作为输入,for循环根据实验设计样本名批量双端序列合并。注意星号(\*)和问号(?),分别代表多个和单个字符。当然大家更不能溜号,行分割的代码行末有一个\\
mkdir -p temp/concat
# 双端合并为单个文件
for i in `tail -n+2 result/metadata.txt|cut -f1`;do cat temp/hr/${i}_?.fastq \> temp/concat/${i}.fq; done
# 查看样品数量和大小
ls -shl temp/concat/*.fq
# 数据太大,计算时间长,可用head对单端分析截取20M序列,即3G,行数为80M行,详见附录:HUMAnN2减少输入文件加速2.2 HUMAnN计算物种和功能组成
* 物种组成调用MetaPhlAn4
* 输入文件:temp/concat/*.fq 每个样品质控后双端合并后的fastq序列
* 输出文件:temp/humann3/ 目录下
* C1_pathabundance.tsv
* C1_pathcoverage.tsv
* C1_genefamilies.tsv
* 整合后的输出:
* result/metaphlan4/taxonomy.tsv 物种丰度表
* result/metaphlan4/taxonomy.spf 物种丰度表(用于stamp分析)
* result/humann3/pathabundance_relab_unstratified.tsv 通路丰度表
* result/humann3/pathabundance_relab_stratified.tsv 通路物种组成丰度表
* stratified(每个菌对此功能通路组成的贡献)和unstratified(功能组成)
启动humann3环境,检查数据库配置
conda activate humann3
# 备选source加载指定环境
# source ~/miniconda3/envs/humann3/bin/activate
mkdir -p temp/humann3
humann --version # v3.7
humann_config单样本1.25M PE150运行测试,8p,2.5M,1\~2h;0.2M, 34m;0.1M,30m;0.01M,25m;16p,18m
i=C1
# 3p,26m; 数据库使用ssd缩短到19m;16p,8m
time humann --input temp/concat/${i}.fq --output temp/humann3 --threads 3 --metaphlan-options '--bowtie2db /db/metaphlan4 --index mpa_vOct22_CHOCOPhlAnSGB_202212 --offline'多样本并行计算,测试数据约30m,推荐16p,3小时/样本
# 如果服务器性能好,请设置--threads值为8/16/32
tail -n+2 result/metadata.txt | cut -f1 | rush -j 2 \"humann --input temp/concat/{1}.fq \--output temp/humann3/ --threads 3 --metaphlan-options '--bowtie2db /db/metaphlan4 --index mpa_vOct22_CHOCOPhlAnSGB_202212 --offline'"# 移动重要文件至humann3目录
# $(cmd) 与 `cmd` 通常是等价的;`cmd`写法更简单,但要注意反引号是键盘左上角ESC下面的按键,$(cmd)更通用,适合嵌套使用
for i in $(tail -n+2 result/metadata.txt | cut -f1); do mv temp/humann3/${i}_humann_temp/${i}_metaphlan_bugs_list.tsv temp/humann3/
done
# 删除临时文件,极占用空间
/bin/rm -rf temp/concat/* temp/humann3/*_humann_temp(可选)单独运行MetaPhlAn4
mkdir -p temp/humann3
i=C1
# 仅物种注释极快4p, 2m, 1m读取数据库
time metaphlan --input_type fastq temp/qc/${i}_1.fastq \temp/humann3/${i}.txt --bowtie2db /db/metaphlan4 --index mpa_vOct22_CHOCOPhlAnSGB_202212 --offline \--nproc 42.3 物种组成表
**样品结果合并**
mkdir -p result/metaphlan4
# 合并、修正样本名、预览
merge_metaphlan_tables.py temp/humann3/*_metaphlan_bugs_list.tsv | \sed 's/_metaphlan_bugs_list//g' | tail -n+2 | sed '1 s/clade_name/ID/' | sed '2i #metaphlan4'> result/metaphlan4/taxonomy.tsv
csvtk -t stat result/metaphlan4/taxonomy.tsv
head -n5 result/metaphlan4/taxonomy.tsv**转换为stamp的spf格式**
# metaphlan4较2增加更多unclassified和重复结果,用sort和uniq去除
metaphlan_to_stamp.pl result/metaphlan4/taxonomy.tsv \|sort -r | uniq > result/metaphlan4/taxonomy.spf
head result/metaphlan4/taxonomy.spf
# STAMP不支持unclassified,需要过滤掉再使用
grep -v 'unclassified' result/metaphlan4/taxonomy.spf > result/metaphlan4/taxonomy2.spf
# 下载metadata.txt和taxonomy2.spf使用stamp分析2.4 功能组成分析
功能组成样本合并合并
mkdir -p result/humann3
humann_join_tables --input temp/humann3 \--file_name pathabundance \--output result/humann3/pathabundance.tsv
# 样本名调整:删除列名多余信息
sed -i 's/_Abundance//g' result/humann3/pathabundance.tsv
# 统计和预览
csvtk -t stat result/humann3/pathabundance.tsv
head -n5 result/humann3/pathabundance.tsv标准化为相对丰度relab(1)或百万比cpm(1,000,000)
humann_renorm_table \--input result/humann3/pathabundance.tsv \--units relab \--output result/humann3/pathabundance_relab.tsv
head -n5 result/humann3/pathabundance_relab.tsv分层结果:功能和对应物种表(stratified)和功能组成表(unstratified)
humann_split_stratified_table \--input result/humann3/pathabundance_relab.tsv \--output result/humann3/ 差异比较和柱状图
两样本无法组间比较,在pcl层面替换为HMP数据进行统计和可视化。
* 输入数据:通路丰度表格 result/humann3/pathabundance.tsv和实验设计 result/metadata.txt
* 中间数据:包含分组信息的通路丰度表格文件 result/humann3/pathabundance.pcl
* 输出结果:result/humann3/associate.txt
在通路丰度中添加分组
## 提取样品列表
head -n1 result/humann3/pathabundance.tsv | sed 's/# Pathway/SampleID/' | tr '\t' '\n' > temp/header
## 对应分组,本示例分组为第2列($2),根据实际情况修改
awk 'BEGIN{FS=OFS="\t"}NR==FNR{a[$1]=$2}NR>FNR{print a[$1]}' result/metadata.txt temp/header | tr '\n' '\t'|sed 's/\t$/\n/' > temp/group
# 合成样本、分组+数据
cat <(head -n1 result/humann3/pathabundance.tsv) temp/group <(tail -n+2 result/humann3/pathabundance.tsv) \> result/humann3/pathabundance.pcl
head -n5 result/humann3/pathabundance.pcl
tail -n5 result/humann3/pathabundance.pcl组间比较,样本量少无差异,结果为4列的文件:通路名字,通路在各个分组的丰度,差异P-value,校正后的Q-value。
演示数据2样本无法统计,此处替换为HMP的结果演示统计和绘图(上传hmp\_pathabund.pcl,替换pathabundance.pcl为hmp\_pathabund.pcl)。
wget -c http://www.imeta.science/github/EasyMetagenome/result/humann2/hmp_pathabund.pcl
/bin/cp -f hmp_pathabund.pcl result/humann3/
# 设置输入文件名
pcl=result/humann3/hmp_pathabund.pcl
# 统计表格行、列数量
csvtk -t stat ${pcl}
head -n3 ${pcl} | cut -f 1-5
# 按分组KW检验,注意第二列的分组列名
humann_associate --input ${pcl} \--focal-metadatum Group --focal-type categorical \--last-metadatum Group --fdr 0.05 \--output result/humann3/associate.txt
wc -l result/humann3/associate.txt
head -n5 result/humann3/associate.txtbarplot展示通路的物种组成,如:腺苷核苷酸合成
# 指定差异通路,如 P163-PWY,--sort sum metadata 按丰度和分组排序
path=P163-PWY
humann_barplot \--input ${pcl} --focal-feature ${path} \--focal-metadata Group --last-metadata Group \--output result/humann3/barplot_${path}.pdf --sort sum metadata KEGG注释
支持GO、PFAM、eggNOG、level4ec、KEGG的D级KO等注释,详见`humann_regroup_table -h`。
# 转换基因家族为KO(uniref90_ko),可选eggNOG(uniref90_eggnog)或酶(uniref90_level4ec)
for i in `tail -n+2 result/metadata.txt|cut -f1`;dohumann_regroup_table \-i temp/humann3/${i}_genefamilies.tsv \-g uniref90_ko \-o temp/humann3/${i}_ko.tsv
done
# 合并,并修正样本名
humann_join_tables \--input temp/humann3/ \--file_name ko \--output result/humann3/ko.tsv
sed -i '1s/_Abundance-RPKs//g' result/humann3/ko.tsv
tail result/humann3/ko.tsv
# 与pathabundance类似,可进行标准化renorm、分层stratified、柱状图barplot等操作# 分层结果:功能和对应物种表(stratified)和功能组成表(unstratified)
humann_split_stratified_table \--input result/humann3/ko.tsv \--output result/humann3/
wc -l result/humann3/ko*# KO合并为高层次L2, L1通路代码KO to level 1/2/3
summarizeAbundance.py \-i result/humann3/ko_unstratified.tsv \-m ${db}/EasyMicrobiome/kegg/KO1-4.txt \-c 2,3,4 -s ',+,+,' -n raw \-o result/humann3/KEGG
wc -l result/humann3/KEGG*2.5 GraPhlAn图
metaphlan2 to graphlan
conda activate humann2
export2graphlan.py --skip_rows 1,2 -i result/metaphlan4/taxonomy.tsv \--tree temp/merged_abundance.tree.txt \--annotation temp/merged_abundance.annot.txt \--most_abundant 1000 --abundance_threshold 20 --least_biomarkers 10 \--annotations 3,4 --external_annotations 7
# 参数说明见PPT,或运行 export2graphlan.py --help
# graphlan annotation
graphlan_annotate.py --annot temp/merged_abundance.annot.txt \temp/merged_abundance.tree.txt temp/merged_abundance.xml
# output PDF figure, annoat and legend
graphlan.py temp/merged_abundance.xml result/metaphlan4/graphlan.pdf \--external_legends
# GraPhlAn Plot(测试中)
graphlan_plot.r --input result/metaphlan4/taxonomy.spf \--design result/metadata.txt --number 100 \--group all --type heatmap \--output result/metaphlan4/heatmap2.6 LEfSe差异分析物种
* 输入文件:物种丰度表result/metaphlan2/taxonomy.tsv
* 输入文件:样品分组信息 result/metadata.txt
* 中间文件:整合后用于LefSe分析的文件 result/metaphlan2/lefse.txt,这个文件可以提供给www\.ehbio.com/ImageGP 用于在线LefSE分析
* LefSe结果输出:result/metaphlan2/目录下lefse开头和feature开头的文件
前面演示数据仅有2个样本,无法进行差异比较。下面使用result12目录中由12个样本生成的结果表进行演示
# 设置结果目录,自己的数据使用result,演示用result12
result=result12
# 如果没有,请下载演示数据
wget -c http://www.imeta.science/db/EasyMetagenome/result12.zip
unzip result12.zip准备输入文件,修改样本品为组名(可手动修改)
# 提取样本行替换为每个样本一行,修改ID为SampleID
head -n1 $result/metaphlan2/taxonomy.tsv|tr '\t' '\n'|sed '1 s/ID/SampleID/' >temp/sampleid
head -n3 temp/sampleid
# 提取SampleID对应的分组Group(假设为metadata.txt中第二列$2),替换换行\n为制表符\t,再把行末制表符\t替换回换行
awk 'BEGIN{OFS=FS="\t"}NR==FNR{a[$1]=$2}NR>FNR{print a[$1]}' $result/metadata.txt temp/sampleid|tr '\n' '\t'|sed 's/\t$/\n/' >groupid
cat groupid
# 合并分组和数据(替换表头)
cat groupid <(tail -n+2 $result/metaphlan2/taxonomy.tsv) > $result/metaphlan2/lefse.txt
head -n3 $result/metaphlan2/lefse.txt方法1. 推荐在线 <https://www.bic.ac.cn/ImageGP/> 中LEfSe一键分析
方法2. LEfSe命令行分析
conda activate lefse
result=result12
# 格式转换为lefse内部格式
lefse-format_input.py $result/metaphlan2/lefse.txt \temp/input.in -c 1 -o 1000000
# 运行lefse(样本必须有重复和分组)
run_lefse.py temp/input.in temp/input.res# 绘制物种树注释差异
lefse-plot_cladogram.py temp/input.res \$result/metaphlan2/lefse_cladogram.pdf --format pdf# 绘制所有差异features柱状图
lefse-plot_res.py temp/input.res \$result/metaphlan2/lefse_res.pdf --format pdf# 绘制单个features柱状图
# 查看显著差异features,按丰度排序
grep -v '-' temp/input.res | sort -k3,3n
# 手动选择指定feature绘图,如Firmicutes
lefse-plot_features.py -f one --format pdf \--feature_name "k__Bacteria.p__Firmicutes" \temp/input.in temp/input.res \$result/metaphlan2/lefse_Firmicutes.pdf# 批量绘制所有差异features柱状图
lefse-plot_features.py -f diff \--archive none --format pdf \temp/input.in temp/input.res \$result/metaphlan2/lefse_2.7 Kraken2+Bracken物种注释和丰度估计
Kraken2可以快速完成读长(read)层面的物种注释和定量,还可以进行重叠群(contig)、基因(gene)、宏基因组组装基因组(MAG/bin)层面的序列物种注释。
# 启动kraken2工作环境
conda activate kraken2
# 记录软件版本
kraken2 --version # 2.1.2
mkdir -p temp/kraken2Kraken2物种注释
输入:temp/qc/{1}_?.fastq 质控后的数据,{1}代表样本名;
参考数据库:-db ${db}/kraken2/pluspfp16g/
输出结果:每个样本单独输出,temp/kraken2/中的{1}_report和{1}_output
整合物种丰度表输出结果:result/kraken2/taxonomy_count.txt
(可选) 单样本注释,5m,50G大数据库较5G库注释比例提高10~20%。以C1为例,在2023/3/14版中,8g: 31.75%; 16g: 52.35%; 150g: 71.98%;同为16g,2023/10/9版本为63.88%
# 根据电脑内存由小到大选择下面3个数据库
# pluspf16g/pluspfp(55G)/plusppfp(120G)
i=C1
time kraken2 --db ${db}/kraken2/pluspf16g/ \--paired temp/qc/${i}_?.fastq \--threads 2 --use-names --report-zero-counts \--report temp/kraken2/${i}.report \--output temp/kraken2/${i}.output多样本并行生成report,1样本8线程逐个运行,内存大但速度快,不建议用多任务并行
for i in `tail -n+2 result/metadata.txt | cut -f1`;dokraken2 --db ${db}/kraken2/pluspf16g \--paired temp/qc/${i}_?.fastq \--threads 2 --use-names --report-zero-counts \--report temp/kraken2/${i}.report \--output temp/kraken2/${i}.output; done使用krakentools转换report为mpa格式
for i in `tail -n+2 result/metadata.txt | cut -f1`;dokreport2mpa.py -r temp/kraken2/${i}.report \--display-header -o temp/kraken2/${i}.mpa; done合并样本为表格
mkdir -p result/kraken2
# 输出结果行数相同,但不一定顺序一致,要重新排序
tail -n+2 result/metadata.txt | cut -f1 | rush -j 1 \'tail -n+2 temp/kraken2/{1}.mpa | LC_ALL=C sort | cut -f 2 | sed "1 s/^/{1}\n/" > temp/kraken2/{1}_count '
# 提取第一样本品行名为表行名
header=`tail -n 1 result/metadata.txt | cut -f 1`
echo $header
tail -n+2 temp/kraken2/${header}.mpa | LC_ALL=C sort | cut -f 1 | \sed "1 s/^/Taxonomy\n/" > temp/kraken2/0header_count
head -n3 temp/kraken2/0header_count
# paste合并样本为表格
ls temp/kraken2/*count
paste temp/kraken2/*count > result/kraken2/tax_count.mpa
# 检查表格及统计
csvtk -t stat result/kraken2/tax_count.mpa
head -n 5 result/kraken2/tax_count.mpaBracken丰度估计
参数简介:
* -d为数据库,-i为输入kraken2报告文件
* r是读长,此处为100,通常为150,o输出重新估计的值
* l为分类级,可选域D、门P、纲C、目O、科F、属G、种S级别丰度估计
* t是阈值,默认为0,越大越可靠,但可用数据越少
循环重新估计每个样品的丰度,请修改tax分别重新计算P和S各1次
# 设置估算的分类级别D,P,C,O,F,G,S,常用门P和种S
# 测序6G起样本-t 10过滤低丰度物种
tax=S
mkdir -p temp/bracken
for i in `tail -n+2 result/metadata.txt | cut -f1`;do# i=C1bracken -d ${db}/kraken2/pluspf16g/ \-i temp/kraken2/${i}.report \-r 100 -l ${tax} -t 0 \-o temp/bracken/${i}.brk \-w temp/bracken/${i}.report; done
# bracken结果合并成表: 需按表头排序,提取第6列reads count,并添加样本名
tail -n+2 result/metadata.txt | cut -f1 | rush -j 1 \'tail -n+2 temp/bracken/{1}.brk | LC_ALL=C sort | cut -f6 | sed "1 s/^/{1}\n/" \> temp/bracken/{1}.count'
# 提取第一样本品行名为表行名
h=`tail -n1 result/metadata.txt|cut -f1`
tail -n+2 temp/bracken/${h}.brk | sort | cut -f1 | \sed "1 s/^/Taxonomy\n/" > temp/bracken/0header.count
# 检查文件数,为n+1
ls temp/bracken/*count | wc
# paste合并样本为表格,并删除非零行
paste temp/bracken/*count > result/kraken2/bracken.${tax}.txt
# 统计行列,默认去除表头
csvtk -t stat result/kraken2/bracken.${tax}.txt
# 按频率过滤,-r可标准化,-e过滤(microbiome_helper)
Rscript ${db}/EasyMicrobiome/script/filter_feature_table.R \-i result/kraken2/bracken.${tax}.txt \-p 0.01 \-o result/kraken2/bracken.${tax}.0.01
csvtk -t stat result/kraken2/bracken.${tax}.0.01个性化结果筛选
# 门水平去除脊索动物(人)
grep 'Chordata' result/kraken2/bracken.P.0.01
grep -v 'Chordata' result/kraken2/bracken.P.0.01 > result/kraken2/bracken.P.0.01-H# 按物种名手动去除宿主污染,以人为例(需按种水平计算相关结果)
# 种水平去除人类P:Chordata,S:Homo sapiens
grep 'Homo sapiens' result/kraken2/bracken.S.0.01
grep -v 'Homo sapiens' result/kraken2/bracken.S.0.01 \> result/kraken2/bracken.S.0.01-H分析后清理每条序列的注释大文件
/bin/rm -rf temp/kraken2/*.output多样性和可视化alpha多样性计算:Berger Parker’s (BP), Simpson’s (Si), inverse Simpson’s (ISi), Shannon’s (Sh)
# Fisher’s (F)依赖scipy.optimize包,默认未安装mkdir -p result/kraken2
echo -e "SampleID\tBerger Parker\tSimpson\tinverse Simpson\tShannon" > result/kraken2/alpha.txt
for i in `tail -n+2 result/metadata.txt|cut -f1`;doecho -e -n "$i\t" >> result/kraken2/alpha.txtfor a in BP Si ISi Sh;doalpha_diversity.py -f temp/bracken/${i}.brk -a $a | cut -f 2 -d ':' | tr '\n' '\t' >> result/kraken2/alpha.txtdoneecho "" >> result/kraken2/alpha.txt
done
cat result/kraken2/alpha.txtbeta多样性计算
beta_diversity.py -i temp/bracken/*.brk --type bracken \> result/kraken2/beta.txt
cat result/kraken2/beta.txtKrona图
for i in `tail -n+2 result/metadata.txt|cut -f1`;dokreport2krona.py -r temp/bracken/${i}.report -o temp/bracken/${i}.krona --no-intermediate-ranksktImportText temp/bracken/${i}.krona -o result/kraken2/krona.${i}.html
donePavian桑基图:https://fbreitwieser.shinyapps.io/pavian/ 在线可视化:,左侧菜单,Upload sample set (temp/bracken/*.report),支持多样本同时上传;Sample查看结果,Configure Sankey配置图样式,Save Network下载图网页
多样性分析/物种组成,详见3StatPlot.sh,Kraken2结果筛选序列见附录
三、组装分析流程 Assemble-based
组装
# 启动工作环境
conda activate megahit### MEGAHIT组装Assembly# 删除旧文件夹,否则megahit无法运行
# /bin/rm -rf temp/megahit
# 组装,10~30m,TB级数据需几天至几周
megahit -t 3 \-1 `tail -n+2 result/metadata.txt|cut -f1|sed 's/^/temp\/hr\//;s/$/_1.fastq/'|tr '\n' ','|sed 's/,$//'` \-2 `tail -n+2 result/metadata.txt|cut -f1|sed 's/^/temp\/hr\//;s/$/_2.fastq/'|tr '\n' ','|sed 's/,$//'` \-o temp/megahit
# 统计大小通常300M~5G,如果contigs太多,可以按长度筛选,降低数据量,提高基因完整度,详见附录megahit
seqkit stat temp/megahit/final.contigs.fa
# 预览重叠群最前6行,前60列字符
head -n6 temp/megahit/final.contigs.fa | cut -c1-60# 备份重要结果
mkdir -p result/megahit/
ln -f temp/megahit/final.contigs.fa result/megahit/
# 删除临时文件
/bin/rm -rf temp/megahit/intermediate_contigs(可选)metaSPAdes精细组装
# 精细但使用内存和时间更多,15~65m
/usr/bin/time -v -o metaspades.py.log metaspades.py -t 3 -m 100 \`tail -n+2 result/metadata.txt|cut -f1|sed 's/^/temp\/qc\//;s/$/_1.fastq/'|sed 's/^/-1 /'| tr '\n' ' '` \`tail -n+2 result/metadata.txt|cut -f1|sed 's/^/temp\/qc\//;s/$/_2.fastq/'|sed 's/^/-2 /'| tr '\n' ' '` \-o temp/metaspades
# 查看软件时间User time和内存Maximum resident set size
cat metaspades.py.log
# 2.3M,contigs体积更大
ls -sh temp/metaspades/contigs.fasta
seqkit stat temp/metaspades/contigs.fasta# 备份重要结果
mkdir -p result/metaspades/
ln -f temp/metaspades/contigs.fasta result/metaspades/
# 删除临时文件
/bin/rm -rf temp/metaspades注:metaSPAdes支持二、三代混合组装,见附录,此外还有OPERA-MS组装二、三代方案
---
QUAST评估
# QUAST评估,生成report文本tsv/txt、网页html、PDF等格式报告
quast.py result/megahit/final.contigs.fa \-o result/megahit/quast -t 2(可选) megahit和metaspades比较
quast.py --label "megahit,metapasdes" \result/megahit/final.contigs.fa \result/metaspades/contigs.fasta \-o result/quast(可选)metaquast评估,更全面,但需下载相关数据库,受网速影响可能时间很长(经常失败)
# metaquast based on silva, and top 50 species genome, 18min
time metaquast.py result/megahit/final.contigs.fa \-o result/megahit/metaquast3.2 基因预测、去冗余和定量Gene prediction, cluster & quantitfy
metaProdigal基因预测Gene prediction
# 输入文件:组装的序列 result/megahit/final.contigs.fa
# 输出文件:prodigal预测的基因序列 temp/prodigal/gene.fa
# 基因大,可参考附录prodigal拆分基因文件,并行计算
mkdir -p temp/prodigal
# prodigal的meta模式预测基因,>和2>&1记录分析过程至gene.log
prodigal -i result/megahit/final.contigs.fa \-d temp/prodigal/gene.fa \-o temp/prodigal/gene.gff \-p meta -f gff > temp/prodigal/gene.log 2>&1
# 查看日志是否运行完成,有无错误
tail temp/prodigal/gene.log
# 统计基因数量
seqkit stat temp/prodigal/gene.fa
# 统计完整基因数量,数据量大可只用完整基因部分
grep -c 'partial=00' temp/prodigal/gene.fa
# 提取完整基因(完整片段获得的基因全为完整,如成环的细菌基因组)
grep 'partial=00' temp/prodigal/gene.fa | cut -f1 -d ' '| sed 's/>//' > temp/prodigal/full_length.id
seqkit grep -f temp/prodigal/full_length.id temp/prodigal/gene.fa > temp/prodigal/full_length.fa
seqkit stat temp/prodigal/full_length.facd-hit基因聚类/去冗余cluster & redundancy
# 输入文件:prodigal预测的基因序列 temp/prodigal/gene.fa
# 输出文件:去冗余后的基因和蛋白序列:result/NR/nucleotide.fa, result/NR/protein.fa
mkdir -p result/NR
# aS覆盖度,c相似度,G局部比对,g最优解,T多线程,M内存0不限制
# 2万基因2m,2千万需要2000h,多线程可加速
cd-hit-est -i temp/prodigal/gene.fa \-o result/NR/nucleotide.fa \-aS 0.9 -c 0.95 -G 0 -g 0 -T 0 -M 0
# 统计非冗余基因数量,单次拼接结果数量下降不大,多批拼接冗余度高
grep -c '>' result/NR/nucleotide.fa
# 翻译核酸为对应蛋白序列, --trim去除结尾的*
seqkit translate --trim result/NR/nucleotide.fa \> result/NR/protein.fa
# 两批数据去冗余使用cd-hit-est-2d加速,见附录salmon基因定量quantitfy
# 输入文件:去冗余后的基因序列:result/NR/nucleotide.fa
# 输出文件:Salmon定量:result/salmon/gene.count, gene.TPM
mkdir -p temp/salmon
salmon -v # 1.8.0# 建索引, -t序列, -i 索引,10s
salmon index -t result/NR/nucleotide.fa \-p 3 -i temp/salmon/index # 定量,l文库类型自动选择,p线程,--meta宏基因组模式, 2个任务并行2个样
tail -n+2 result/metadata.txt | cut -f1 | rush -j 2 \"salmon quant -i temp/salmon/index -l A -p 3 --meta \-1 temp/qc/{1}_1.fastq -2 temp/qc/{1}_2.fastq \-o temp/salmon/{1}.quant"合并
mkdir -p result/salmon
salmon quantmerge --quants temp/salmon/*.quant \-o result/salmon/gene.TPM
salmon quantmerge --quants temp/salmon/*.quant \--column NumReads -o result/salmon/gene.count
sed -i '1 s/.quant//g' result/salmon/gene.*# 预览结果表格
head -n3 result/salmon/gene.*3.3 功能基因注释Functional gene annotation
# 输入数据:上一步预测的蛋白序列 result/NR/protein.fa
# 中间结果:temp/eggnog/protein.emapper.seed_orthologs
# temp/eggnog/output.emapper.annotations
# temp/eggnog/output# COG定量表:result/eggnog/cogtab.count
# result/eggnog/cogtab.count.spf (用于STAMP)# KO定量表:result/eggnog/kotab.count
# result/eggnog/kotab.count.spf (用于STAMP)# CAZy碳水化合物注释和定量:result/dbcan3/cazytab.count
# result/dbcan3/cazytab.count.spf (用于STAMP)# 抗生素抗性:result/resfam/resfam.count
# result/resfam/resfam.count.spf (用于STAMP)# 这部分可以拓展到其它数据库eggNOG基因注释gene annotation(COG/KEGG/CAZy)
软件主页:https://github.com/eggnogdb/eggnog-mapper
# 运行并记录软件版本
conda activate eggnog
emapper.py --version
# emapper-2.1.7 / Expected eggNOG DB version: 5.0.2
# Diamond version found: diamond version 2.0.15# 运行emapper,18m,默认diamond 1e-3
mkdir -p temp/eggnog
time emapper.py --data_dir ${db}/eggnog \-i result/NR/protein.fa --cpu 3 -m diamond --override \-o temp/eggnog/output# 格式化结果并显示表头
grep -v '^##' temp/eggnog/output.emapper.annotations | sed '1 s/^#//' \> temp/eggnog/output
csvtk -t headers -v temp/eggnog/output生成COG/KO/CAZy丰度汇总表
mkdir -p result/eggnog
# 显示帮助
summarizeAbundance.py -h
# 汇总,7列COG_category按字母分隔,12列KEGG_ko和19列CAZy按逗号分隔,原始值累加
summarizeAbundance.py \-i result/salmon/gene.TPM \-m temp/eggnog/output --dropkeycolumn \-c '7,12,19' -s '*+,+,' -n raw \-o result/eggnog/eggnog
sed -i 's#^ko:##' result/eggnog/eggnog.KEGG_ko.raw.txt
sed -i '/^-/d' result/eggnog/eggnog*
head -n3 result/eggnog/eggnog*
# eggnog.CAZy.raw.txt eggnog.COG_category.raw.txt eggnog.KEGG_ko.raw.txt# 添加注释生成STAMP的spf格式
awk 'BEGIN{FS=OFS="\t"} NR==FNR{a[$1]=$2} NR>FNR{print a[$1],$0}' \${db}/EasyMicrobiome/kegg/KO_description.txt \result/eggnog/eggnog.KEGG_ko.raw.txt | \sed 's/^\t/Unannotated\t/' \> result/eggnog/eggnog.KEGG_ko.TPM.spf
head -n 5 result/eggnog/eggnog.KEGG_ko.TPM.spf
# KO to level 1/2/3
summarizeAbundance.py \-i result/eggnog/eggnog.KEGG_ko.raw.txt \-m ${db}/EasyMicrobiome/kegg/KO1-4.txt \-c 2,3,4 -s ',+,+,' -n raw --dropkeycolumn \-o result/eggnog/KEGG
head -n3 result/eggnog/KEGG*# CAZy
awk 'BEGIN{FS=OFS="\t"} NR==FNR{a[$1]=$2} NR>FNR{print a[$1],$0}' \${db}/EasyMicrobiome/dbcan2/CAZy_description.txt result/eggnog/eggnog.CAZy.raw.txt | \sed 's/^\t/Unannotated\t/' > result/eggnog/eggnog.CAZy.TPM.spf
head -n 3 result/eggnog/eggnog.CAZy.TPM.spf# COG
awk 'BEGIN{FS=OFS="\t"} NR==FNR{a[$1]=$2"\t"$3} NR>FNR{print a[$1],$0}' \${db}/EasyMicrobiome/eggnog/COG.anno result/eggnog/eggnog.COG_category.raw.txt > \result/eggnog/eggnog.COG_category.TPM.spf
head -n 3 result/eggnog/eggnog.COG_category.TPM.spf
CAZy碳水化合物酶
# 比对CAZy数据库, 用时2~18m
mkdir -p temp/dbcan3 result/dbcan3
# --sensitive慢10倍,dbcan3e值为1e-102,此处以1e-3演示
time diamond blastp \--db ${db}/dbcan3/CAZyDB \--query result/NR/protein.fa \--threads 2 -e 1e-3 --outfmt 6 --max-target-seqs 1 --quiet \--out temp/dbcan3/gene_diamond.f6
wc -l temp/dbcan3/gene_diamond.f6
# 提取基因与dbcan分类对应表
format_dbcan2list.pl \-i temp/dbcan3/gene_diamond.f6 \-o temp/dbcan3/gene.list
# 按对应表累计丰度,依赖
summarizeAbundance.py \-i result/salmon/gene.TPM \-m temp/dbcan3/gene.list \-c 2 -s ',' -n raw --dropkeycolumn \-o result/dbcan3/TPM
# 添加注释生成STAMP的spf格式,结合metadata.txt进行差异比较
awk 'BEGIN{FS=OFS="\t"} NR==FNR{a[$1]=$2} NR>FNR{print a[$1],$0}' \${db}/EasyMicrobiome/dbcan2/CAZy_description.txt result/dbcan3/TPM.CAZy.raw.txt | \sed 's/^\t/Unannotated\t/' \> result/dbcan3/TPM.CAZy.raw.spf
head result/dbcan3/TPM.CAZy.raw.spf
# 检查未注释数量,有则需要检查原因
grep 'Unannotated' result/dbcan3/TPM.CAZy.raw.spf|wc -lCARD耐药基因
CARD在线分析平台:https://card.mcmaster.ca/
本地软件使用教程: https://github.com/arpcard/rgi
参考文献:http://doi.org/10.1093/nar/gkz935
结果说明:protein.json,在线可视化;protein.txt,注释基因列表
mkdir -p result/card
# 启动rgi环境和记录版本
conda activate rgi6
rgi main -v # 6.0.3# 简化蛋白ID
cut -f 1 -d ' ' result/NR/protein.fa > temp/protein.fa
# 这个错误忽略即可,不是报错,没有任何影响 grep: 写错误: 断开的管道
grep '>' result/NR/protein.fa | head -n 3
grep '>' temp/protein.fa | head -n 3
# 蛋白层面注释ARG
rgi main -i temp/protein.fa -t protein \-n 9 -a DIAMOND --include_loose --clean \-o result/card/protein
head -n3 result/card/protein.txt# 基因层面注释ARG
cut -f 1 -d ' ' result/NR/nucleotide.fa > temp/nucleotide.fa
grep '>' temp/nucleotide.fa | head -n3
rgi main -i temp/nucleotide.fa -t contig \-n 9 -a DIAMOND --include_loose --clean \-o result/card/nucleotide
head -n3 result/card/nucleotide.txt# 重叠群层面注释ARG
cut -f 1 -d ' ' result/megahit/final.contigs.fa > temp/contigs.fa
grep '>' temp/contigs.fa | head -n3
rgi main -i temp/contigs.fa -t contig \-n 9 -a DIAMOND --include_loose --clean \-o result/card/contigs
head result/card/contigs.txt3.4 基因物种注释
# Generate report in default taxid output
conda activate kraken2
kraken2 --db ${db}/kraken2/pluspf16g \result/NR/nucleotide.fa \--threads 3 \--report temp/NRgene.report \--output temp/NRgene.output
# Genes & taxid list
grep '^C' temp/NRgene.output | cut -f 2,3 | sed '1 i Name\ttaxid' \> temp/NRgene.taxid
# Add taxonomy
awk 'BEGIN{FS=OFS="\t"} NR==FNR{a[$1]=$0} NR>FNR{print $1,a[$2]}' \$db/EasyMicrobiome/kraken2/taxonomy.txt \temp/NRgene.taxid > result/NR/nucleotide.tax
conda activate eggnog
summarizeAbundance.py \-i result/salmon/gene.TPM \-m result/NR/nucleotide.tax --dropkeycolumn \-c '2,3,4,5,6,7,8,9' -s ',+,+,+,+,+,+,+,' -n raw \-o result/NR/tax
wc -l result/NR/tax*|sort -n四、分箱挖掘单菌基因组Binning
4.1 MetaWRAP混合样本分箱 Samples binning
主页:https://github.com/bxlab/metaWRAP
教程: https://github.com/bxlab/metaWRAP/blob/master/Usage_tutorial.md
挖掘单菌基因组,需要研究对象复杂度越低、测序深度越大,结果质量越好。要求单样本6GB+,复杂样本如土壤推荐数据量30GB+,至少3个样本
演示数据12个样仅140MB,无法获得单菌基因组,这里使用官方测序数据演示讲解
软件和数据库布置需1-3天,演示数据分析过程超10h,30G样也需1-30天,由服务器性能决定。
# 设置并进入工作目录
wd=~/meta/binning
mkdir -p ${wd} && cd ${wd}
# 初始化项目
mkdir -p temp/hr seq result
# 启动metawrap环境
conda activate metawrap数据和环境变量 Data and enviroment
这里基于质控clean数据和拼接好的重叠群contigs,基于上游结果继续分析。由于上游测试数据过小,分箱无结果。 本次采用软件推荐的7G数据,我们进入一个新文件夹开展分析。
输入输出文件介绍:
# 输入:质控后序列,文件名格式为*_1.fastq和*_2.fastq,temp/qc 目录下,如C1_1.fastq、C1_2.fastq
# 组装的重叠群文件:result/megahit/final.contigs.fa# 输出:
# Binning结果:temp/binning
# 提纯后的Bin统计结果:temp/bin_refinement/metawrap_50_10_bins.stats
# Bin定量结果文件和图:binning/temp/bin_quant/bin_abundance_table.tab 和 bin_abundance_heatmap.png
# Bin物种注释:binning/temp/bin_classify/bin_taxonomy.tab
# Prokka基因预测:binning/temp/bin_annotate/prokka_out/bin.*.ffn 核酸序列
# Bin可视化图表:binning/temp/bloblogy/final.contigs.binned.blobplot (数据表) 和 blobplot_figures (可视化图)准备输入文件:原始数据+组装结果
质控后数据位于temp/qc中,此处需下载并解压
# 方法1. 直接拷贝
/bin/cp -rf /db/metawrap/*.fastq ~/meta/binning/temp/hr/
# 方法2. 在线下载
cd temp/hr
for i in `seq 7 9`;dowget -c ftp.sra.ebi.ac.uk/vol1/fastq/ERR011/ERR01134${i}/ERR01134${i}_1.fastq.gzwget -c ftp.sra.ebi.ac.uk/vol1/fastq/ERR011/ERR01134${i}/ERR01134${i}_2.fastq.gz
done
gunzip -k *.gz
# 批量修改扩展名fq为fastq
# rename .fq .fastq *.fqmegahit拼接结果
cd ${wd}
mkdir -p temp/megahit
cd temp/megahit
# 可从EasyMetagenome目录复制,或链接下载
wget -c http://www.imeta.science/db/metawrap/final.contigs.fa.gz
gunzip -k *.gz
cd ${wd}分箱Binning
# 加载运行环境
cd ${wd}
conda activate metawrap
metawrap -v # 1.3.2# 输入文件为contig和clean reads
# 调用maxbin2, metabat2,8p线程2h,24p耗时1h;-concoct 3h
nohup metawrap binning -o temp/binning \-t 3 -a temp/megahit/final.contigs.fa \--metabat2 --maxbin2 \temp/hr/*.fastq
# --concoct > /dev/null 2>&1 增加3~10倍计算量,添加/dev/null清除海量Warning信息分箱提纯Bin refinement
# 8线程2h, 24p 1.3h;2方法16p 20m
metawrap bin_refinement \-o temp/bin_refinement \-A temp/binning/metabat2_bins/ \-B temp/binning/maxbin2_bins/ \-c 50 -x 10 -t 8
# -C temp/binning/concoct_bins/ \
# 统计高质量Bin的数量,2方法6个,3方法9个
tail -n+2 temp/bin_refinement/metawrap_50_10_bins.stats|wc -l
# 分析比较图见 temp/bin_refinement/figures/所有分箱至同一目录All bins in one directory
mkdir -p temp/drep_in
# 混合组装分箱链接和重命名
ln -s `pwd`/temp/bin_refinement/metawrap_50_10_bins/bin.* temp/drep_in/
ls -l temp/drep_in/
# 改名CentOS
rename 'bin.' 'Mx_All_' temp/drep_in/bin.*
# 改名Ubuntu
rename s/bin./Mx_All_/ temp/drep_in/bin.*
ls temp/drep_in/Mx*(可选Opt)单样本分箱Single sample binning
多样本受硬件、计算时间限制无法完成时,需要单样本组装、分箱。多样本信息丰度,分箱结果更多,更容易降低污染。详见:- [Nature Methods | 单样本与多样本宏基因组分箱的比较揭示了广泛存在的隐藏性污染](https://mp.weixin.qq.com/s/i5C-rCVhZyjRK_Dsk36vBQ)
**设置全局线程、并行任务数和筛选分箱的条件**
# p:threads线程数,job任务数,complete完整度x:contaminate污染率
conda activate metawrap
p=16
j=3
c=50
x=10(可选)并行需要样本列表,请提前编写metadata.txt保存于result中
# 快速读取文件生成样本ID列表再继续编写
ls temp/hr/ | grep _1 | cut -f 1 -d '_' | sort -u | sed '1 i SampleID' > result/metadata.txt
# 预览
cat result/metadata.txt**组装Assemble**
单样本并行组装;支持中断继续运行
tail -n+2 result/metadata.txt|cut -f1|rush -j ${j} \"metawrap assembly -m 200 -t ${p} --megahit \-1 temp/hr/{}_1.fastq -2 temp/hr/{}_2.fastq \-o temp/megahit/{}"**分箱binning**
单样本并行分箱,192p, 15m (concoct使用超多线程);16p 2d/sample, >/dev/null 16p 12h/sample
time tail -n+2 result/metadata.txt|cut -f1|rush -j ${j} \"metawrap binning \-o temp/binning/{} -t ${p} \-a temp/megahit/{}/final_assembly.fasta \--metabat2 --maxbin2 --concoct \temp/hr/{}_*.fastq > /dev/null 2>&1" **分箱提纯bin refinement**
time tail -n+2 result/metadata.txt|cut -f1|rush -j ${j} \"metawrap bin_refinement \-o temp/bin_refinement/{} -t ${p} \-A temp/binning/{}/metabat2_bins/ \-B temp/binning/{}/maxbin2_bins/ \-C temp/binning/{}/concoct_bins/ \-c ${c} -x ${x} "
# 分别为1,2,2个
tail -n+2 result/metadata.txt|cut -f1|rush -j 1 \"tail -n+2 temp/bin_refinement/{}/metawrap_50_10_bins.stats|wc -l "单样品分箱链接和重命名
for i in `tail -n+2 result/metadata.txt|cut -f1`;doln -s `pwd`/temp/bin_refinement/${i}/metawrap_50_10_bins/bin.* temp/drep_in/# CentOSrename 'bin.' "Sg_${i}_" temp/drep_in/bin.*# Ubunturename "s/bin./Sg_${i}_/" temp/drep_in/bin.*
done
# 删除空白中无效链接
/bin/rm -f temp/drep_in/*\*
# 统计混合和单样本来源数据,10个混,5个单;不同系统结果略有差异
ls temp/drep_in/|cut -f 1 -d '_'|uniq -c
# 统计混合批次/单样本来源
ls temp/drep_in/|cut -f 2 -d '_'|cut -f 1 -d '.' |uniq -c(可选Opt)分组分箱 Subgroup binning
样本>30或数据量>300G在1TB内存胖结点上完成混合组装和分箱可能内存不足、且时间>1周甚至1月,需要对研究相近条件、地点进行分小组,且每组编写一个metadata??.txt。
conda activate metawrap
# 小组ID: A1/A2/A3
g=A1**组装Assemble**:<30个或<300G样本,~12hmetawrap assembly -m 600 -t 32 --megahit \-1 `tail -n+2 result/metadata${g}.txt|cut -f1|sed 's/^/temp\/hr\//;s/$/_1.fastq/'|tr '\n' ','|sed 's/,$//'` \-2 `tail -n+2 result/metadata${g}.txt|cut -f1|sed 's/^/temp\/hr\//;s/$/_2.fastq/'|tr '\n' ','|sed 's/,$//'` \-o temp/megahit_${g}**分箱Binning**,~18h# 链接文件到临时位置
mkdir -p temp/${g}/
for i in `tail -n+2 result/metadata${g}.txt|cut -f1`;doln -s `pwd`/temp/hr/${i}*.fastq temp/${g}/
done
# 按组分箱
metawrap binning -o temp/binning_${g} \-t 32 -a temp/megahit_${g}/final_assembly.fasta \--metabat2 --maxbin2 \temp/${g}/*.fastq**分箱提纯Bin refinement**
metawrap bin_refinement \-o temp/bin_refinement_${g} \-A temp/binning_${g}/metabat2_bins/ \-B temp/binning_${g}/maxbin2_bins/ \-c 50 -x 10 -t 32
# 统计高质量Bin的数量
wc -l temp/bin_refinement_${g}/metawrap_50_10_bins.stats**改名汇总 Rename & merge**mkdir -p temp/drep_in
# 混合组装分箱链接和重命名
ln -s `pwd`/temp/bin_refinement_${g}/metawrap_50_10_bins/bin.* temp/drep_in/
# 改名
rename "s/bin./Gp_${g}_/" temp/drep_in/bin.* # Ubuntu
# rename 'bin.' "Gp_${g}_" temp/drep_in/bin.* # CentOS
# 统计
mkdir -p result/bin
echo -n $g >> result/bin/groupNo.txt
ls temp/drep_in/Gp_${g}_*|wc>> result/bin/groupNo.txt
cat result/bin/groupNo.txt4.2 dRep去冗余种/株基因组集
# 进入虚拟环境drep和工作目录
conda activate drep
cd ${wd}按种水平去冗余:6~40min,15个为10个,8个来自混拼,2个来自单拼mkdir -p temp/drep95
# /bin/rm -rf temp/drep95/data/checkM
time dRep dereplicate temp/drep95/ \-g temp/drep_in/*.fa \-sa 0.95 -nc 0.30 -comp 50 -con 10 -p 5
# 报错日志在temp/drep95/log/cmd_logs中查看,加-d显示更多
ls temp/drep95/dereplicated_genomes/|cut -f 1 -d '_'|sort|uniq -c主要结果temp/drep95中:
* 非冗余基因组集:temp/drep95/dereplicated_genomes/*.fa
* 聚类信息表:temp/drep95/data_tables/Cdb.csv
* 聚类和质量图:temp/drep95/figures/*clustering*
(可选)按株水平99%去冗余,20-30min,本处也为10个
mkdir -p temp/drep99
time dRep dereplicate temp/drep99/ \-g temp/drep_in/*.fa \-sa 0.99 -nc 0.30 -comp 50 -con 10 -p 5
ls -l temp/drep99/dereplicated_genomes/ | grep '.fa' | wc -l4.3 CoverM基因组定量
# 启动环境
conda activate coverm
mkdir -p temp/coverm# (可选)单样本测试, 3min
i=ERR011347
time coverm genome --coupled temp/hr/${i}_1.fastq temp/hr/${i}_2.fastq \--genome-fasta-directory temp/drep95/dereplicated_genomes/ -x fa \-o temp/coverm/${i}.txt
cat temp/coverm/${i}.txt# 并行计算, 4min
tail -n+2 result/metadata.txt|cut -f1|rush -j 2 \"coverm genome --coupled temp/hr/{}_1.fastq temp/hr/{}_2.fastq \--genome-fasta-directory temp/drep95/dereplicated_genomes/ -x fa \-o temp/coverm/{}.txt"# 结果合并
mkdir -p result/coverm
conda activate humann3
sed -i 's/_1.fastq Relative Abundance (%)//' temp/coverm/*.txt
humann_join_tables --input temp/coverm \--file_name txt \--output result/coverm/abundance.tsv# 按组求均值,需要metadata中有3列且每个组有多个样本
Rscript ${db}/EasyMicrobiome/script/otu_mean.R --input result/coverm/abundance.tsv \--metadata result/metadata.txt \--group Group --thre 0 \--scale TRUE --zoom 100 --all TRUE --type mean \--output result/coverm/group_mean.txt
# https://www.bic.ac.cn/ImageGP/ 直接选择热图可视化4.4 GTDB-tk物种注释和进化树
启动软件所在虚拟环境
conda activate gtdbtk2.3
export GTDBTK_DATA_PATH="${db}/gtdb"
gtdbtk -v # 2.3.2代表性细菌基因组物种注释
mkdir -p temp/gtdb_classify
# 10个基因组,24p,100min 152G内存; 6p, 22基因组,1h
gtdbtk classify_wf \--genome_dir temp/drep95/dereplicated_genomes \--out_dir temp/gtdb_classify \--extension fa --skip_ani_screen \--prefix tax \--cpus 6
# less -S按行查看,按q退出
less -S temp/gtdb_classify/tax.bac120.summary.tsv
less -S temp/gtdb_classify/tax.ar53.summary.tsv
代表种注释:以上面鉴定的10个种为例,注意扩展名要与输入文件一致,可使用压缩格式gz。主要结果文件描述:此9个细菌基因组在tax.bac120.summary.tsv。古菌在tax.ar53开头的文件中。
(可选)所有MAG物种注释
mkdir -p temp/gtdb_all
# 10000个基因组,32p,100min
time gtdbtk classify_wf \--genome_dir temp/drep_in/ \--out_dir temp/gtdb_all \--extension fa --skip_ani_screen \--prefix tax \--cpus 6
less -S temp/gtdb_all/tax.bac120.summary.tsv
less -S temp/gtdb_all/tax.ar53.summary.tsv
多序列对齐结果建树
# 以9个细菌基因组的120个单拷贝基因建树,1s
mkdir -p temp/gtdb_infer
gtdbtk infer --msa_file temp/gtdb_classify/align/tax.bac120.user_msa.fasta.gz \--out_dir temp/gtdb_infer --prefix tax --cpus 3树文件`tax.unrooted.tree`可使用iTOL在线美化,也可使用GraphLan本地美化。
制作树注释文件:以gtdb-tk物种注释(tax.bac120.summary.tsv)和drep基因组评估(Widb.csv)信息为注释信息
mkdir -p result/itol
# 制作分类学表
tail -n+2 temp/gtdb_classify/tax.bac120.summary.tsv|cut -f 1-2|sed 's/;/\t/g'|sed '1 s/^/ID\tDomain\tPhylum\tClass\tOrder\tFamily\tGenus\tSpecies\n/' \> result/itol/tax.txt
head result/itol/tax.txt
# 基因组评估信息
sed 's/,/\t/g;s/.fa//' temp/drep95/data_tables/Widb.csv|cut -f 1-7,11|sed '1 s/genome/ID/' \> result/itol/genome.txt
head result/itol/genome.txt
# 整合注释文件
awk 'BEGIN{OFS=FS="\t"} NR==FNR{a[$1]=$0} NR>FNR{print $0,a[$1]}' result/itol/genome.txt result/itol/tax.txt|cut -f 1-8,10- > result/itol/annotation.txt
head result/itol/annotation.txt
# 添加各样本相对丰度(各组替换均值)
awk 'BEGIN{OFS=FS="\t"} NR==FNR{a[$1]=$0} NR>FNR{print $0,a[$1]}' <(sed '1 s/Genome/ID/' result/coverm/abundance.tsv) result/itol/annotation.txt|cut -f 1-15,17- > result/itol/annotation2.txt
head result/itol/annotation2.txt CheckM2重新评估
conda activate checkm2
mkdir -p temp/checkm2 result/checkm2
# 10 genomes, 2m
time checkm2 predict --input temp/drep95/dereplicated_genomes/* --output-directory temp/checkm2 --threads 8
ln temp/checkm2/quality_report.tsv result/checkm2/
# 查看结果
less result/checkm2//quality_report.tsv 五、(可选)单菌基因组
5.1 Fastp质量控制
# 每个样本~30s,173个j2共
mkdir -p temp/qc/
time tail -n+2 result/metadata.txt | cut -f1 | rush -j 2 \"time fastp -i seq/{1}_1.fq.gz -I seq/{1}_2.fq.gz \-j temp/qc/{1}_fastp.json -h temp/qc/{1}_fastp.html \-o temp/qc/{1}_1.fastq -O temp/qc/{1}_2.fastq \> temp/qc/{1}.log 2>&1"5.2 metaspades组装
conda activate megahit
spades.py -v # v3.15.4,>3.14.0才支持--isolate模式
mkdir -p temp/spades result/spades
# 127 genoms, 1m17s
time tail -n+2 result/metadata.txt|cut -f1|rush -j 3 \
"spades.py --pe1-1 temp/qc/{1}_1.fastq \--pe1-2 temp/qc/{1}_2.fastq \-t 16 --isolate --cov-cutoff auto \-o temp/spades/{1}" # 筛选>1k的序列并汇总、统计
time tail -n+2 result/metadata.txt|cut -f1|rush -j 3 \"seqkit seq -m 1000 temp/spades/{1}/contigs.fasta \> temp/spades/{1}.fa"
seqkit stat temp/spades/*.fa | sed 's/temp\/spades\///;s/.fa//' > result/spades/stat1k.txt5.3 checkm质量评估
checkm评估质量conda activate drep
checkm # CheckM v1.1.2
mkdir -p temp/checkm result/checkm
# 127 genoms, 1m17s
time checkm lineage_wf -t 8 -x fa temp/spades/ temp/checkm
# format checkm jason to tab
checkmJason2tsv.R -i temp/checkm/storage/bin_stats_ext.tsv \-o temp/checkm/bin_stats.txt
csvtk -t pretty temp/checkm/bin_stats.txt | less(可选)checkm2评估(测试中...)
conda activate checkm2
mkdir -p temp/checkm2
time checkm2 predict --threads 8 --input temp/spades/ --output-directory temp/checkm2筛选污染和高质量基因组 >5% contamination and high quailty
awk '$5<90 || $10>5' temp/checkm/bin_stats.txt | csvtk -t cut -f 1,5,10,4,9,2 > temp/checkm/contamination5.txt
tail -n+2 temp/checkm/contamination5.txt|wc -l
# 筛选高质量用于下游分析 <5% high-quality for down-stream analysis
awk '$5>=90 && $10<=5' temp/checkm/bin_stats.txt | csvtk -t cut -f 1,5,10,4,9,2 | sed '1 i ID\tCompleteness\tContamination\tGC\tN50\tsize' > result/checkm/Comp90Cont5.txt
tail -n+2 result/checkm/Comp90Cont5.txt|wc -l
# 链接高质量基因组至新目录,单菌完整度通常>99%
mkdir -p temp/drep_in/
for n in `tail -n+2 result/checkm/Comp90Cont5.txt|cut -f 1`;doln temp/spades/${n}.fa temp/drep_in/
done5.4 混菌metawarp分箱
分箱和提纯binning & refinement
conda activate metawrap
mkdir -p temp/binning temp/bin
time tail -n+2 temp/checkm/contamination5.txt|cut -f1|rush -j 3 \"metawrap binning \-o temp/binning/{} -t 8 \-a temp/spades/{}/contigs.fasta \--metabat2 --maxbin2 \temp/qc/{}_*.fastq"
time tail -n+2 temp/checkm/contamination5.txt|cut -f1|rush -j 15 \"metawrap bin_refinement \-o temp/bin/{} -t 8 \-A temp/binning/{}/metabat2_bins/ \-B temp/binning/{}/maxbin2_bins/ \-c 50 -x 10"分箱结果汇总
echo -n -e "" > temp/bin/metawrap.stat
for m in `tail -n+2 temp/checkm/contamination5.txt|cut -f1`;doecho ${m} >> temp/bin/metawrap.statcut -f1-4,6-7 temp/bin/${m}/metawrap_50_10_bins.stats >> temp/bin/metawrap.stat
done
# 分箱后的按b1,b2,b3重命名共培养,单菌也可能减少污染
for m in `tail -n+2 temp/checkm/contamination5.txt|cut -f1`;doc=1for n in `tail -n+2 temp/bin/$m/metawrap_50_10_bins.stats|cut -f 1`;docp temp/bin/$m/metawrap_50_10_bins/${n}.fa temp/drep_in/${m}b${c}.fa((c++))
done
done分箱前后统计比较
# 如107个测序分箱为352个基因组,共418个基因组
tail -n+2 temp/checkm/contamination5.txt|wc -l
ls temp/drep_in/*b?.fa | wc -l
ls temp/drep_in/*.fa | wc -l
# 重建新ID列表,A代表所有,B代表Bin分箱过的单菌
ls temp/drep_in/*.fa|cut -f 3 -d '/'|sed 's/.fa//'|sed '1 i ID'|less -S>result/metadataA.txt
ls temp/drep_in/*b?.fa|cut -f 3 -d '/'|sed 's/.fa//'|sed '1 i ID'|less -S>result/metadataB.txt可视化混菌中覆盖度分布,以第一污染菌为例
i=`tail -n+2 temp/checkm/contamination5.txt|head -n1|cut -f1`
# grep '>' temp/spades/${i}.fa|cut -f 2,4,6 -d '_'|sed 's/^/C/;s/_/\t/g'|sed '1i Contig\tLength\tCoverage'>temp/spades/${i}.len
grep '>' temp/drep_in/${i}*|cut -f 3 -d '/'|sed 's/.fa:>NODE//'|cut -f 1,2,4,6 -d '_'|sed 's/_/\t/g'|sed '1i Genome\tContig\tLength\tvalue' > temp/drep_in/${i}.cov
sp_lines.sh -f temp/drep_in/${i}.cov -m TRUE -A TRUE -a Contig -H Genome5.5 drep基因组去冗余
mkdir -p temp/drep95/
conda activate drep
# 相似度sa 0.99995 去重复, 0.99 株水平, 0.95 种水平
dRep dereplicate \-g temp/drep_in/*.fa \-sa 0.95 -nc 0.3 -p 16 -comp 50 -con 10 \temp/drep95
# 统计总和使用基因组数据,确保没有异常丢弃stat total and used genomes, keep no abnormal discard
ls temp/drep_in/*.fa|wc -l
grep 'passed checkM' temp/drep95/log/logger.log|sed 's/[ ][ ]*/ /g'|cut -f 4 -d ' '
# 去冗余后数量,418变为49种
ls temp/drep95/dereplicated_genomes/|wc -l
# 唯一和重复的基因组unique and duplicate genome
csvtk cut -f 11 temp/drep95/data_tables/Widb.csv | sort | uniq -c
# 整理种列表
echo "SampleID" > result/metadataS.txt
ls temp/drep95/dereplicated_genomes/|sed 's/\.fa//' >> result/metadataS.txt5.6 gtdb物种注释
conda activate gtdbtk2.3# 所有基因组注释,400g, 1h, 1T
mkdir -p temp/gtdb_all result/gtdb_all
memusg -t gtdbtk classify_wf \--genome_dir temp/drep_in/ \--out_dir temp/gtdb_all/ \--extension fa --skip_ani_screen \--prefix tax \--cpus 16
# 基因组信息genomeInfo.csv
sed 's/,/\t/g;s/.fa//' temp/drep95/data_tables/genomeInfo.csv |sed '1 s/genome/ID/' > result/gtdb_all/genome.txt# 95%聚类种基因组注释,40g, 1h, 500G
mkdir -p temp/gtdb_95 result/gtdb_95
# Taxonomy classify
memusg -t gtdbtk classify_wf \--genome_dir temp/drep95/dereplicated_genomes/ \--out_dir temp/gtdb_95 \--extension fa --skip_ani_screen \--prefix tax \--cpus 8
# Phylogenetic tree infer
memusg -t gtdbtk infer \--msa_file temp/gtdb_95/align/tax.bac120.user_msa.fasta.gz \--out_dir temp/gtdb_95 \--cpus 8 --prefix g >> temp/gtdb_95/infer.log 2>&1
ln `pwd`/temp/gtdb_95/infer/intermediate_results/g.unrooted.tree result/gtdb_95/# 细菌format to standard 7 levels taxonomy
tail -n+2 temp/gtdb_95/classify/tax.bac120.summary.tsv|cut -f 1-2|sed 's/;/\t/g'|sed '1 s/^/ID\tKingdom\tPhylum\tClass\tOrder\tFamily\tGenus\tSpecies\n/' > result/gtdb_95/tax.bac.txt
# 古菌(可选)
tail -n+2 temp/gtdb_95/classify/tax.ar122.summary.tsv|cut -f 1-2|sed 's/;/\t/g'|sed '1 s/^/ID\tKingdom\tPhylum\tClass\tOrder\tFamily\tGenus\tSpecies\n/' > result/gtdb_95/tax.ar.txt
cat result/gtdb_95/tax.bac.txt <(tail -n+2 result/gtdb_95/tax.ar.txt) > result/gtdb_95/tax.txt# Widb.csv 非冗余基因组信息
sed 's/,/\t/g;s/.fa//' temp/drep95/data_tables/Widb.csv|cut -f 1-7,11|sed '1 s/genome/ID/' > result/gtdb_95/genome.txt
# 整合物种注释和基因组信息 Integrated taxonomy and genomic info
awk 'BEGIN{OFS=FS="\t"} NR==FNR{a[$1]=$0} NR>FNR{print $0,a[$1]}' result/gtdb_95/genome.txt result/gtdb_95/tax.txt|cut -f 1-8,10- > result/gtdb_95/annotation.txt
# csvtk -t headers -v result/gtdb_95/annotation.txt# 制作itol files
cd result/gtdb_95
table2itol.R -D plan1 -a -c double -i ID -l Genus -t %s -w 0.5 annotation.txt
table2itol.R -D plan2 -a -d -c none -b Phylum -i ID -l Genus -t %s -w 0.5 annotation.txt
table2itol.R -D plan3 -c keep -i ID -t %s annotation.txt
table2itol.R -D plan4 -a -c factor -i ID -l Genus -t %s -w 0 annotation.txt
# Stat each level
echo -e 'Taxonomy\tKnown\tNew' > tax.stat
for i in `seq 2 8`;dohead -n1 tax.txt|cut -f ${i}|tr '\n' '\t' >> tax.stattail -n+2 tax.txt|cut -f ${i}|grep -v '__$'|sort|uniq -c|wc -l|tr '\n' '\t' >> tax.stattail -n+2 tax.txt|cut -f ${i}|grep '__$'|wc -l >> tax.stat; done
cat tax.stat
tail -n+2 tax.txt|cut -f3|sort|uniq -c|awk '{print $2"\t"$1}'|sort -k2,2nr > count.phylum
cat count.phylum
cd ../..5.7 功能注释eggnog/dbcan/arg/antismash
基因注释
mkdir -p temp/prodigal
conda activate eggnog
prodigal -v # V2.6.3
# 50g, 31s, 4m
time tail -n+2 result/metadataS.txt|cut -f1|rush -j 10 \
"prodigal \-i temp/drep95/dereplicated_genomes/{1}.fa \-o temp/prodigal/{1}.gff \-a temp/prodigal/{1}.faa \-d temp/prodigal/{1}.ffn \-p single -f gff"
seqkit stat temp/prodigal/*.ffn | sed 's/temp\/prodigal\///;s/\.ffn//;s/[[:blank:]]\{1,\}/\t/g' | cut -f 1,3- \> result/prodigal.txt碳水化合物注释
mkdir -p temp/dbcan3 result/dbcan3
time tail -n+2 result/metadataS.txt|cut -f1|rush -j 9 \
"diamond blastp \--db ${db}/dbcan3/CAZyDB \--query temp/prodigal/{1}.faa \--outfmt 6 --threads 8 --quiet --log \--evalue 1e-102 --max-target-seqs 1 --sensitive \--block-size 6 --index-chunks 1 \--out temp/dbcan3/{1}_diamond.f6"
wc -l temp/dbcan3/*.f6|head -n-1|awk '{print $2"\t"$1}'|cut -f3 -d '/'|sed 's/_diamond.f6//'|sed '1 i ID\tCAZy'|less -S > result/dbcan3/gene.count
# format blast2genelist
for i in `tail -n+2 result/metadataS.txt|cut -f1`;do
format_dbcan3list.pl \-i temp/dbcan3/${i}_diamond.f6 \-o temp/dbcan3/${i}.list
done
# CAZy type count
for i in `tail -n+2 result/metadataS.txt|cut -f1`;dotail -n+2 temp/dbcan3/${i}.list|cut -f2|sort|uniq -c|awk '{print $2"\t"$1}'|sed "1 i CAZy\t${i}"|less -S > temp/dbcan3/${i}_CAZy.tsv
done
# merge2table
conda activate humann3
humann_join_tables \--input temp/dbcan3/ --file_name CAZy \--output result/dbcan3/cazy.txt
csvtk -t stat result/dbcan3/cazy.txt
# merge to level1
paste <(cut -f1 result/dbcan3/cazy.txt) <(cut -f1 result/dbcan3/cazy.txt|tr '0-9' ' '|sed 's/ //g') | sed '1 s/\tCAZy/\tLevel1/' > result/dbcan3/cazy.L1
summarizeAbundance.py \-i result/dbcan3/cazy.txt \-m result/dbcan3/cazy.L1 \-c 2 -s ',' -n raw --dropkeycolumn \-o result/dbcan3/sum
# 基因相似度
echo -e 'Name\tCAZy\tIdentity\tGenome' > result/dbcan3/identity.txt
for i in `tail -n+2 result/metadataS.txt|cut -f1`;docsvtk -t replace -f 2 -p "\d+" -r "" temp/dbcan3/${i}.list | uniq | tail -n+2 | sed "s/$/\t${i}/" >> result/dbcan3/identity.txt
done
csvtk -t stat result/dbcan3/identity.txt
sp_boxplot.sh -f result/dbcan3/identity.txt -m T -F CAZy -d Identity耐药基因
mkdir -p temp/card result/card
conda activate rgi6
# load database 加载数据库
rgi load -i ${db}/card/card.json \--card_annotation ${db}/card/card.fasta --local
# Annotation 蛋白注释
# 默认为0, --include_loose 可极大增加结果,519/4657=11.14%; --exclude_nudge结果不变,但jason为空
time for i in `tail -n+2 result/metadataS.txt|cut -f1`;do
# i=X004b2
cut -f 1 -d ' ' temp/prodigal/${i}.faa | sed 's/\*//' > temp/prodigal/protein_${i}.fa
rgi main \--input_sequence temp/prodigal/protein_${i}.fa \--output_file temp/card/${i} \--input_type protein --clean \--num_threads 8 --alignment_tool DIAMOND > temp/log 2>&1
done# 附录:常见分析问题和补充代码
## 计算时间统计表
在60核(p)及以上服务器,单样本3个并行推荐16p,混合组装分箱推荐32p。
小数据:2个7.5M样本,在72个核、512GB服务器上测试。
大数据:20个10G样本,在192个核、2TB大内存服务器上测试。
| 步骤 | 数据小(6Gx3) | 数据(10Gx20) | 备注 |
| --- | --- | --- | --- |
| fastqc | 33s | 15m | |
| seqkit | 2s | 15m | |
| fastp | 2s | 40m | |
| kneaddata | 25s | 5h | |
| humann | 34m | 30h | |
| megahit | 39s | 15h | |
| binning | 6h | 16h | -concoct |
| binrefine | 2h | 3h | |
| coverm | 4m | 30m | |
注:m为分,h为小时,d为天
## 补充代码Supplementary scripts
**for循环批量处理样本列表**
# 基于样本元数据提取样本列表命令解析
# 去掉表头
tail -n+2 result/metadata.txt
# 提取第一列样本名
tail -n+2 result/metadata.txt|cut -f1
# 循环处理样本
for i in `tail -n+2 result/metadata.txt|cut -f1`;do echo "Processing "$i; done
# ` 反引号为键盘左上角Esc键下面的按键,一般在数字1的左边,代表运行命令返回结果
## 质控去宿主KneadData
**双端序列质控后是否配对的检查**
双端序列质控后序列数量不一致是肯定出错了。但即使序列数量一致,也可能序列不对。在运行metawrap分箱时会报错。可以kneaddata运行时添加--reorder来尝试解决。以下提供了检查双端序列ID是否配对的比较代码
# 文件
i=C1
seqkit seq -n -i temp/qc/${i}_1_kneaddata_paired_1.fastq|cut -f 1 -d '/' | head > temp/header_${i}_1
seqkit seq -n -i temp/qc/${i}_1_kneaddata_paired_2.fastq|cut -f 1 -d '/' | head > temp/header_${i}_2
cmp temp/header_${i}_?
如果序列双端名称一致,且单样本质控结果异常时使用,适合旧版本:新版全kneaddata 1.12已经将此功能添加至流程,以下代码运行返倒引起错误
序列改名,解决NCBI SRA数据双端ID重名问题,详见[《MPB:随机宏基因组测序数据质量控制和去宿主的分析流程和常见问题》](https://mp.weixin.qq.com/s/ovL4TwalqZvwx5qWb5fsYA)。
# 以0.12.0为例,序列中间不一至存在:1和:2会无结果,
@A01909:80:HFT7YDSX5:2:1101:1289:1000 1:N:0:ATGAATAT+TTAAGTGG
@A01909:80:HFT7YDSX5:2:1101:1289:1000 2:N:0:ATGAATAT+TTAAGTGG
# 以0.12.0为例,最终的结果为无空格,结果/1和/2
@A01909:80:HFT7YDSX5:2:1101:1289:1000.1:N:0:ATGAATAT+TTAAGTGG/1
@A01909:80:HFT7YDSX5:2:1101:1289:1000.1:N:0:ATGAATAT+TTAAGTGG/2
# 以单个样本修改
i=A01
sed -i '1~4 s/ 1:/.1:/;1~4 s/$/\/1/' temp/qc/${i}_1.fastq
sed -i '1~4 s/ 2:/.1:/;1~4 s/$/\/2/' temp/qc/${i}_2.fastq
# 批量修改
sed -i '1~4 s/ 1:/.1:/;1~4 s/$/\/1/' temp/qc/${i}_1.fastq
sed -i '1~4 s/ 1:/.1:/;1~4 s/$/\/1/' temp/qc/${i}_2.fastq
**Perl环境不匹配**
报错'perl binaries are mismatched'的解决
e=~/miniconda3/envs/meta
PERL5LIB=${e}/lib/5.26.2:${e}/lib/5.26.2/x86_64-linux-thread-multi
**Java环境错误**
出现错误 Unrecognized option: -d64,为版本不匹配——重装Java运行环境解决:
conda install -c cyclus java-jdk
若出现错误 Error message returned from Trimmomatic :
Error: Invalid or corrupt jarfile \~/miniconda3/envs/kneaddata/share/trimmomatic/trimmomatic;找不到程序,修改配置文件指定脚本名称
sed -i 's/trimmomatic\*/trimmomatic.jar/' ~/miniconda3/envs/kneaddata/lib/python3.10/site-packages/kneaddata/config.py
**Python环境不匹配-找不到包module**
ModuleNotFoundError: No module named 'importlib.metadata'
找不到包,一般是环境变量错误,先确定是否正常启动conda环境,没有重复启动 conda activate kneaddata。已启动检测环境变量
echo $PATH
# /public/software/env01/bin:/public/home/liuyongxin/miniconda3/envs/kneaddata/bin:/public/home/liuyongxin/miniconda3/condabin:/usr/local/bin:/usr/bin:/usr/local/sbin:/usr/sbin
确认conda环境是否为第一个路径,此处kneaddata路径前还有更高优先级的目录在前,重设PATH变量,即删除当前conda环境前的所有路径
PATH=/public/home/liuyongxin/miniconda3/envs/kneaddata/bin:/public/home/liuyongxin/miniconda3/condabin:/usr/local/bin:/usr/bin:/usr/local/sbin:/usr/sbin
## HUMANN物种功能定量
**metaphlan_to_stamp.pl**
# 如果出现下面的错误:
# bash: /db/EasyMicrobiome/script/metaphlan_to_stamp.pl: /usr/bin/perl^M: 解释器错误: 没有那个文件或目录
# sed -i 's/\r//' ${db}/EasyMicrobiome/script/*.pl
**GraPhlAn图**
# metaphlan2 to graphlan
export2graphlan.py --skip_rows 1,2 -i result/metaphlan2/taxonomy.tsv \
--tree temp/merged_abundance.tree.txt \
--annotation temp/merged_abundance.annot.txt \
--most_abundant 1000 --abundance_threshold 20 --least_biomarkers 10 \
--annotations 3,4 --external_annotations 7
# 参数说明见PPT,或运行 export2graphlan.py --help
# graphlan annotation
graphlan_annotate.py --annot temp/merged_abundance.annot.txt \
temp/merged_abundance.tree.txt temp/merged_abundance.xml
# output PDF figure, annoat and legend
graphlan.py temp/merged_abundance.xml result/metaphlan2/graphlan.pdf \
--external_legends
**LEfSe差异分析物种**
* 输入文件:物种丰度表result/metaphlan2/taxonomy.tsv
* 输入文件:样品分组信息 result/metadata.txt
* 中间文件:整合后用于LefSe分析的文件 result/metaphlan2/lefse.txt,这个文件可以提供给www\.ehbio.com/ImageGP 用于在线LefSE分析
* LefSe结果输出:result/metaphlan2/目录下lefse开头和feature开头的文件
前面演示数据仅有2个样本,无法进行差异比较。下面使用result12目录中由12个样本生成的结果表进行演示
# 设置结果目录,自己的数据使用result,演示用result12
result=result12
# 如果没有,请下载演示数据
# wget -c http://www.imeta.science/db/EasyMetagenome/result12.zip
# unzip result12.zip
准备输入文件,修改样本品为组名(可手动修改)
# 预览输出数据
head -n3 $result/metaphlan2/taxonomy.tsv
# 提取样本行,替换为每个样本一行,修改ID为SampleID
head -n1 $result/metaphlan2/taxonomy.tsv|tr '\t' '\n'|sed '1 s/ID/SampleID/' >temp/sampleid
head -n3 temp/sampleid
# 提取SampleID对应的分组Group(假设为metadata.txt中第二列$2),替换换行\n为制表符\t,再把行末制表符\t替换回换行
awk 'BEGIN{OFS=FS="\t"}NR==FNR{a[$1]=$2}NR>FNR{print a[$1]}' $result/metadata.txt temp/sampleid|tr '\n' '\t'|sed 's/\t$/\n/' >groupid
cat groupid
# 合并分组和数据(替换表头)
cat groupid <(tail -n+2 $result/metaphlan2/taxonomy.tsv) > $result/metaphlan2/lefse.txt
head -n3 $result/metaphlan2/lefse.txt
方法1. 推荐在线 <https://www.bic.ac.cn/ImageGP/> 中LEfSe一键分析
方法2. LEfSe命令行分析
conda activate lefse
result=result12
# 格式转换为lefse内部格式
lefse-format_input.py $result/metaphlan2/lefse.txt \
temp/input.in -c 1 -o 1000000
# 运行lefse(样本无重复、分组将报错)
run_lefse.py temp/input.in temp/input.res
# 绘制物种树注释差异
lefse-plot_cladogram.py temp/input.res \
$result/metaphlan2/lefse_cladogram.pdf --format pdf
# 绘制所有差异features柱状图
lefse-plot_res.py temp/input.res \
$result/metaphlan2/lefse_res.pdf --format pdf
# 绘制单个features柱状图
# 查看显著差异features,按丰度排序
grep -v '-' temp/input.res | sort -k3,3n
# 手动选择指定feature绘图,如Firmicutes
lefse-plot_features.py -f one --format pdf \
--feature_name "k__Bacteria.p__Firmicutes" \
temp/input.in temp/input.res \
$result/metaphlan2/lefse_Firmicutes.pdf
# 批量绘制所有差异features柱状图
lefse-plot_features.py -f diff \
--archive none --format pdf \
temp/input.in temp/input.res \
$result/metaphlan2/lefse_
**HUMAnN2减少输入文件加速**
HUMAnN2是计算非常耗时的步骤,如果上百个10G+的样本,有时需要几周至几月的分析。以下介绍两种快速完成分析,而且结果变化不大的方法。替换下面for循环为原文中的“双端合并为单个文件”部分代码
方法1. 软件分析不考虑双端信息,只用一端可获得相近结果,且速度提高1倍。链接质控结果左端高质量至合并目录
for i in `tail -n+2 result/metadata.txt|cut -f1`;do
ln -sf `pwd`/temp/hr/${i}_1.fastq temp/concat/${i}.fq
done
方法2. 控制标准样比对时间。测序数据量通常为6~50G,同一样本分析时间可达10h~100h,严重浪费时间而浪费硬盘空间。
可用head对单端分析截取20M序列,即3G,则为80M行
for i in `tail -n+2 result/metadata.txt|cut -f1`;do
head -n80000000 temp/qc/${i}_1_kneaddata_paired_1.fastq > temp/concat/${i}.fq
done
**metaphlan2无法找到数据库**
正常在首次运行时,会自动下载数据库。有时会失败,解决方法:
方法1. 使用软件安装的用户运行一下程序即可下载成功
方法2. 将我们预下载好的数据索引文件,链接到软件安装目录
db=~/db
soft=~/miniconda3
mkdir -p ${soft}/bin/db_v20
ln -s ${db}/metaphlan2/* ${soft}/bin/db_v20/
mkdir -p ${soft}/bin/databases
ln -s ${db}/metaphlan2/* ${soft}/bin/databases/
**CRITICAL ERROR: Can not call software version for bowtie2**
解决问题思路:
查看文件位置是否处在conda环境中:`type bowtie2`。如果不在需要手动设置环境变量的顺序,如果位置正确如在(\~/miniconda2/envs/humann2/bin/bowtie2),请往下看;
检测bowtie2运行情况:`bowtie2 -h`,报错`wd.c: loadable library and perl binaries are mismatched (got handshake key 0xde00080, needed 0xed00080)`。 错误原因为Perl库版本错误,检查Perl库位置:`echo $PERL5LIB`,错误原因没有指向环境,并手动修改perl库位置
# 设置你环境变量位置,最好用绝对路径
e=~/miniconda2/envs/humann2
PERL5LIB=${e}/lib/5.26.2:${e}/lib/5.26.2/x86_64-linux-thread-multi
**metaphlan_hclust_heatmap.py报错AttributeError: Unknown property axisbg**
在网上搜索,axisbg和axis_bgcolor为过时的函数,新版为facecolor,修改为新名称即可 (参考:<https://blog.csdn.net/qq_41185868/article/details/81842971>)
# 定位文件绝对路径
file=`type metaphlan_hclust_heatmap.py|cut -f 2 -d '('|sed 's/)//'`
# 替换函数名称为新版
sed -i 's/axisbg/facecolor/g' $file
**metaphlan2-共有或特有物种网络图**
awk 'BEGIN{OFS=FS="\t"}{if(FNR==1) {for(i=9;i<=NF;i++) a[i]=$i; print "Tax\tGroup"} \
else {for(i=9;i<=NF;i++) if($i>0.05) print "Tax_"FNR, a[i];}}' \
result/metaphlan2/taxonomy.spf > result/metaphlan2/taxonomy_highabundance.tsv
awk 'BEGIN{OFS=FS="\t"}{if(FNR==1) {print "Tax\tGrpcombine";} else a[$1]=a[$1]==""?$2:a[$1]$2;}END{for(i in a) print i,a[i]}' \
result/metaphlan2/taxonomy_highabundance.tsv > result/metaphlan2/taxonomy_group.tsv
cut -f 2 result/metaphlan2/taxonomy_group.tsv | tail -n +2 | sort -u >group
for i in `cat group`; do printf "#%02x%02x%02x\n" $((RANDOM%256)) $((RANDOM%256)) $((RANDOM%256)); done >colorcode
paste group colorcode >group_colorcode
awk 'BEGIN{OFS=FS="\t"}ARGIND==1{a[$1]=$2;}ARGIND==2{if(FNR==1) {print $0, "Grpcombinecolor"} else print $0,a[$2]}' \
group_colorcode result/metaphlan2/taxonomy_group.tsv > result/metaphlan2/taxonomy_group2.tsv
awk 'BEGIN{OFS=FS="\t"}{if(FNR==1) {print "Tax",$1,$2,$3,$4, $5, $6, $7, $8 } else print "Tax_"FNR, $1,$2,$3,$4, $5,$6, $7, $8}' \
result/metaphlan2/taxonomy.spf > result/metaphlan2/taxonomy_anno.tsv
## 生物标志鉴定LEfSe
**lefse-plot_cladogram.py:Unknown property axis_bgcolor**
若出现错误 Unknown property axis\_bgcolor,则修改`lefse-plot_cladogram.py`里的`ax_bgcolor`替换成`facecolor`即可。
# 查看脚本位置,然后使用RStudio或Vim修改
type lefse-plot_cladogram.py
## 物种分类Kraken2
**合并样本为表格combine_mpa.py**
krakentools中combine_mpa.py,需手动安装脚本,且结果还需调整样本名
combine_mpa.py \
-i `tail -n+2 result/metadata.txt|cut -f1|sed 's/^/temp\/kraken2\//;s/$/.mpa/'|tr '\n' ' '` \
-o temp/kraken2/combined_mpa
**序列筛选/去宿主extract_kraken_reads.py**
提取非植物33090和动物(人)33208序列、选择细菌2和古菌2157
mkdir -p temp/kraken2_qc
parallel -j 3 \
"/db/script/extract_kraken_reads.py \
-k temp/kraken2/{1}.output \
-r temp/kraken2/{1}.report \
-1 temp/qc/{1}_1_kneaddata_paired_1.fastq \
-2 temp/qc/{1}_1_kneaddata_paired_2.fastq \
-t 33090 33208 --include-children --exclude \
--max 20000000 --fastq-output \
-o temp/kraken2_qc/{1}_1.fq \
-o2 temp/kraken2_qc/{1}_2.fq" \
::: `tail -n+2 result/metadata.txt|cut -f1`
## 组装Megahit
**序列长度筛选**
megahit默认>200,可选 > 500 / 1000 bp,并统计前后变化;如此处筛选 > 500 bp,序列从15万变为3.5万条,总长度从7M下降到3M
mv temp/megahit/final.contigs.fa temp/megahit/raw.contigs.fa
seqkit seq -m 500 temp/megahit/raw.contigs.fa > temp/megahit/final.contigs.fa
seqkit stat temp/megahit/raw.contigs.fa
seqkit stat temp/megahit/final.contigs.fa
**数据太大导致程序中断**
报错信息:126 - Too many vertices in the unitig graph (8403694648 >= 4294967294), you may increase the kmer size to remove tons
解决方法:需要增加k-mer,如最小k-mer改为29,不行继续增加或将数据分批次组装
添加参数: --k-min 29 --k-max 141 --k-step 20
## 二三代混合组装
**metaspades**
# 3G数据,耗时3h
i=SampleA
time metaspades.py -t 48 -m 500 \
-1 seq/${i}_1.fastq -2 seq/${i}L_2.fastq \
--nanopore seq/${i}.fastq \
-o temp/metaspades_${i}
**二三代混合组装OPERA-MS**
结果卡在第9步polishing,可添加--no-polishing参数跳过此步;短序列只支持成对文件,多个文件需要cat合并
perl ../OPERA-MS.pl \
--short-read1 R1.fastq.gz \
--short-read2 R2.fastq.gz \
--long-read long_read.fastq \
--no-ref-clustering \
--num-processors 32 \
--out-dir RESULTS
**二代组装+三代优化**
perl ~/soft/OPERA-MS/OPERA-MS.pl \
--contig-file temp/megahit/final.contigs.fa \
--short-read1 R1.fastq.gz \
--short-read2 R2.fastq.gz \
--long-read long_read.fastq \
--num-processors 32 \
--no-ref-clustering \
--no-strain-clustering \
--no-polishing \
--out-dir temp/opera
结果可用quast或seqkit stat统计对二代组装的改进效果
## 基因序列prodigal
**序列拆分并行预测基因**
(可选)以上注释大约1小时完成1M个基因的预测。加速可将contigs拆分,并行基因预测后再合并。
# 拆分contigs,按1M条每个文件
n=10000
seqkit split result/megahit/final.contigs.fa -s $n
# 生成拆分文件序列列表
ls result/megahit/final.contigs.fa.split/final.contigs.part_*.fa|cut -f 2 -d '_'|cut -f 1 -d '.' \
> temp/split.list
# 9线程并行基因预测,此步只用单线程且读写强度不大
time parallel -j 9 \
"prodigal -i result/megahit/final.contigs.fa.split/final.contigs.part_{}.fa \
-d temp/gene{}.fa \
-o temp/gene{}.gff -p meta -f gff \
> temp/gene{}.log 2>&1 " \
::: `cat temp/split.list`
# 合并预测基因和gff注释文件
cat temp/gene*.fa > temp/prodigal/gene.fa
cat temp/gene*.gff > temp/prodigal/gene.gff
## 基因去冗余cd-hit
**两批基因合并cd-hit-est-2d**
cd-hit-est-2d 两批次构建非冗余基因集
A和B基因集,分别有M和N个非冗余基因,两批数据合并后用cd-hit-est去冗余,计算量是(M + N) X (M + N -1)
cd-hit-est-2d比较,只有M X N的计算量
# 计算B中特有的基因
cd-hit-est-2d -i A.fa -i2 B.fa -o B.uni.fa \
-aS 0.9 -c 0.95 -G 0 -g 0 \
-T 96 -M 0 -d 0
# 合并为非冗余基因集
cat A.fa B.uni.fa > NR.fa
**cd-hit合并多批基因salmon索引时提示ID重复**
# [error] In FixFasta, two references with the same name but different sequences: k141_2390219_1. We require that all input records have a unique name up to the first whitespace (or user-provided separator) character.
# 错误解决
mv temp/NRgene/gene.fa temp/NRgene/gene.fa.bak
# 15G,2m,4G
seqkit rename temp/NRgene/gene.fa.bak -o temp/NRgene/gene.fa
## 基因定量salmon
**找不到库文件liblzma.so.0**
* 报错信息:error while loading shared libraries: liblzma.so.0
* 问题描述:直接运行salmon报告,显示找不到lib库,
* 解决方法:可使用程序完整路径解决问题,`alias salmon="${soft}/envs/metagenome_env/share/salmon/bin/salmon"`
## 基因功能数据库
**综合功能注释KEGG描述整理**
脚本位于 /db/script 目录,<https://www.kegg.jp/kegg-bin/show_brite?ko00001.keg> 下载htext,即为最新输入文件 ko00001.keg
kegg_ko00001_htext2tsv.pl -i ko00001.keg -o ko00001.tsv
# 原核蛋白数据库(需付费购买)建索引
conda activate eggnog
diamond --version # 2.0.13
cd genes/fasta
# 3m, 2G
memusg -t diamond makedb --in prokaryotes.pep.gz \
--db prokaryotes.pep
# ID转换为KO
zless prokaryotes.dat.gz|cut -f1,2|sed "1i Kgene\tKO">prokaryotes.gene2KO
**抗生素抗性CARD**
# 使用3.1.0和3.1.2均有警告,修改序列名至纯字母数数字也无效
# WARNING 2021-07-08 08:58:00,478 : Exception : <class 'KeyError'> -> '5141' -> Model(1692) missing in database. Please generate new database.
# WARNING 2021-07-08 08:58:00,478 : Exception : <class 'KeyError'> -> '5141' -> Model(1692)
# WARNING 2021-07-08 08:58:00,479 : tetM ---> hsp.bits: 60.8 <class 'float'> ? <class 'str'>
**抗生素抗性ResFam**
数据库:<http://www.dantaslab.org/resfams>
参考文献:<http://doi.org/10.1038/ismej.2014.106>
mkdir -p temp/resfam result/resfam
# 比对至抗生素数据库 1m
time diamond blastp \
--db ${db}/resfam/Resfams-proteins \
--query result/NR/protein.fa \
--threads 9 --outfmt 6 --sensitive \
-e 1e-5 --max-target-seqs 1 --quiet \
--out temp/resfam/gene_diamond.f6
# 提取基因对应抗性基因列表
cut -f 1,2 temp/resfam/gene_diamond.f6 | uniq | \
sed '1 i Name\tResGeneID' > temp/resfam/gene_fam.list
# 统计注释基因的比例, 488/19182=2.5%
wc -l temp/resfam/gene_fam.list result/salmon/gene.count
# 按列表累计丰度
summarizeAbundance.py \
-i result/salmon/gene.TPM \
-m temp/resfam/gene_fam.list \
-c 2 -s ',' -n raw \
-o result/resfam/TPM
# 结果中添加FAM注释,spf格式用于stamp分析
awk 'BEGIN{FS=OFS="\t"} NR==FNR{a[$1]=$4"\t"$3"\t"$2} NR>FNR{print a[$1],$0}' \
${db}/resfam/Resfams-proteins_class.tsv result/resfam/TPM.ResGeneID.raw.txt \
> result/resfam/TPM.ResGeneID.raw.spf
## 分箱MetaWRAP
**Bin refined报错CheckM**
Something went wrong with running CheckM. Exiting...
重新运行1次,错误仍在;
删除concoct输入,尝试提纯
**MetaWRAP分箱注释Bin classify & annotate**
# Taxator-tk对每条contig物种注释,再估计bin整体的物种,11m (用时66 min)
metawrap classify_bins -b temp/bin_refinement/metawrap_50_10_bins \
-o temp/bin_classify -t 3 &
# 注释结果见`temp/bin_classify/bin_taxonomy.tab`
# export LD_LIBRARY_PATH=/conda2/envs/metagenome_env/lib/:${LD_LIBRARY_PATH}
# 这是动态链接库找不到时的一个简单的应急策略
#ln -s /conda2/envs/metagenome_env/lib/libssl.so.1.0.0 .
#ln -s /conda2/envs/metagenome_env/lib/libcrypto.so.1.0.0 .
# 基于prokka基因注释,4m
metaWRAP annotate_bins -o temp/bin_annotate \
-b temp/bin_refinement/metawrap_50_10_bins -t 5
# 每个bin基因注释的gff文件bin_funct_annotations,
# 核酸ffn文件bin_untranslated_genes,
# 蛋白faa文件bin_translated_genes
ls -sh temp/bin_annotate/prokka_out/bin.1/
**分箱定量Bin quantify**
# 耗时3m,系统用时10m,此处可设置线程,但salmon仍调用全部资源
metawrap quant_bins -b temp/bin_refinement/metawrap_50_10_bins -t 4 \
-o temp/bin_quant -a temp/megahit/final.contigs.fa temp/qc/ERR*.fastq
# 文件名字改变
# 结果包括bin丰度热图`temp/bin_quant/bin_abundance_heatmap.png`
# 原始数据位于`temp/bin_quant/bin_abundance_table.tab`
ls -l temp/bin_quant/bin_abundance_heatmap.png
**GTDB的文件名不存在错**
# ERROR: ['BMN5'] are not present in the input list of genome to process,但并无此菌,可能是名称 中存在"-"或".",替换为i
# 修改metadata
sed 's/-/i/;s/\./i/' result/metadatab.txt > result/metadata.txt
# 修改文件名
awk 'BEGIN{OFS=FS="\t"}{system("mv temp/antismash/"$1".fna temp/antismash/"$2".fna")ll }' <(paste result/metadatab.txt result/metadata.txt|tail -n+2)
EasyAmplicon (易扩增子)-扩增子高通量序列分析软件流程及脚本-详细使用方法——来自刘永鑫团队的秘籍-CSDN博客
Microbiome-易扩增子、易宏基因组等分析流程依赖常用软件、脚本文件和数据库注释文件-CSDN博客
















)
)
)