文章目录

Hi,你好。我是茶桁。
上节课,我们讲了多层神经网络的原理,并且明白了,数据量是层级无法超过3层的主要原因。
然后我们用一张图来解释了整个链式求导的过程:
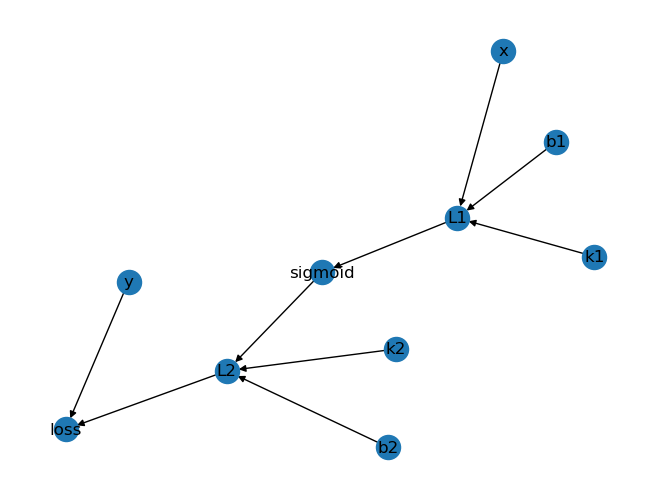
那么,我们如何将这张图里的节点关系来获得它的求导过程呢?
假如我们现在定义一个函数get_output:
def get_output(graph, node):outputs = []for n, links in graph.items():if node == n: outputs += linksreturn outputs
get_output(computing_graph, 'k1')---
['L1']
我们可以根据k1获得l1。
来,让我们整理一下思路,问:如何获得k1的偏导:
- 获得k1的输出节点
- 获得k1输出节点的输出节点
- …直到我们找到最后一个节点
computing_order = []target = 'k1'
out = get_output(computing_graph, target)[0]
computing_order.append(target)while out:computing_order.append(out)out = get_output(computing_graph, out)if out: out = out[0]computing_order---
['k1', 'L1', 'sigmoid', 'L2', 'loss']
我们从k1出发,它可以获得这么一套顺序。那么现在如果要计算k1的偏导,我们的这个偏导顺序就等于从后到前给它求解一遍。
order = ''for i, n in enumerate(computing_order[:-1]):order += '*∂{} / ∂{}'.format(n, computing_order[i+1])order
---
'*∂k1 / ∂L1*∂L1 / ∂sigmoid*∂sigmoid / ∂L2*∂L2 / ∂loss'
现在k1的求导顺序计算机就给它自动求解出来了, 我们把它放到了一个图里面,然后它自动就求解出来了。只不过唯一的问题是现在这个order是反着的,需要把它再反过来。
for i, n in enumerate(computing_order[:-1]):order.append((computing_order[i + 1], n))# order += ' * ∂{} / ∂{}'.format(n, computing_order[i+1])' * '.join(['∂{}/∂{}'.format(a, b) for a, b in order[::-1]])---
'∂loss/∂L2 * ∂L2/∂sigmoid * ∂sigmoid/∂L1 * ∂L1/∂k1'
这个过程用计算机实现之后,我们就可以拿它来看一下其他的参数,比如说b1:
computing_order = []target = 'b1'
out = get_output(computing_graph, target)[0]
computing_order.append(target)while out:computing_order.append(out)out = get_output(computing_graph, out)if out: out = out[0]order = []for i, n in enumerate(computing_order[:-1]):order.append((computing_order[i + 1], n))# order += ' * ∂{} / ∂{}'.format(n, computing_order[i+1])' * '.join(['∂{}/∂{}'.format(a, b) for a, b in order[::-1]])---
'∂loss/∂L2 * ∂L2/∂sigmoid * ∂sigmoid/∂L1 * ∂L1/∂b1'
k2:
...
target = 'k2'
...---
'∂loss/∂L2 * ∂L2/∂k2'
到这里, 我们能够自动的求解各个参数的导数了。
然后我们将其封装一下,然后循环一下每一个参数:
def get_paramter_partial_order(p):...target = p...return ...for p in ['b1', 'k1', 'b2', 'k2']:print(get_paramter_partial_order(p))---
∂loss/∂L2 * ∂L2/∂sigmoid * ∂sigmoid/∂L1 * ∂L1/∂b1
∂loss/∂L2 * ∂L2/∂sigmoid * ∂sigmoid/∂L1 * ∂L1/∂k1
∂loss/∂L2 * ∂L2/∂b2
∂loss/∂L2 * ∂L2/∂k2
到这一步你就能够发现,每一个参数的导数的偏导我们都可以求解了。而且我们还发现一个问题,不管是['b1', 'k1', 'b2', 'k2']中的哪一个,我们都需要求求解∂loss/∂L2。
所以现在如果有一个内存能够记录结果,先把∂loss/∂L2的值求解下来,把这个值先存下来,只要算出来这一个值之后,再算['b1', 'k1', 'b2', 'k2']的时候直接拿过来就行了。
也就是说我们首先需要记录的就是这个值,其次,如果我们把L2和sigmoid的值记下来,求解b1和k1的时候直接拿过来用就行,不需要再去计算一遍,这个时候我们的效率就会提升很多。
首先把共有的一个基础∂loss/∂L2计算了, 第二步,有了∂loss/∂L2,把∂L2/∂sigmoid再记录一遍, 第三个是∂sigmoid/∂L1,然后后面以此就是∂L1/∂b1, ∂L1/∂k1,∂L2/∂b2, ∂L2/∂k2。
现在的问题就是就是怎么样让计算机自动得到这个顺序,计算机得到这个顺序的时候,把这些值都存在某个地方。
这个所谓的顺序就是我们非常重要的一个概念,在计算机科学,算法里面非常重要的一个概念:「拓朴排序」。
那拓朴排序该如何实现呢?来,我们一起来实现一下:
首先,我们定义一个方法,咱们输入的是一个图,这个图的定义方式是一个Dictionary,然后里面有一些节点,里面的很多个连接的点:
def topologic(graph):'''graph: dict{node: [node1, node2, ..., noden]}'''return None
因为我们要把它的结果存在一个变量里边,当我们不断的检查看这个图,看看它是否为空,然后我们来定义两个存储变量:
def topologic(graph):sorted_node = []while graph:all_inputs = []all_outputs = list(graph.keys())return sorted_node
这里的两个变量,all_inputs和all_outputs, 一个是用来存储所有的输入节点,一个是存储所有的输出节点。
我们还记得我们那个图的格式是什么样的吗?
computing_graph = {'k1': ['L1'],'b1': ['L1'],'x': ['L1'],'L1':['sigmoid'],'sigmoid': ['L2'],'k2': ['L2'],'b2': ['L2'],'L2': ['loss'],'y': ['loss']
}
我们看这个数据,那所有的输出节点是不是就是其中的key啊?
打比方说,我们拿一个短小的数据来做示例:
simple_graph = {'a': [1, 2],'b': [3, 4]
}list(simple_graph.keys())---
['a', 'b']
那我们这样就拿到了输出节点,并将其放在了一个列表内。
这里说点其他的,Python 3.9及以上的版本其实都实现了自带拓朴排序,但是如果你的Python版本较低,那还是需要自己去实现。这个也是Python 3.9里面一个比较重要的更新。
那为什么我们的value定义的是一个列表呢?这是因为这个key,也就是输出值可能会输出到好几个函数里面,因为我们现在拿的是一个比较简单的模型,但是在真实场景中,有可能会输出到更多的节点中。
这里,就获得了所有有输入的节点, simple_graph中,a输出给了[1,2], b输出给了[2,3]。
那我们怎么获得所有输入的节点呢?那就应该是value。
list(simple_graph.values())---
[[1, 2], [3, 4]]
这样就获得所有有输入的节点。然后就是怎么样把这两个list合并。可以有一个简单的方法,一个叫做reduce的方法。
reduce(lambda a, b: a+b, list(simple_graph.values()))---
[1, 2, 3, 4]
这样就把它给它连起来了。
那我们还需要找一个,就是只有输出没有输入的节点,这些该怎么去找呢?其实也就是我们的[k1, b1, k2, b2, y]这些值。
来,我们还是拿刚才的simple_graph来举例,但是这次我们改一下里面的内容:
simple_graph = {'a': ['a', 2],'b': ['b', 4]
}a = list(simple_graph.keys())
b = reduce(lambda a, b: a+b, list(simple_graph.values()))print(list(set(b) - set(a)))---
[2, 4]
我们没有用循环,而是将其变成了一个集合,然后利用集合的加减来做。
我们的实际代码就可以这样写:
def topologic(graph):sorted_node = []while graph:all_inputs = reduce(lambda a, b: a+b, list(graph.values()))all_outputs = list(graph.keys())all_inputs = set(all_inputs)all_outputs = set(all_outputs)need_remove = all_outputs - all_inputsreturn sorted_node
那现在我们继续往后,如果找到了这些只有输出没有输入的节点之后,我们做一个判断,然后定义一个节点,用来保存随机选择的节点:
if len(need_remove) > 0:node = random.choice(list(need_remove))
这个时候x, b, k, y都有可能,那么我们随机找一个就行。然后将这个找到的节点从graph给它删除。并且将其插入到sorted_node中去,并且返回出来。
if ...:node = random.choice(list(need_remove))graph.pop(node)sorted_node.append(node)return sorted_node
然后这里还会出一个小问题,我们还是拿一个示例来说:
simple_graph = {'a': ['sigmoid'],'b': ['loss'],'c': ['loss']
}simple_graph.pop('b')
simple_graph---
{'a': ['sigmoid'], 'c': ['loss']}
看,我们在删除node的时候,其所对应的value也就一起删除了,那这个时候,我们最后的输出列表里会丢失最后一个node。所以,我们在判断为最后一个的时候,需要额外的将其加上, 放在pop方法执行之前。那我们整个代码需要调整一下先后顺序。
def topologic(graph):sorted_node = []while graph:...if len(need_remove) > 0:node = random.choice(list(need_remove))sorted_node.append(node)if len(graph) == 1: sorted_node += graph[node] graph.pop(node)return sorted_node
现在其实这个代码就已经OK了,我们来再加几句话:
...
if len(need_remove) > 0:...for _, links in graph.items():if node in links: links.remove(node)
else:raise TypeError('This graph has circle, which cannot get topological order.')
...
我们把它的连接关系,例如现在选择了k1,我们要把k1的连接关系从这些里边给它删掉。
遍历一下graph,遍历的时候如果删除的node在它的输出里边,我们就把它删除。
加上else判断,如果图不是空的,但是最终没有找到,也就是这两个集合作减法,但是得到一个空集,没有找到,那我们就来输出一个错误:This graph has circle, which cannot get topological order.
现在我们可以来实验一下了:
x, k, b, linear, sigmoid, y, loss = 'x', 'k', 'b', 'linear', 'sigmoid', 'y', 'loss'
test_graph = {x: [linear],k: [linear],b: [linear],linear: [sigmoid],sigmoid: [loss],y: [loss]
}topologic(test_graph)---
['x', 'b', 'k', 'y', 'linear', 'sigmoid', 'loss']
好, 现在让我们来声明一个class node:
class Node:pass
然后我们先来抽象一下这些节点:
## Our Simple Model Elementsnode_x = Node(inputs=None, outputs=[node_linear])
node_y = Node(inputs=None, outputs=[node_loss])
node_k = Node(inputs=None, outputs=[node_linear])
node_b = Node(inputs=None, outputs=[node_linear])
node_linear = Node(inputs=[node_x, node_k, node_b], outputs=[node_sigmoid])
node_sigmoid = Node(inputs=[node_linear], outputs=[node_loss])
node_loss = Node(inputs=[node_sigmoid, node_y], outputs=None)
现在咱们就把图中每个节点已经给它抽象好了,但是我们发现节点写成这个样子代码是比较冗余。打比方说:node_linear = Node(input=[node_x, node_k, node_b], outputs=[node_sigmoid]), 既然我们已经告诉程序node_linear这个节点的输入是[node_x, node_k, node_b], 那其实也就是告诉程序这些节点的输出是node_linear。
好,我们接下来要在class Node里定义一个方法:
def __init__(self, inputs, outputs):self.inputs = inputsself.outputs = outputs
现在我们根据上面对代码冗余的分析,可以加上这样简单的一句:
def __init__(self, inputs=[]):self.inputs = inputsself.outputs = []for node in inputs:node.outputs.append(self)
把这句加上之后, 就可以只在里面输入inputs就行了,不用再输入outputs,代码就变得简单多了:
## Our Simple Model Elements
### version - 02
node_x = Node()
node_y = Node()
node_k = Node()
node_b = Node()
node_linear = Node(inputs=[node_x, node_k, node_b])
node_sigmoid = Node(inputs=[node_linear])
node_loss = Node(inputs=[node_sigmoid, node_y])
我们是把每个节点给它做出来了,那么怎么样能够把这个节点给它像串珠子一样串起来变成一张图呢?
其实我们只要去考察所有的边沿节点就可以了,把所有的x,y,k和b这种外层的函数给个变量:
need_expend = [node_x, node_y, node_k, node_b]
咱们再生成一个变量,这个变量是用来通过外沿这些节点,把连接图给生成出来。
computing_graph = defaultdict(list)while need_expend:n = need_expend.pop(0)if n in computing_graph: continuefor m in n.outputs:computing_graph[n].append(m)need_expend.append(m)
while里面,当外沿节点的list不为空的时候,我们就在里面来取一个点,我们就取第一个吧,取出来并删除。
然后如果这个点我们已经考察过了, 那就continue,如果没有,我们对于所有的这个n里边的outputs,插入到computing_graph的n的位置。再插入到外沿节点的list内。因为我们现在多了一个扩充节点,所以我们需要给插入进去。
比方说我们这次找出来了linear,把linear也加到这个需要扩充的点一行,然后就可以从linear再找到sigmoid了。
来,我们看下现在的这个computing_graph:
computing_graph---
defaultdict(list,{<__main__.Node at 0x12053e080>: [<__main__.Node at 0x12053dc30>],<__main__.Node at 0x12053e9b0>: [<__main__.Node at 0x12053ef50>],<__main__.Node at 0x12053d510>: [<__main__.Node at 0x12053dc30>],<__main__.Node at 0x12053c280>: [<__main__.Node at 0x12053dc30>],<__main__.Node at 0x12053dc30>: [<__main__.Node at 0x1202860e0>],<__main__.Node at 0x1202860e0>: [<__main__.Node at 0x12053ef50>]})
这样就获得出来了,其实是把它变成了刚刚的那个图。这样呢,我们就可以应用topologic来进行拓朴排序了。
topologic(computing_graph)---
[<__main__.Node at 0x12053c280>,<__main__.Node at 0x12053d510>,<__main__.Node at 0x12053e080>,<__main__.Node at 0x12053e9b0>,<__main__.Node at 0x12053dc30>,<__main__.Node at 0x1202860e0>,<__main__.Node at 0x12053ef50>]
但是我们打出来的内容都是一些内存地址,我们还需要改一下这个程序。我们在我们的class Node里多增加一个方法,用于return它的名字:
def __init__(self, inputs=[], name=None):...self.name = namedef __repr__(self):return 'Node:{} '.format(self.name)
这样之后,我们还需要改一下节点,在里面增加一个变量name=''。
node_x = Node(name='x')
...
node_loss = Node(inputs=[node_sigmoid, node_y], name='loss')
每一个都需要加上,我用...简化了代码。
然后我们再来看:
topologic(computing_graph)---
[Node:k , Node:x , Node:b , Node:linear , Node:sigmoid , Node:y , Node:loss ]
然后我们来将这段封装起来,变成一个函数:
feed_dict = {node_x: 3, node_y: random.random(),node_k: random.random(),node_b: 0.38
}def convert_feed_dict_to_graph(feed_dict):need_expend = [n for n in feed_dict]...return computing_graph
一般来说,很多大厂在建立代码的时候,x, y, k, b这种东西会被称为placeholder, 我们创建的need_expend会被称为是feed_dict。所以我们做了这样一个修改,将need_expend拿到方法里取重新获取。
这些节点刚开始的时候没有值,那我们给它一个初始值,我这里的值都是随意给的。
这样,就不仅把最外沿的节点给找出来了,而且还把值给他送进去了,相对来说就会更简单一些。所有定义出来的节点,我们都可以把它变成图关系。
topologic(convert_feed_dict_to_graph(feed_dict))---
[Node:k , Node:y , Node:b , Node:x , Node:linear , Node:sigmoid , Node:loss ]
咱们现在再定一个点,我们用一个变量存起来:
sorted_nodes = topologic(convert_feed_dict_to_graph(feed_dict))
那么咱们现在来模拟一下它的计算过程,模拟神经网络的计算过程。
class Node:...def fowward(self):print('I am {}, I calculate myself value!!!'.format(self.name))for node in sorted_nodes:node.forward()---
I am y, I calculate myself value!!!
I am x, I calculate myself value!!!
I am b, I calculate myself value!!!
I am k, I calculate myself value!!!
I am linear, I calculate myself value!!!
I am sigmoid, I calculate myself value!!!
I am loss, I calculate myself value!!!
我们在Node里定义了一个方法forward,从前往后运算,这个时候我们在每个里面加一个向前运算。
这个就是拓朴排序的作用,经过排序之后,那需要在后面计算的节点,就一定会放在后面再进行计算。
好,那我们现在需要区分两个内容,一个是被赋值的内容,一个是需要计算的内容。
刚才我们说过,在大厂的这些地方,x,y,k,b这种东西都被定义为占位符,那我们来修改一下代码:
class Node:def __init__(self, inputs=[], name=None):...def forward(self):print('I am {}, 我需要自己计算自己的值。'.format(self.name))...class Placeholder(Node):def __init__(self, name=None):Node.__init__(self, name = name)def forward(self):print('I am {}, 我已经被人为赋值了。'.format(self.name))def __repr__(self):return 'Node:{} '.format(self.name)### version - 02
node_x = Placeholder(name='x')
node_y = Placeholder(name='y')
node_k = Placeholder(name='k')
node_b = Placeholder(name='b')
node_linear = Node(inputs=[node_x, node_k, node_b], name='linear')
node_sigmoid = Node(inputs=[node_linear], name='sigmoid')
node_loss = Node(inputs=[node_sigmoid, node_y], name='loss')
我们创建了一个Placeholder类,继承了Node, 然后我们取修改初始化方法,它是是没有input的,只有一个name。
然后forward我们改一下,改成打印已经被赋值的语句。父类Node里的forward也改一下,改成需要自己计算自己的值。
那我们这个时候将赋值的四个节点改成调用Placeholder。
接下来,我们需要修改convert_feed_dict_to_graph方法了:
def convert_feed_dict_to_graph(feed_dict):...while need_expend:...if isinstance(n, Placeholder): n.value = feed_dict[n]......
我们来检查这个节点是否是Placeholder,如果是的话,将当前的feed_dict赋值给n.value。来看下结果:
for node in sorted_nodes:node.forward()---
I am b, 我已经被人为赋值了。
I am x, 我已经被人为赋值了。
I am k, 我已经被人为赋值了。
I am y, 我已经被人为赋值了。
I am linear, 我需要自己计算自己的值。
I am sigmoid, 我需要自己计算自己的值。
I am loss, 我需要自己计算自己的值。
好,到现在为止,咱们只是打了一段文字,问题是对于linear, sigmoid和loss, 到底是怎么计算的呢?
这个问题,咱们放到下一节课里面去讲,现在咱们这篇文章已经超标了,目测应该超过万字了吧。
好,下节课记得来看咱们具体如何在实现拓朴排序后将计算加进去。



)
鲜花的识别)
 python操作redis)










矩阵)

·逻辑运算)
