miRNA测序数据生信分析——第四讲,未知物种的生信分析实例
- miRNA测序数据生信分析——第四讲,未知物种的生信分析实例
- 1. 下载测序数据
- 2. 原始数据质控——软件fastqc
- 3. 注释tRNA和rRNA,使用Rfam数据库——软件blast,Rfam_statistics.py脚本
- 4. 注释miRNA,包括种类,序列及定量,靶基因和绘图
- 4.1 鉴定,使用miRBase数据库——软件blast
- 4.2 定量和miRNA序列提取
- 4.3 miRNA靶基因,使用miRanda和TargetScan软件进行预测
- 4.3.1 miRanda软件
- 4.3.2 TargetScan软件
- 4.3.3 整合两个软件预测结果——脚本Total_Target.py
- 4.4 绘制miRNA-靶基因互作图——软件Cytoscape
- 5. 总结
miRNA测序数据生信分析——第四讲,未知物种的生信分析实例
再次强调:这里未知物种,是指进行miRNA测序的物种在miRBase、miRDB、miRTarbase数据库都不存在miRNA信息。而不是不知道进行miRNA测序的物种
1. 下载测序数据
和博文(miRNA测序数据生信分析——第三讲,已知物种的生信分析实例)中的测序数据一样。假设这个数据的物种是Oecanthus indicus。
SRA号:DRR463940 单端测序 测序类型:miRNA-seq 文件DRR463940.fastq
2. 原始数据质控——软件fastqc
3. 注释tRNA和rRNA,使用Rfam数据库——软件blast,Rfam_statistics.py脚本
4. 注释miRNA,包括种类,序列及定量,靶基因和绘图
测序物种Oecanthus indicus(Oin)。该物种在miRBase、miRDB、miRTarbase数据库都不存在miRNA信息。
4.1 鉴定,使用miRBase数据库——软件blast
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/01miRBase
#查询物种Oecanthus indicus是否在数据库miRBase中
grep “Oecanthus indicus” /home/zhaohuiyao/Database/miRBase/organisms.txt #没有返回

#自己编辑三个字符简写为:oin
/home/zhaohuiyao/Biosoft/general/ncbi-blast-2.10.0+/bin/makeblastdb -in /home/zhaohuiyao/Database/miRBase/mature.fa -dbtype nucl -out /home/zhaohuiyao/Database/miRBase/mature
#只保留一个比对结果
/home/zhaohuiyao/Biosoft/general/ncbi-blast-2.10.0+/bin/blastn -task blastn-short -db /home/zhaohuiyao/Database/miRBase/mature -query …/…/00Rawdata/DRR463940.fasta -out DRR463940_miRBase.annotations -outfmt 6 -evalue 1e-5 -num_alignments 1
head DRR463940_miRBase.annotations

#统计
wc -l ./DRR463940_miRBase.annotations #63113条比对结果(63113/311289=20.27%)
cut -f 2 ./DRR463940_miRBase.annotations | awk ‘{name=substr($1,5,length($1)); print name}’ | sort | uniq | wc -l #420种miRNA(不关注物种,只关注miRNA种类)
4.2 定量和miRNA序列提取
miRNA序列提取:
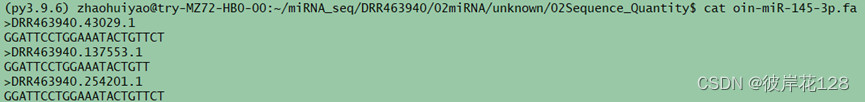
步骤一:提取一种miRNA(不关注物种)对应的测序Reads序列名称,并在测序Reads中进行序列提取,拿到该miRNA的所有可能序列
步骤二:对所有可能序列进行多序列比对(MATTF)
步骤三:依据MAFFT比对结果,提取一致性序列,该一致性序列为该测序物种miRNA序列
步骤四:整合所有miRNA序列
miRNA序列定量:
在运行上面步骤一时,会生成序列定量文件
步骤一:提取一种miRNA(不关注物种)对应的测序Reads序列名称,并在测序Reads中进行序列提取,拿到该miRNA的可能序列。
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/02Sequence_Quantity
python ./miRBase_sequence_unknown.py -i …/01miRBase/DRR463940_miRBase.annotations -data /home/zhaohuiyao/miRNA_seq/DRR463940/00Rawdata/DRR463940.fasta -s oin -o ./

#miRNA定量结果

#某一miRNA对应的是所有测序Reads序列
步骤二:对所有可能序列进行多序列比对(MATTF)
#安装MAFFT
官网下载最新安装包:https://mafft.cbrc.jp/alignment/software/
cd /home/zhaohuiyao/Biosoft/general
wget https://mafft.cbrc.jp/alignment/software/mafft-7.505-with-extensions-src.tgz
tar -zxvf ./mafft-7.505-with-extensions-src.tgz
cd mafft-7.505-with-extensions/core/
#编辑文件,vim Makefile。修改第一行内容,PREFIX = /usr/local PREFIX = /home/zhaohuiyao/Biosoft/general/mafft-7.505-with-extensions
make clean
make
make install
#可执行文件:/home/zhaohuiyao/Biosoft/general/mafft-7.505-with-extensions/bin/mafft
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/02Sequence_Quantity
mkdir MAFFT && cd MAFFT/
#编辑脚本mafft_run.sh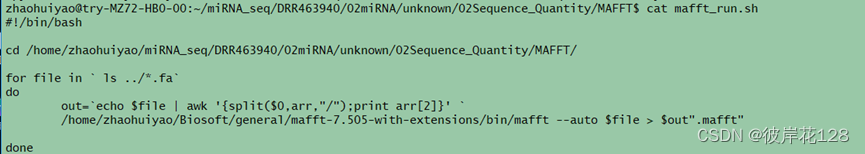
/bin/bash ./mafft_run.sh
#每个miRNA序列文件对应一个.mafft结果文件
步骤三:依据MAFFT比对结果,提取一致性序列,该一致性序列为本物种miRNA序列
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/02Sequence_Quantity/MAFFT/
mkdir consensus && cd consensus/
#编辑脚本consensus_run.sh以及consensus_seq.py
一致性序列的标准:依据多序列比对结果,统计每一个位置碱基的频次。频次最高的碱基为该位置碱基。
其中可能涉及的问题:①当该位置频次最高的是“-”字符时,忽略该位置。②当该位置频次最高的字符出现多个时,涉及到简并碱基的问题,这次暂不考虑,随机选择一种。
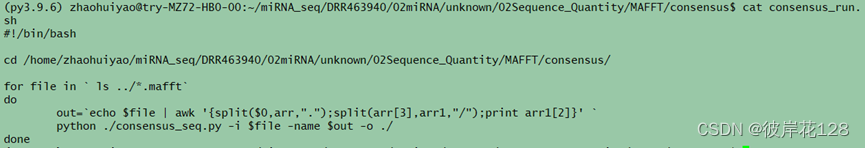
/bin/bash ./consensus_run.sh
#每个.mafft结果文件对应一个.concensus文件
步骤四:整合所有miRNA序列
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/02Sequence_Quantity
cat ./MAFFT/consensus/oin-* > DRR463940_miRBase.annotations.fa
4.3 miRNA靶基因,使用miRanda和TargetScan软件进行预测
#三个子目录miRanda/、TargetScan/和Total/
#两个软件的运行均需要准备两个文件。miRNA序列文件和mRNA序列文件。
miRNA序列文件:①成熟的miRNA序列,长度20-24nt;②种子序列,成熟miRNA 5’端的第2-8个核苷酸。
mRNA序列文件:①可以包括其他ncRNA序列(例如lncRNA也能与miRNA靶向结合);②可以是完整的mRNA序列,也可以是mRNA的仅3`UTR序列;③可以一个基因对应一个mRNA序列,也可以多个(最后会整合到靶基因水平。但是需要提供mRNA—Gene关系文件)
#准备文件
#miRNA序列文件:上面4.2拿到的miRNA序列文件
#mRNA序列文件:这个文件包括了其他ncRNA序列,完整mRNA序列,一个基因对应多个mRNA序列。
#mRNA—Gene关系文件
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/03Target/
#mRNA序列文件
wget https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/001/405/GCF_000001405.40_GRCh38.p14/GCF_000001405.40_GRCh38.p14_rna.fna.gz
gunzip ./GCF_000001405.40_GRCh38.p14_rna.fna.gz
#mRNA—Gene关系文件
grep ">" GCF_000001405.40_GRCh38.p14_rna.fna | awk -v OFS="\t" '{split($0,arr," "); name=substr(arr[1],2); split($0,arr1,")"); split(arr1[1],arr2,"("); print name,arr2[2]}' > mrna_gene.info#将fasta进行多行转单行
/home/zhaohuiyao/Biosoft/seqkit seq -i -w 0 GCF_000001405.40_GRCh38.p14_rna.fna > GCF_000001405.40_GRCh38.p14_rna.fna.tmp
rm GCF_000001405.40_GRCh38.p14_rna.fna
mv GCF_000001405.40_GRCh38.p14_rna.fna.tmp GCF_000001405.40_GRCh38.p14_rna.fna
4.3.1 miRanda软件
#安装软件miRanda
cd /home/zhaohuiyao/Biosoft/general
wget http://cbio.mskcc.org/microrna_data/miRanda-aug2010.tar.gz
tar -zxvf ./miRanda-aug2010.tar.gz
cd miRanda-3.3a/
./configure --prefix=/home/zhaohuiyao/Biosoft/general/miRanda-3.3a
make
make install
#可执行文件:/home/zhaohuiyao/Biosoft/general/miRanda-3.3a/bin/miranda
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/03Target/miRanda
ln -s …/GCF_000001405.40_GRCh38.p14_rna.fna oin_mrna.fasta
ln -s …/mrna_gene.info oin_gene_mrna.info.txt
/home/zhaohuiyao/Biosoft/general/miRanda-3.3a/bin/miranda …/…/02Sequence_Quantity/DRR463940_miRBase.annotations.fa oin_mrna.fasta -quiet -out DRR463940_miRBase.annotations.miRanda.original
#参数-quiet:表示不输出没有发生靶向的miRNA-mRNA关系
#结果文件DRR463940_miRBase.annotations.miRanda.original,这个文件中只有以">>"开头的行,才是预测的miRNA-靶mRNA的信息行
grep “>>” DRR463940_miRBase.annotations.miRanda.original > DRR463940_miRBase.annotations.miRanda.tmp
#结合mRNA—Gene的关系文件(oin_gene_mrna.info.txt),获得mRNA-靶基因关系文件
python3 ./miRanda_Target.py -i ./DRR463940_miRBase.annotations.miRanda.tmp -db ./oin_gene_mrna.info.txt -o ./
#结果文件DRR463940_miRBase.annotations.miRanda

4.3.2 TargetScan软件
#安装软件TargetScan
cd /home/zhaohuiyao/Biosoft/general
mkdir targetscan_50 && cd targetscan_50/
wget https://www.targetscan.org/vert_50/vert_50_data_download/targetscan_50.zip
unzip ./targetscan_50.zip && rm ./targetscan_50.zip
#可执行文件:/home/zhaohuiyao/Biosoft/general/targetscan_50/targetscan_50.pl
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/03Target/TargetScan
ln -s …/GCF_000001405.40_GRCh38.p14_rna.fna oin_mrna.fasta
ln -s …/mrna_gene.info oin_gene_mrna.info.txt
#TargetScan对输入的文件格式有要求。请依据要求对miRDB用到的文件进行修改,得到新的输入文件DRR463940_miRBase.annotations.fa.TargetScan和oin_mrna.fasta.TargetScan
#miRNA只需要种子序列(miRNA 5’端的第2~8个核苷酸),物种号:NCBI的Taxonomy数据库中查看1982312
awk ‘{if($0~/^>/){printf “%s\t”, substr($0,2) } else { printf “%s\t%s\n”, substr($0,2,7),“1982312”}}’ …/…/02Sequence_Quantity/DRR463940_miRBase.annotations.fa > RR463940_miRBase.annotations.fa.TargetScan
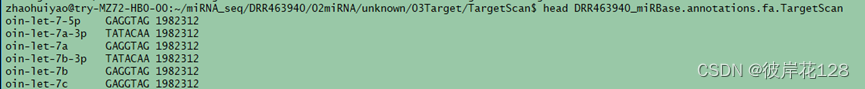
awk ‘{if($0~/^>/){printf “%s\t”, substr($0,2) } else { printf “%s\t%s\n”, “1982312”,$0}}’ oin_mrna.fasta > oin_mrna.fasta.TargetScan

perl /home/zhaohuiyao/Biosoft/general/targetscan_50/targetscan_50.pl DRR463940_miRBase.annotations.fa.TargetScan oin_mrna.fasta.TargetScan DRR463940_miRBase.annotations.TargetScan.original
#结合mRNA—Gene的关系文件(oin_gene_mrna.info.txt),获得mRNA-靶基因关系文件
python ./TargetScan_Target.py -i ./DRR463940_miRBase.annotations.TargetScan.original -db ./oin_gene_mrna.info.txt -o ./
#结果文件DRR463940_miRBase.annotations.TargetScan

4.3.3 整合两个软件预测结果——脚本Total_Target.py
#取并集,获得最终miRNA-Gene关系文件
cd /home/zhaohuiyao/miRNA_seq/DRR463940/02miRNA/unknown/03Target/Total
python ./Total_Target.py -db1 …/miRanda/DRR463940_miRBase.annotations.miRanda -db2 …/TargetScan/DRR463940_miRBase.annotations.TargetScan -o ./
#结果文件DRR463940_miRBase.annotations.target
4.4 绘制miRNA-靶基因互作图——软件Cytoscape
5. 总结
以上就是针对未知物种的miRNA分析。与已知物种的分析之间存在重叠,重点是两个预测软件miRanda和TargetScan的使用。上面步骤中涉及了很多脚本,但都是很简单的文件内容提取比对。
prometheus监控DMHS操作步骤)




)
特别慢,php 7.0 unserialize 特别慢)

, sort()))




)
-Eureka服务注册中心)
 disconnected)



