目录
主要内容
模型研究
部分代码
结果一览
下载链接
主要内容
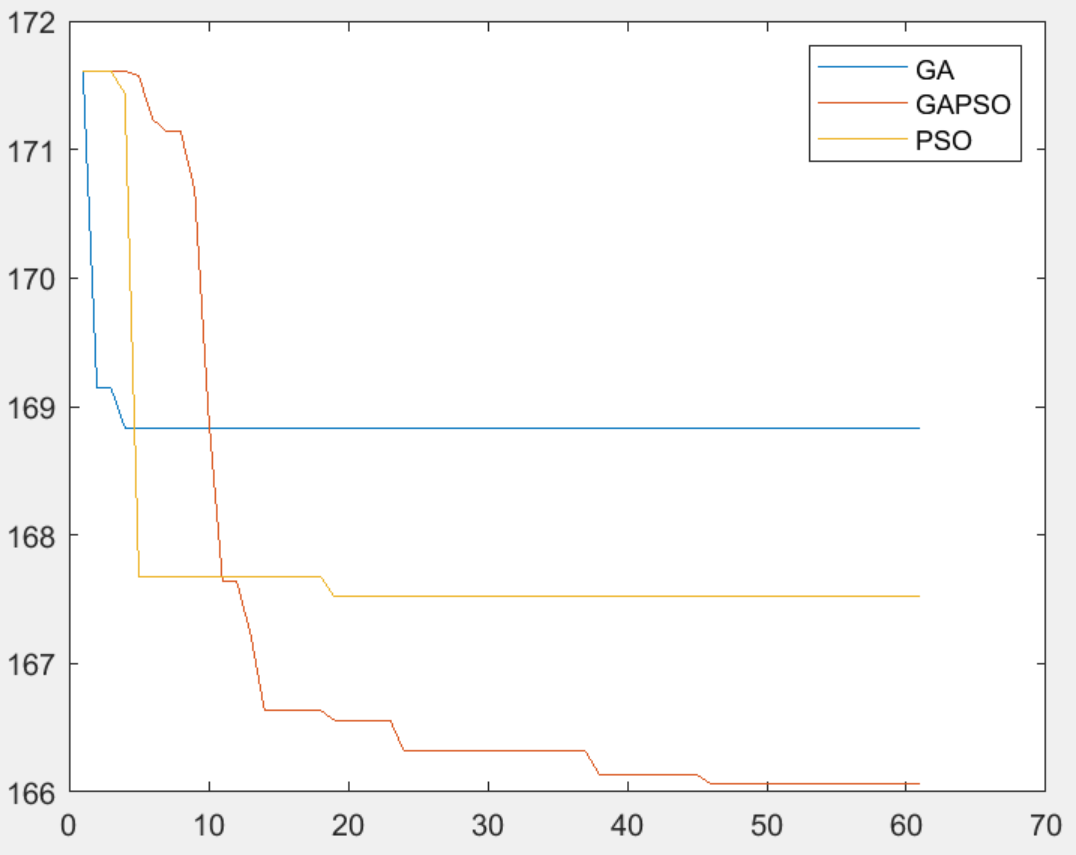
为了更高效地完成复杂未知环境下的无人机快速探索任务,很多智能算法被应用于无人机路径规划方面的研究,但是传统粒子群算法存在粒子更新思路单一、随机性受限、收敛速度慢、 易陷入局部最优等问题,因此,需要结合无人机探索特点,将粒子群算法和遗传算法结合形成改进粒子群算法,提高算法的全局寻优能力,从而增强无人机路径规划的效率。
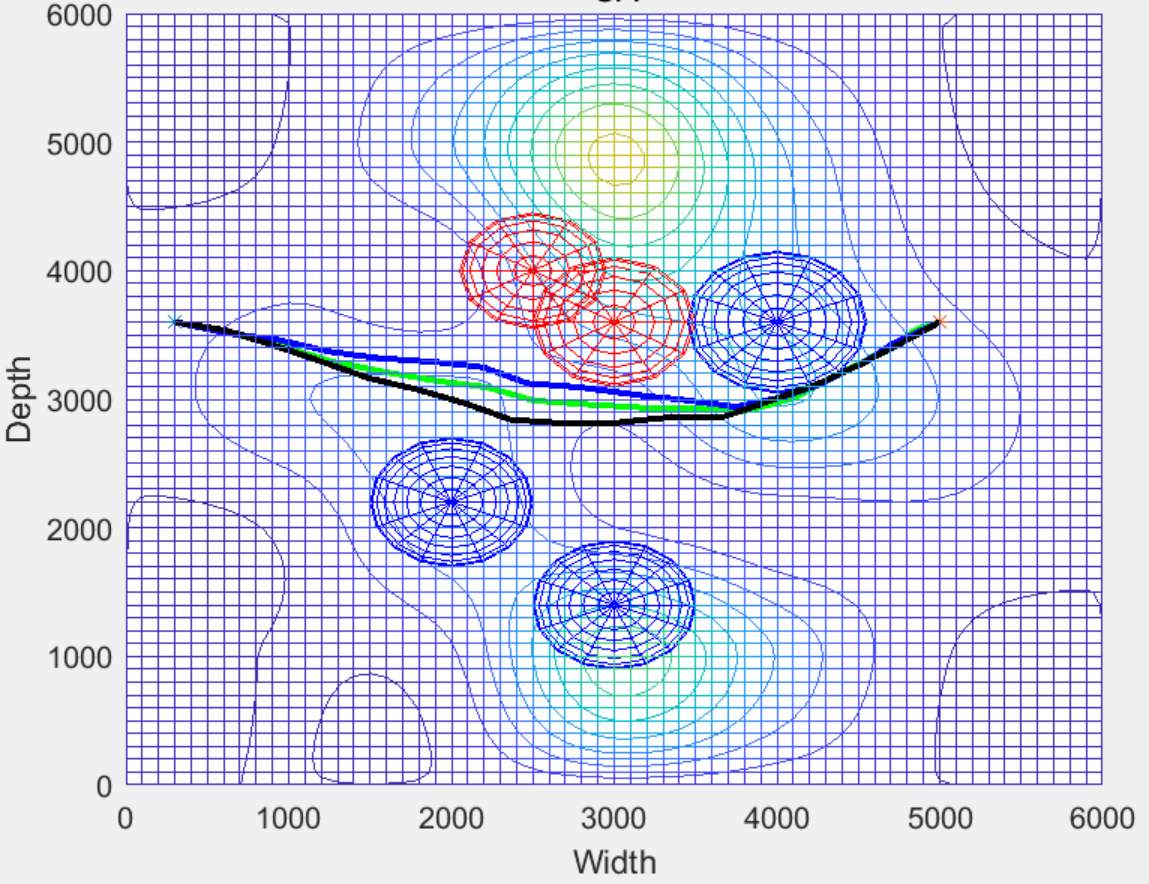
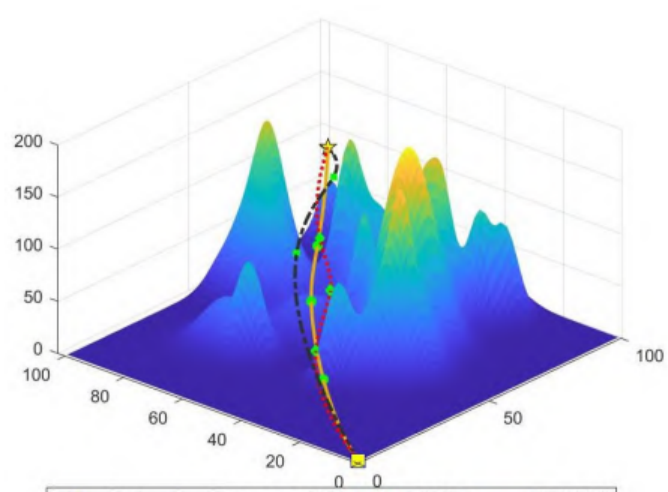
程序采用遗传粒子群、基本粒子群和遗传算法三种算法进行路径规划,并对比优化效果,在综合考虑路径最优性、平滑性和安全性的基础上构建了聚合适应度函数,程序模块化编程,方便研究学习!
模型研究
聚合适应度函数建模
适应度评价函数是衡量当前所选路径优劣的客观标准。为了确保获得最优的路径规划效果,必须根据不同的应用场景选择相适应的评价准则。
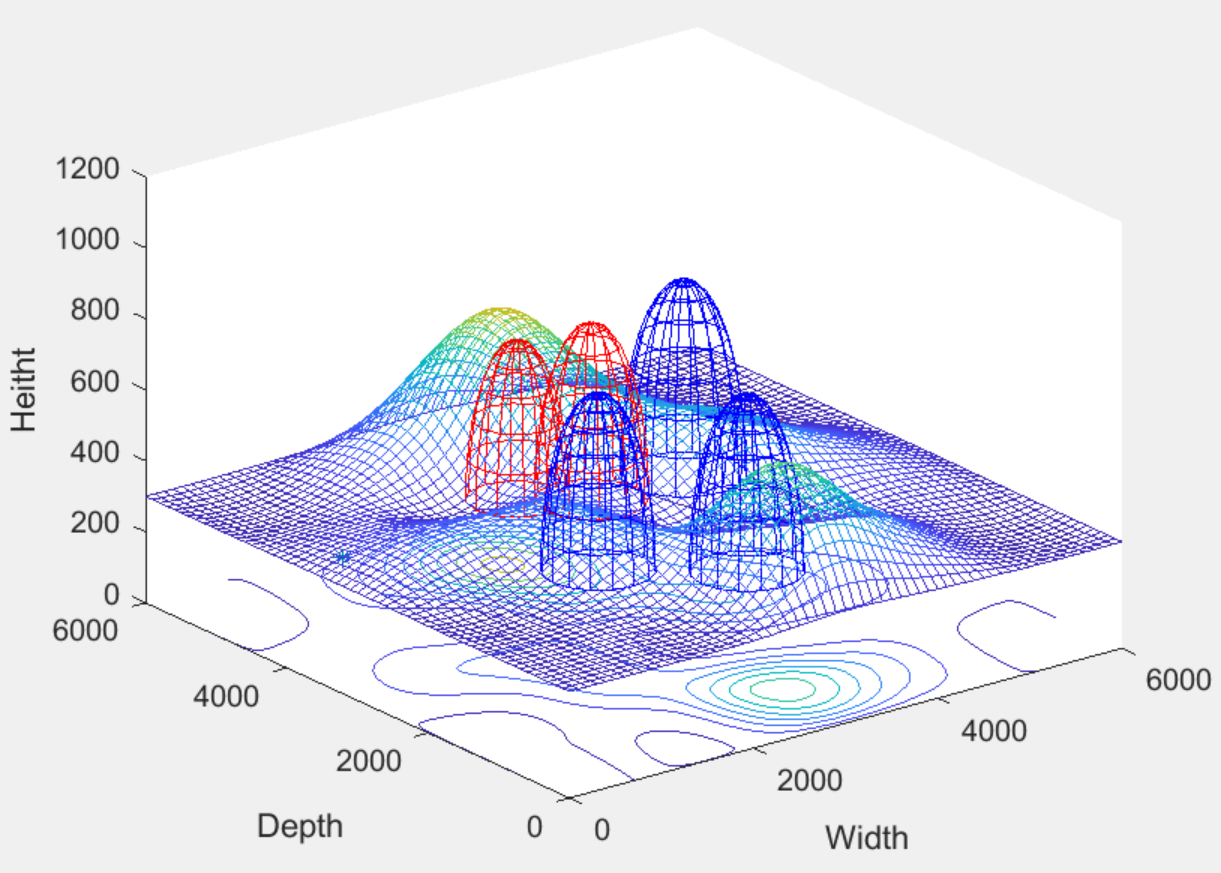
路径长度是无人机路径规划首当其冲需要考虑的,因为无人机续航有限,只有在有限的续航范围内利用最短距离完成探索才能最大程度确保规划方案的可行性。不难得出,路径长度计算公式如下:
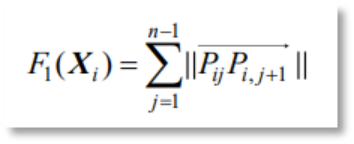
从而可以看出,总的路径长度值是由所有航段求和得到,该值越小说明路径越短。
航迹平滑度是综合考虑无人机能量有限和飞行加减速的影响,在任务执行时,无人机普遍面临内置能量有限、续航时间短的困难。当规划路径包含过多曲折时,无人机在飞行过程中频繁进行加减速,导致能量加速消耗,从而压缩了有效作业时间。因此,为了最大程度提高任务执行效率,对路径平滑性提出了迫切需求。
因此,无人机飞行高度也需要进行限制,从而保证航迹平滑性。
路径安全性考虑无人机和探测雷达的位置关系,以保证飞行过程的安全性。
部分代码
%初始染色体个数 chromosome = repmat(my_chromosome,model.NP,1); %子代染色体 next_chromosome = repmat(my_chromosome,model.NP,1); %种群的适应度值 seeds_fitness=zeros(1,model.NP); %全局最优 p_global.cost=inf; %适应度最优值保留 best=zeros(model.MaxIt+1,1); best(1)=model.globel.cost; %种群初始化 for i=1:model.NPchromosome(i).pos=model.chromosome(i).pos;chromosome(i).alpha=model.chromosome(i).alpha;chromosome(i).beta=model.chromosome(i).beta;chromosome(i).atkalpha=model.chromosome(i).atkalpha;chromosome(i).atkbeta=model.chromosome(i).atkbeta;chromosome(i).T=model.chromosome(i).T;chromosome(i).sol=model.chromosome(i).sol;chromosome(i).cost=model.chromosome(i).cost;chromosome(i).IsFeasible=model.chromosome(i).IsFeasible; seeds_fitness(i)=model.seeds_fitness(i);for d=1:3chromosome(i).vel(d,:)= zeros(1,model.dim);end%更新历史最优粒子chromosome(i).best.pos =chromosome(i).pos;chromosome(i).best.alpha =chromosome(i).alpha;chromosome(i).best.beta =chromosome(i).beta;chromosome(i).best.T =chromosome(i).T;chromosome(i).best.sol =chromosome(i).sol;chromosome(i).best.cost =chromosome(i).cost;%更新全局最优例子if p_global.cost > chromosome(i).best.costp_global = chromosome(i).best;endend for it=1:model.MaxIt%得到最大和平均适应度值model.f_max =max(seeds_fitness);model.f_avg =mean(seeds_fitness);%按照适应度对染色体排序sort_array =zeros(model.NP,2);for i=1:model.NPsort_array(i,:)= [i,chromosome(i).cost];end%以cost从小到大进行排序sort_array =sortrows(sort_array,2);model.p_global =p_global;%只保留前一半的染色体,后一般抛弃
结果一览