目录
一、原理
1.数据中心化
2.白数据与目标
3.协方差与协方差矩阵
4.特征值与特征向量
5.最终结果构造
二、代码
代码解释:
三、结果
结果解释:
pca优缺点分析:
参考视频:
一、原理
目的:
pca是为了将原始的数据集的特征维度减少。

如上图,横纵坐标代表两个特征,可以看到这两个特征正相关程度很大,都快可以用一条直线连起来了。
假如能把它变成像下图这样的,能很大程度的保留样本点的信息,又能够减少特征维度,那就实现了pca的降维(将两个特征用一个主成分表示)。

一些概念:
1.数据中心化
假设原来的数据是二维的,并且将样本点放在坐标系中是这样

中心化后,结果如下图。

实现:
对每个特征,先求出他的均值:
再将每个样本的该特征,都减去这个均值
对每个特征都执行了这样的操作就结束了。
2.白数据与目标
先假设假设数据中心化后如左图,为了引出目标,假设右图就是他的白数据。
 变成
变成
设白数据样本集为

样本的特征x和特征y是独立的,并且服从标准正态分布。
其中D是样本集,列数是样本数量,行数为特征数量。
拉伸:
现将一个矩阵![]() 乘上数据集,得到:
乘上数据集,得到:

结果如下图

就可以实现将这个样本集在X轴上“拉长”
旋转:
假设SD左乘一个矩阵![]() ,就变为下图:
,就变为下图:

现在我们手上的数据便是这个“RSD”,其中这个R是最重要的,PCA需要求出这个矩阵R,也就是旋转坐标轴的角度。
一些补充:
3.协方差与协方差矩阵
协方差定义:
其中:n 是样本数量,Xi 和 Yi 分别表示第 i 个样本的 X 和 Y 值,分别是 X 和 Y 的均值。
在PCA中,X和Y即为特征。
协方差的意义是:表示X与Y的相关程度,若正相关,协方差大于0,负相关则小于0,若几乎为0,则不相关。
显然的,当Y=X时,协方差分子即为方差。
由于已经去中心化了,所以均值都为0。
协方差矩阵:

将协方差带入:
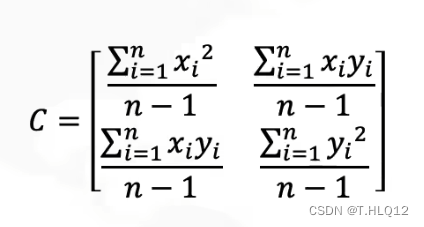

![]()
其中D是上面的白数据。
将D替换为RSD,则已经中心化后的数据的协方差矩阵为:

第4行中,因为![]() ,其中白数据的X与Y是不相关的,所以协方差为0,而对角线上为方差,并且X与Y是标准正态分布,所以为1。
,其中白数据的X与Y是不相关的,所以协方差为0,而对角线上为方差,并且X与Y是标准正态分布,所以为1。
第5行中,RT = R-1在上面有补充,第6行中,L定义为S*ST。
算完这个玩意之后,自然有用处,和接下来的式子形式上相同。
4.特征值与特征向量
假如以下式子成立
,其中A为矩阵,v为列数为1,行数与A相等的矩阵,
为标量。
那么v就为矩阵A的特征向量,就为特征值。
现在先给出以下两个式子(因为现在假设原数据集特征维度为2)

由线性代数基础知识,可由上两式得到:

可以看作:

两边同右乘R-1,又因为R-1=RT,所以得到:

便可以将R中第一列看作特征向量1,第二列看作特征向量2。


这样,求解这个R,就是求出了特征向量了。
而L则是特征值。
5.最终结果构造
求出了这个R后,根据特征值大小对特征向量进行排序,选择前几个特征向量对应的主成分,这些主成分能够解释数据中最多的方差。
然后构造特征向量矩阵:将选择的主要特征向量排列成一个矩阵。假设选择了 k 个主成分,那么这个矩阵的大小将是 d*k,d是原始数据的维度
将中心化后的数据矩阵 X 乘以特征向量矩阵 W,得到的数据矩阵 Z = XW 的大小为 n * k,即为最终结果。
二、代码
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaleriris = datasets.load_iris()
X = iris.data
y = iris.targetscaler = StandardScaler()
X_scaled = scaler.fit_transform(X)pca = PCA(n_components=2)
X_pca = pca.fit_transform(X_scaled)#创建一个图形并设置子图
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(16, 6))#降维前的数据前两个特征
ax1.set_title("origin")
for i, target_name in zip([0, 1, 2], iris.target_names):ax1.scatter(X[y == i, 0], X[y == i, 1], label=target_name)
ax1.set_xlabel('X1')
ax1.set_ylabel('X2')
ax1.legend()#降维后的数据
ax2.set_title("after")
for i, target_name in zip([0, 1, 2], iris.target_names):ax2.scatter(X_pca[y == i, 0], X_pca[y == i, 1], label=target_name)
ax2.set_xlabel('X1')
ax2.set_ylabel('X2')
ax2.legend()plt.show()
代码解释:
由于这次老师说可以用库函数,所以比较简单。
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X_scaled)这里是主要部分,第一行就是创建个pca对象,指定主成分数量为2(也就是接下来往这个对象中传入数据,返回出来的是二维特征的数据),然后第二行就是将数据集扔进去,也就是X_scaled,
返回的X_pca即是处理完的数据集。
三、结果

结果解释:
使用鸢尾花数据集来作为本次实验的数据集。
左图是原数据使用前两个特征展现出的图,右图是pca的结果。
可以看到左图的黄绿点比较混杂,而右图的黄绿点左右分的比较开,两张图的蓝点都比黄绿点分的开。而把黄绿点分的比左图开的右图,就是pca的功劳与结果。
pca优缺点分析:
优点:
-
降低数据维度:PCA通过投影数据到较低维度的主成分空间,实现了数据的降维。这有助于减少数据存储和计算成本,并简化后续分析过程。
-
消除特征间相关性:PCA通过转换数据,使得新的特征向量(主成分)之间彼此正交,从而消除原始特征之间的相关性。这有助于减少多重共线性问题,并提高后续模型的准确性和稳定性。
-
保留最大方差信息:PCA将数据投影到方差最大的主成分上,这意味着它保留了数据中最重要的信息。这使得我们能够更好地理解数据集的结构和变异性,以及更好地解释数据。
-
可视化数据:降维后的数据可以更容易地可视化,因为它们通常只涉及两个或三个主成分。这有助于发现数据中的模式、聚类和异常值。
缺点:
-
信息损失:降维过程中,为了将数据映射到较低维度,必然会丢失一部分信息。降维后的数据无法完全还原原始数据,因此可能会损失一些细节和特征。
-
主成分的可解释性:虽然PCA可以帮助我们找到方差最大的主成分,但这些主成分通常很难直接解释。它们是原始特征的线性组合,因此不容易与现实世界中的具体含义对应。
-
对离群值敏感:PCA对离群值比较敏感,因为它是基于数据的协方差矩阵或相关矩阵计算的。离群值可能会对协方差或相关性的计算产生较大的影响,从而导致降维结果不准确。
-
计算复杂性:PCA的计算复杂度较高,特别是在处理大规模数据集时。计算协方差矩阵或相关矩阵需要较大的计算和存储开销。
参考视频:
参考视频1
参考视频2
参考视频3






)











)

