与细胞检测识别相关的项目在之前的文章中也有不少的实践,感兴趣的话可以自行移步阅读即可。
《服务医学,基于目标检测模型实现细胞检测识别》
《基于轻量级ShuffleNetv2+YOLOv5的DIC-C2DH-HeLa细胞检测识别分析系统》
《基于YOLOv5开发构建荧光染色条件下膀胱脱落细胞检测识别分析系统》
本文的主要目的就是想要基于目标检测模型来实现对支气管肺泡灌洗液细胞的检测识别与计数分析,首先看下实例效果,如下所示:

简单看下数据集,如下所示:

实例标注内容如下所示:
0 0.310547 0.347331 0.149414 0.142578
0 0.734131 0.693034 0.120605 0.143880
1 0.022705 0.785156 0.041504 0.065104
1 0.045166 0.578776 0.040527 0.067708
1 0.135498 0.169596 0.057129 0.069661
1 0.218506 0.154948 0.042480 0.062500
1 0.194580 0.392578 0.051270 0.066406
1 0.148926 0.554688 0.048828 0.058594
1 0.168457 0.803385 0.048828 0.062500
1 0.214844 0.729818 0.045898 0.070312
1 0.220947 0.550781 0.054199 0.066406
1 0.255859 0.256836 0.048828 0.072266
1 0.316650 0.277344 0.050293 0.063802
1 0.353027 0.221029 0.047852 0.057943
1 0.431396 0.281901 0.046387 0.066406
1 0.337891 0.447917 0.046875 0.058594
1 0.320801 0.698568 0.055664 0.061198
1 0.394043 0.468099 0.052734 0.062500
1 0.412598 0.534505 0.048828 0.070312
1 0.407959 0.596354 0.056152 0.063802
1 0.414307 0.737956 0.043457 0.056641
1 0.452637 0.759115 0.049805 0.062500
1 0.476074 0.488932 0.054688 0.070312
1 0.500000 0.425130 0.054688 0.067708
1 0.549316 0.200195 0.047852 0.057943
1 0.587158 0.181315 0.042480 0.068359
1 0.598145 0.242188 0.043945 0.062500
1 0.600830 0.713867 0.053223 0.069661
1 0.637207 0.805990 0.044922 0.067708
1 0.639648 0.153646 0.049805 0.070312
1 0.673340 0.205729 0.048828 0.063802
1 0.689697 0.138346 0.054199 0.078776
1 0.741699 0.144531 0.049805 0.069010
1 0.677246 0.366536 0.047852 0.063802
1 0.810059 0.321940 0.051758 0.080078
1 0.826660 0.125977 0.045898 0.070964
1 0.795654 0.782552 0.039551 0.057292
1 0.821045 0.894206 0.046387 0.072266
1 0.818359 0.833984 0.047852 0.062500
1 0.901367 0.828776 0.049805 0.069010
1 0.865723 0.675781 0.048828 0.062500
1 0.919678 0.713216 0.055176 0.073568
1 0.976807 0.263021 0.046387 0.070312
1 0.945801 0.321940 0.052734 0.067057
1 0.921631 0.359375 0.055176 0.066406
1 0.955566 0.396484 0.049805 0.066406
1 0.960693 0.458333 0.039551 0.059896
1 0.968994 0.654948 0.056152 0.082031
2 0.307129 0.608073 0.147461 0.167969
2 0.322510 0.824544 0.140137 0.227214
2 0.515625 0.675130 0.127930 0.213542
2 0.600830 0.419596 0.134277 0.201172
2 0.594482 0.066406 0.127441 0.131510
2 0.739014 0.060872 0.169434 0.120443
2 0.783447 0.217122 0.154785 0.203776
2 0.625244 0.606445 0.122559 0.164714
2 0.559082 0.888021 0.198242 0.223958
2 0.716553 0.937500 0.120605 0.111979
2 0.813721 0.509440 0.281738 0.356120
2 0.927002 0.930339 0.138184 0.139323
2 0.958008 0.775716 0.083984 0.184245
共包含四种目标细胞类别,如下所示:
嗜酸性粒细胞
淋巴细胞
巨噬细胞
中性粒细胞默认使用m系列的模型来开发模型,模型文件如下所示:
# YOLOv5 🚀 by Ultralytics, GPL-3.0 license# Parameters
nc: 4 # number of classes
depth_multiple: 0.67 # model depth multiple
width_multiple: 0.75 # layer channel multiple
anchors:- [10,13, 16,30, 33,23] # P3/8- [30,61, 62,45, 59,119] # P4/16- [116,90, 156,198, 373,326] # P5/32# YOLOv5 v6.0 backbone
backbone:# [from, number, module, args][[-1, 1, Conv, [64, 6, 2, 2]], # 0-P1/2[-1, 1, Conv, [128, 3, 2]], # 1-P2/4[-1, 3, C3, [128]],[-1, 1, Conv, [256, 3, 2]], # 3-P3/8[-1, 6, C3, [256]],[-1, 1, Conv, [512, 3, 2]], # 5-P4/16[-1, 9, C3, [512]],[-1, 1, Conv, [1024, 3, 2]], # 7-P5/32[-1, 3, C3, [1024]],[-1, 1, SPPF, [1024, 5]], # 9]# YOLOv5 v6.0 head
head:[[-1, 1, Conv, [512, 1, 1]],[-1, 1, nn.Upsample, [None, 2, 'nearest']],[[-1, 6], 1, Concat, [1]], # cat backbone P4[-1, 3, C3, [512, False]], # 13[-1, 1, Conv, [256, 1, 1]],[-1, 1, nn.Upsample, [None, 2, 'nearest']],[[-1, 4], 1, Concat, [1]], # cat backbone P3[-1, 3, C3, [256, False]], # 17 (P3/8-small)[-1, 1, Conv, [256, 3, 2]],[[-1, 14], 1, Concat, [1]], # cat head P4[-1, 3, C3, [512, False]], # 20 (P4/16-medium)[-1, 1, Conv, [512, 3, 2]],[[-1, 10], 1, Concat, [1]], # cat head P5[-1, 3, C3, [1024, False]], # 23 (P5/32-large)[[17, 20, 23], 1, Detect, [nc, anchors]], # Detect(P3, P4, P5)]
训练数据配置文件如下所示:
# Dataset
path: ./dataset
train:- images/train
val:- images/test
test:- images/test# Classes
names:0: Eosinophil1: Lymphocyte2: Macrophage3: Neutrophil默认执行100次epoch迭代计算,终端输出如下所示:

训练完成结果输出如下所示:
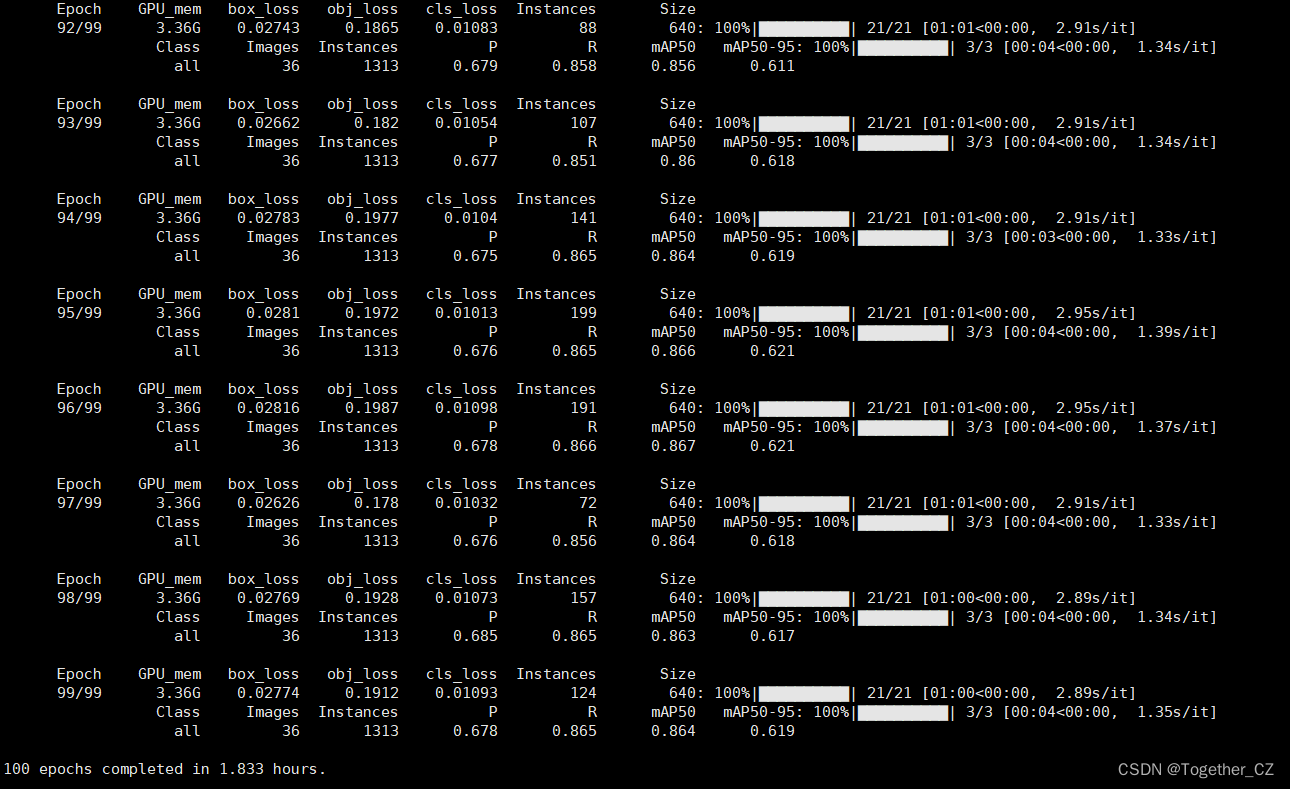
评估结果如下所示:
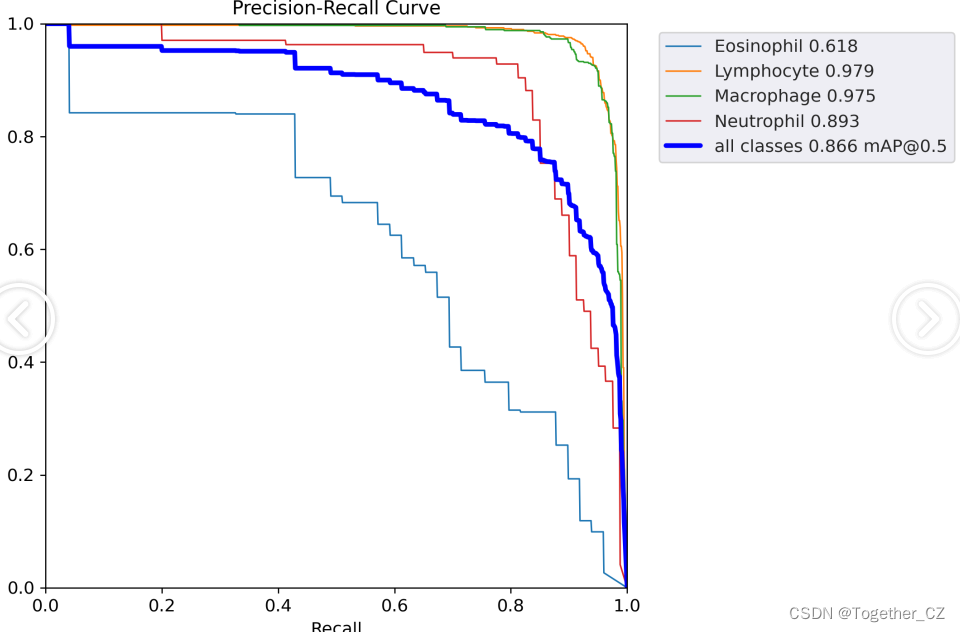
Class Images Instances P R mAP50 mAP50-95: 100%|??????????| 3/3 [00:08<00:00, 2.71s/it] all 36 1313 0.676 0.865 0.866 0.624Eosinophil 36 49 0.627 0.592 0.618 0.399Lymphocyte 36 703 0.726 0.983 0.979 0.756Macrophage 36 481 0.806 0.973 0.975 0.787Neutrophil 36 80 0.547 0.912 0.893 0.553接下来看下结果详情。
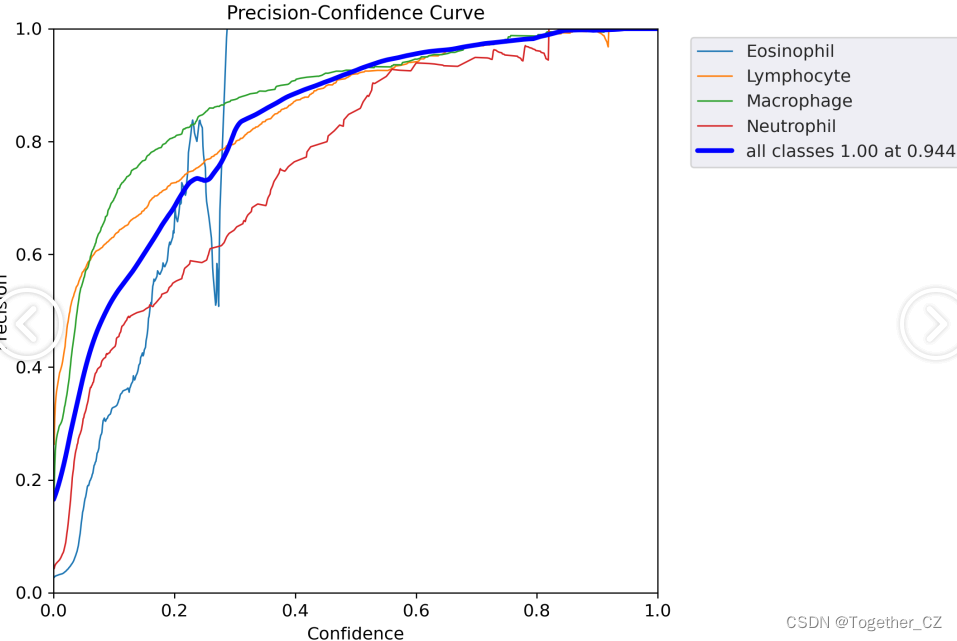
【Precision曲线】
精确率曲线(Precision-Recall Curve)是一种用于评估二分类模型在不同阈值下的精确率性能的可视化工具。它通过绘制不同阈值下的精确率和召回率之间的关系图来帮助我们了解模型在不同阈值下的表现。
精确率(Precision)是指被正确预测为正例的样本数占所有预测为正例的样本数的比例。召回率(Recall)是指被正确预测为正例的样本数占所有实际为正例的样本数的比例。
绘制精确率曲线的步骤如下:
使用不同的阈值将预测概率转换为二进制类别标签。通常,当预测概率大于阈值时,样本被分类为正例,否则分类为负例。
对于每个阈值,计算相应的精确率和召回率。
将每个阈值下的精确率和召回率绘制在同一个图表上,形成精确率曲线。
根据精确率曲线的形状和变化趋势,可以选择适当的阈值以达到所需的性能要求。
通过观察精确率曲线,我们可以根据需求确定最佳的阈值,以平衡精确率和召回率。较高的精确率意味着较少的误报,而较高的召回率则表示较少的漏报。根据具体的业务需求和成本权衡,可以在曲线上选择合适的操作点或阈值。
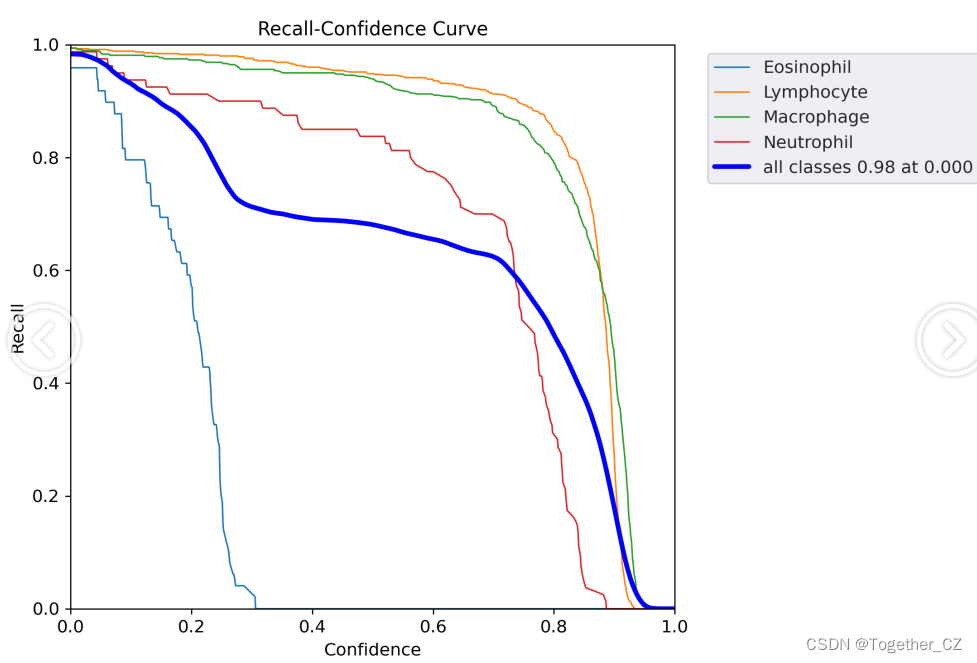
【Recall曲线】
召回率曲线(Recall Curve)是一种用于评估二分类模型在不同阈值下的召回率性能的可视化工具。它通过绘制不同阈值下的召回率和对应的精确率之间的关系图来帮助我们了解模型在不同阈值下的表现。
召回率(Recall)是指被正确预测为正例的样本数占所有实际为正例的样本数的比例。召回率也被称为灵敏度(Sensitivity)或真正例率(True Positive Rate)。
绘制召回率曲线的步骤如下:
使用不同的阈值将预测概率转换为二进制类别标签。通常,当预测概率大于阈值时,样本被分类为正例,否则分类为负例。
对于每个阈值,计算相应的召回率和对应的精确率。
将每个阈值下的召回率和精确率绘制在同一个图表上,形成召回率曲线。
根据召回率曲线的形状和变化趋势,可以选择适当的阈值以达到所需的性能要求。
通过观察召回率曲线,我们可以根据需求确定最佳的阈值,以平衡召回率和精确率。较高的召回率表示较少的漏报,而较高的精确率意味着较少的误报。根据具体的业务需求和成本权衡,可以在曲线上选择合适的操作点或阈值。
【F1值曲线】
F1值曲线是一种用于评估二分类模型在不同阈值下的性能的可视化工具。它通过绘制不同阈值下的精确率(Precision)、召回率(Recall)和F1分数的关系图来帮助我们理解模型的整体性能。
F1分数是精确率和召回率的调和平均值,它综合考虑了两者的性能指标。F1值曲线可以帮助我们确定在不同精确率和召回率之间找到一个平衡点,以选择最佳的阈值。
绘制F1值曲线的步骤如下:
使用不同的阈值将预测概率转换为二进制类别标签。通常,当预测概率大于阈值时,样本被分类为正例,否则分类为负例。
对于每个阈值,计算相应的精确率、召回率和F1分数。
将每个阈值下的精确率、召回率和F1分数绘制在同一个图表上,形成F1值曲线。
根据F1值曲线的形状和变化趋势,可以选择适当的阈值以达到所需的性能要求。
F1值曲线通常与接收者操作特征曲线(ROC曲线)一起使用,以帮助评估和比较不同模型的性能。它们提供了更全面的分类器性能分析,可以根据具体应用场景来选择合适的模型和阈值设置。

【PR曲线】
精确率-召回率曲线(Precision-Recall Curve)是一种用于评估二分类模型性能的可视化工具。它通过绘制不同阈值下的精确率(Precision)和召回率(Recall)之间的关系图来帮助我们了解模型在不同阈值下的表现。
精确率是指被正确预测为正例的样本数占所有预测为正例的样本数的比例。召回率是指被正确预测为正例的样本数占所有实际为正例的样本数的比例。
绘制精确率-召回率曲线的步骤如下:
使用不同的阈值将预测概率转换为二进制类别标签。通常,当预测概率大于阈值时,样本被分类为正例,否则分类为负例。
对于每个阈值,计算相应的精确率和召回率。
将每个阈值下的精确率和召回率绘制在同一个图表上,形成精确率-召回率曲线。
根据曲线的形状和变化趋势,可以选择适当的阈值以达到所需的性能要求。
精确率-召回率曲线提供了更全面的模型性能分析,特别适用于处理不平衡数据集和关注正例预测的场景。曲线下面积(Area Under the Curve, AUC)可以作为评估模型性能的指标,AUC值越高表示模型的性能越好。
整体训练过程loss和metrics评估可视化如下所示:
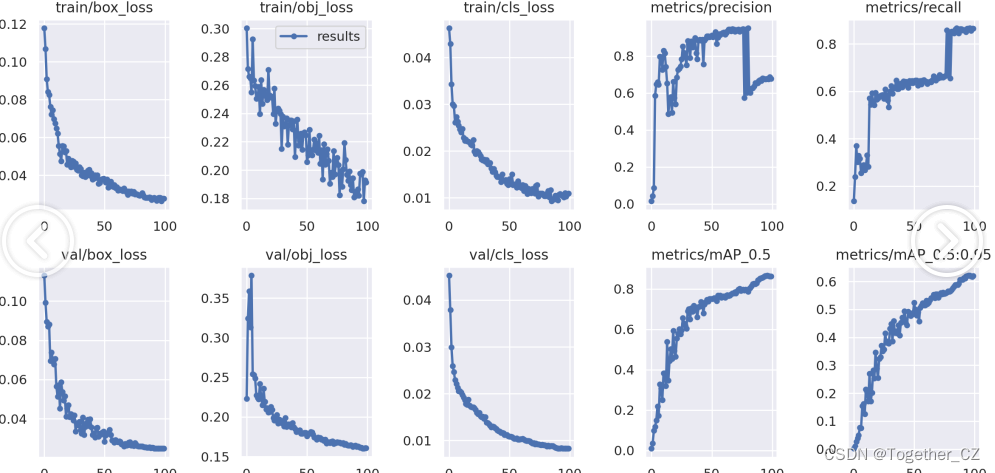
混淆矩阵如下所示:

集成到可视化界面中实现可视化推理,实例如下所示:


感觉这种跟实际比较有联系的项目还是比较有意义的,在实际工作中感兴趣的话都可以多多尝试下。
“软件测试”赛项竞赛规程)



)











![[论文阅读]A ConvNet for the 2020s](http://pic.xiahunao.cn/[论文阅读]A ConvNet for the 2020s)


