
转录组生信分析教程
桓峰基因公众号推出转录组分析教程,有需要生信的老师可以联系我们!转录分析教程整理如下:
RNA 1. 基因表达那些事--基于 GEO
RNA 2. SCI文章中基于GEO的差异表达基因之 limma
RNA 3. SCI 文章中基于T CGA 差异表达基因之 DESeq2
RNA 4. SCI 文章中基于TCGA 差异表达之 edgeR
RNA 5. SCI 文章中差异基因表达之 MA 图
RNA 6. 差异基因表达之-- 火山图 (volcano)
RNA 7. SCI 文章中的基因表达——主成分分析 (PCA)
RNA 8. SCI文章中差异基因表达--热图 (heatmap)
RNA 9. SCI 文章中基因表达之 GO 注释
RNA 10. SCI 文章中基因表达富集之--KEGG
RNA 11. SCI 文章中基因表达富集之 GSEA
RNA 12. SCI 文章中肿瘤免疫浸润计算方法之 CIBERSORT
RNA 13. SCI 文章中差异表达基因之 WGCNA
RNA 14. SCI 文章中差异表达基因之 蛋白互作网络 (PPI)
RNA 15. SCI 文章中的融合基因之 FusionGDB2
RNA 16. SCI 文章中的融合基因之可视化
RNA 17. SCI 文章中的筛选 Hub 基因 (Hub genes)
RNA 18. SCI 文章中基因集变异分析 GSVA
RNA 19. SCI 文章中无监督聚类法 (ConsensusClusterPlus)
RNA 20. SCI 文章中单样本免疫浸润分析 (ssGSEA)
RNA 21. SCI 文章中单基因富集分析
RNA 22. SCI 文章中基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(ESTIMATE)
RNA 23. SCI文章中表达基因模型的风险因子关联图(ggrisk)
RNA 24. SCI文章中基于TCGA的免疫浸润细胞分析 (TIMER)
RNA 25. SCI文章中估计组织浸润免疫细胞和基质细胞群的群体丰度(MCP-counter)
RNA 25. SCI文章中只有生信没有实验该怎么办?
RNA 26. SCI文章中基于转录组数据的基因调控网络推断 (GENIE3)
RNA 27 SCI文章中转录因子结合motif富集到调控网络 (RcisTarget)
RNA 28 SCI 文章中基于RNA-seq数据反褶积揭示肿瘤免疫结构的分子和药理学 (quanTIseq)
RNA 29. SCI文章中基于TCGA的免疫浸润细胞分析 (TIMER2.0)
RNA 30. SCI文章中基于TCGA和GTEx数据挖掘神器(GEPIA2)
RNA 31. SCI文章临床蛋白质组肿瘤在线数据挖掘神器(CPTAC)
RNA 32. SCI文章临床多组学肿瘤在线数据挖掘神器(UALCAN)
RNA 33. SCI文章肿瘤在线数据挖掘神器(cBioportal)
RNA 34. SCI 文章基因蛋白表达时间趋势分析及划分聚类群 (Mfuzz)
RNA 35. SCI 文章基于RNA-seq推断CNVs (RNAseqCNV)
简 介
RNA 测序实验产生大量关于基因表达水平的信息。虽然它们主要用于量化表达水平,但它们包含更多重要的生物学信息,如拷贝数变异 (CNVs)。CaSpER 提出了一种信号处理方法,用于识别,可视化和综合分析多尺度分辨率的焦点和大规模 CNV 事件,使用大量或单细胞 RNA 测序数据。CaSpER 融合了表达信号和等位基因移位信号的多尺度平滑,用于CNV 调用。等位基因位移信号测量杂合度损失 (LOH) 对 CNV 鉴定有价值。CaSpER 采用了一种有效的方法,从 reads 中产生全基因组 B 等位基因频率 (BAF) 信号谱,并用来校正 CNVs 识别。CaSpER 增加了 RNA 测序数据集的实用性,并用于完整表征和可视化单细胞或 RNA-seq 数据的基因组和转录组学分布情况。
B-allele frequency (BAF) is the proportion of hybridized sample that carries the B allele as designated by the Infinium assay. In a normal sample, discrete BAFs of 0.0, 0.5, and 1.0 are expected for each locus (representing AA, AB, and BB).

该软件包的设计思路及流程如下:
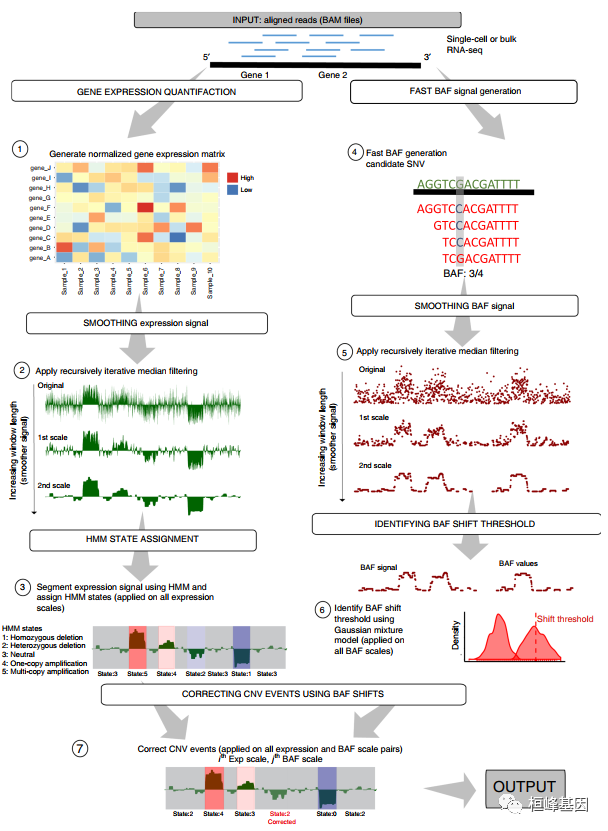
软件包安装
软件包 CaSpER 安装非常简单,直接安装即可:
install.packages("remotes")
remotes::install_github("akdess/CaSpER")数据读取
CaSpER 的输入由对齐的 RNA-Seq reads 和规范化的表达矩阵组成。CaSpER 使用来自 RNA-Seq reads的表达值和 B-allele frequencies (BAF)来估计CNV事件。
基因表达数据
表达值应该使用 TPM 或 FPKM 进行规范化。下面是一个规范化基因表达输入的例子:
library(CaSpER)
library(data.table)
library(knitr)
data <- yale_meningioma$data
loh <- yale_meningioma$loh
loh.name.mapping <- yale_meningioma$loh.name.mapping
control.sample.ids <- yale_meningioma$control.sample.ids
genoMat <- yale_meningioma$genoMat
samps <- yale_meningioma$samps基因组注释文件
归一化基因表达矩阵行表示具有 Ensembl 基因id 或 hgnc 基因符号的基因。列表示样本或细胞。细胞带信息可从UCSC下载。
# http://hgdownload.cse.ucsc.edu/goldenPath/hg19/database/cytoBand.txt.gz
data(hg19_cytoband)
kable(cytoband[1:5, ])注释数据包含基因组中每个基因沿每条染色体的位置,可以使用以下功能生成:
annotation <- generateAnnotation(id_type = "ensembl_gene_id", genes = rownames(yale_meningioma$data),ishg19 = T, centromere)
kable(annotation[1:5, ])SNP文件
使用以下函数读取 BAFExtract 输出文件:
loh <- readBAFExtractOutput(path = "./meningioma_baf\\", sequencing.type = "bulk")
names(loh) <- gsub(".snp", "", names(loh))实例操作
识别CNV事件
用来识别CNV事件的主要函数是CreateCasperObject()和runCaSpER(casper_object)。
创建casper对象
casper对象是执行单细胞和大量RNA-Seq的CNV分析所必需的。Casper对象是使用以下函数创建的:
object <- CreateCasperObject(raw.data = data, loh.name.mapping = loh.name.mapping,sequencing.type = "bulk", cnv.scale = 3, loh.scale = 3, matrix.type = "normalized",expr.cutoff = 4.5, annotation = annotation, method = "iterative", loh = loh,filter = "median", control.sample.ids = control.sample.ids, cytoband = cytoband)kable(yale_meningioma$loh.name.mapping[1:5, ])BAF和表达信号的两两比较
在创建casper对象后,对表达信号的所有尺度执行HMM,并将HMM分段状态与等位基因频移信息相结合。CaSpER算法使用表达式信号和BAF信号分解的所有成对比较来输出CNV调用。
## runCaSpER
final.objects <- runCaSpER(object, removeCentromere = T, cytoband = cytoband, method = "iterative")
## sample plot orders
order.sampleNames <- c("MN-1171", "MN-60835", "MN-1236", "MN-1237", "MN-1137", "MN-1161","MN-60", "MN-5")多个BAF和表达信号成对比较的CNV识别的方法
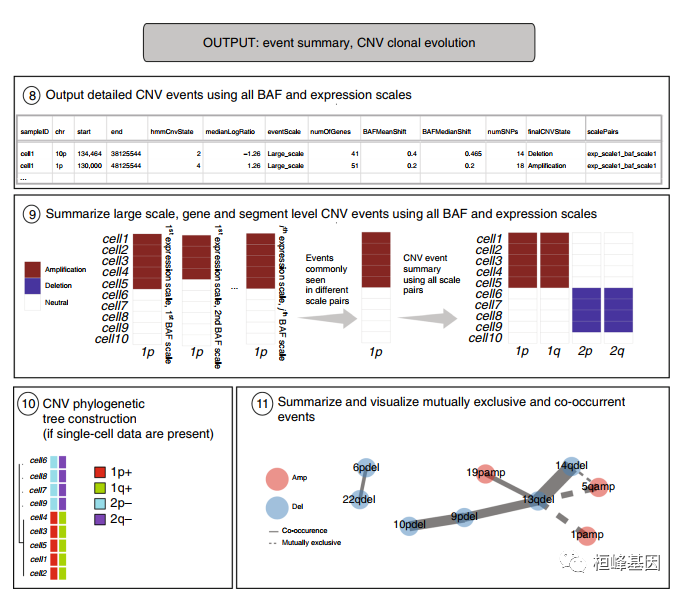
CNV 识别的两两比较和分配产生了大量的信号信息,必须对这些信息进行总结,以便为基因组的每个位置分配关于其 CNV 状态的结果,即amp/del/neutral 。我们使用基于一致性的方法来协调两两比较: 将事件放在一起,如果 CNV 调用在至少一定数量的两两规模比较中是一致的,我们就为一个基因或大型事件分配最终的CNV。CaSpER 通过将 CNV 调用分为大规模的、基于基因的和基于片段的CNV调用来协调和总结CNV调用
a. Large-Scale CNV Summarization.
为每条染色体臂分配了一个大规模的CNV调用,用于每个N×M两两规模比较。接下来,对于每个染色arm,识别大规模CNV调用在至少γ次N×M大规模CNV调用中是否一致。N为表达式信号最高平滑尺度的指标。M为BAF信号的最高平滑尺度指数。THR 代表最小百分比,75%(9个量表中至少7个),一致的CNV调用(在N×M表达量表和BAF量表的比较中),同时将最终的CNV (amp/del/neutral)调用分配给片段/基因/染色arm。
## large scale event summary
finalChrMat <- extractLargeScaleEvents(final.objects, thr = 0.75)
common <- intersect(order.sampleNames, intersect(rownames(finalChrMat), rownames(genoMat)))
finalChrMat <- finalChrMat[match(common, rownames(finalChrMat)), ]
genoMat <- genoMat[match(common, rownames(genoMat)), ]
## calculate TPR and FPR using genotyping array as gold standard
calcROC(chrMat = finalChrMat, chrMat2 = genoMat)b. Segment based CNV Summarization
基于片段的摘要旨在为通过尺度比较计算的最终片段集生成最终的CNV调用集。首先比较来自不同表达尺度的片段,生成一致的片段集。对于最终集合中的每个片段,如果在N×M CNV调用中有超过γ(默认=6)个一致的CNV调用,将一致的CNV调用分配给片段。当调用之间不存在一致性时,给segment分配一个中性的CNV状态。
library(GenomicRanges)
gamma <- 7
all.segments <- do.call(rbind, lapply(final.objects, function(x) x@segments))
segment.summary <- extractSegmentSummary(final.objects)
loss <- segment.summary$all.summary.loss
gain <- segment.summary$all.summary.gain
loh <- segment.summary$all.summary.lohloss.final <- loss[loss$count >= gamma, ]
gain.final <- gain[gain$count >= gamma, ]
loh.final <- loh[loh$count >= gamma, ]c. Gene based CNV Summarization
与大规模总结类似,生成一个矩阵,其中行是样本(细胞),列是基因。矩阵项0表示没有改变,1表示扩增,-1表示删除。如果一个变化在超过γ尺度的比较中是一致的(在N×M比较之外),报告该样本的变化事件。
all.summary <- rbind(loss.final, gain.final)
colnames(all.summary)[2:4] <- c("Chromosome", "Start", "End")
rna <- GRanges(seqnames = Rle(gsub("q", "", gsub("p", "", all.summary$Chromosome))),IRanges(all.summary$Start, all.summary$End))
ann.gr <- makeGRangesFromDataFrame(final.objects[[1]]@annotation.filt, keep.extra.columns = TRUE,seqnames.field = "Chr")
hits <- findOverlaps(rna, ann.gr)
genes <- splitByOverlap(ann.gr, rna, "GeneSymbol")
genes.ann <- lapply(genes, function(x) x[!(x == "")])
all.genes <- unique(final.objects[[1]]@annotation.filt[, 2])
all.samples <- unique(as.character(final.objects[[1]]@segments$ID))
rna.matrix <- gene.matrix(seg = all.summary, all.genes = all.genes, all.samples = all.samples,genes.ann = genes.ann)数据可视化
为了全面地可视化CNV事件,CaSpER首先生成结合所有样本的视觉效果,以便用户可以在所有样本上共同查看事件,并且可以直观地检查共现和互斥模式。为了表征这些模式,CaSpER对样本和检测到的CNVs进行聚类,并生成聚类图。我们在下面总结了这些可视化效果。
A. plotHeatmap
不同平滑尺度下全基因组基因表达信号图的可视化CaSpER输出不同尺度下的表达信号。在这些图中,每一行是一个样本,每一列是染色体。这些对于使用表达信号的全景检查来比较来自不同尺度的输出是有用的。
obj <- final.objects[[9]]
plotHeatmap(object = obj, fileName = "heatmap.png", cnv.scale = 3, cluster_cols = F,cluster_rows = T, show_rownames = T, only_soi = T)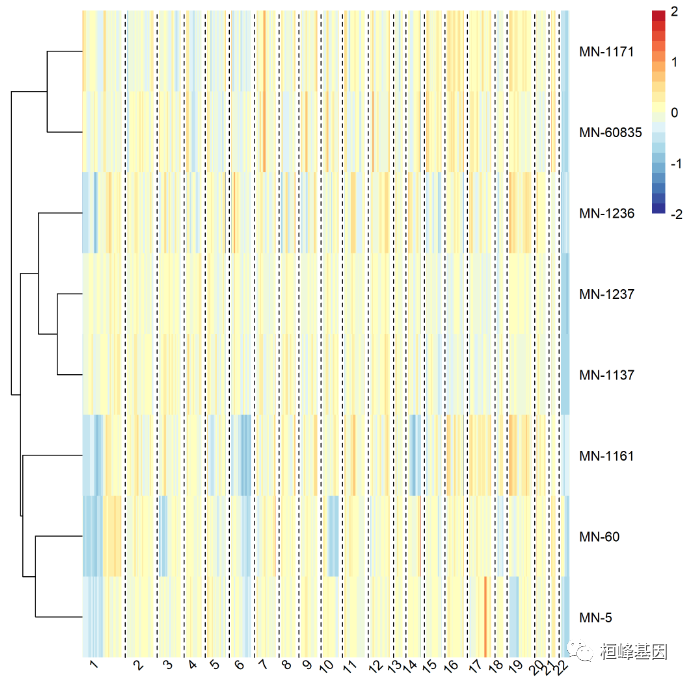
B. plotLargeScaleEvent
所有样本/细胞间大规模CNV事件的可视化。大尺度事件摘要用于总结多个样本上检测到的大尺度CNV事件(删除和扩增)。该图总结了大尺度的CNVs,并可能揭示在较小尺度上可视化数据时可能遗漏的模式。
## plot large scale events
plotLargeScaleEvent(object = obj, fileName = "large.scale.events.pdf")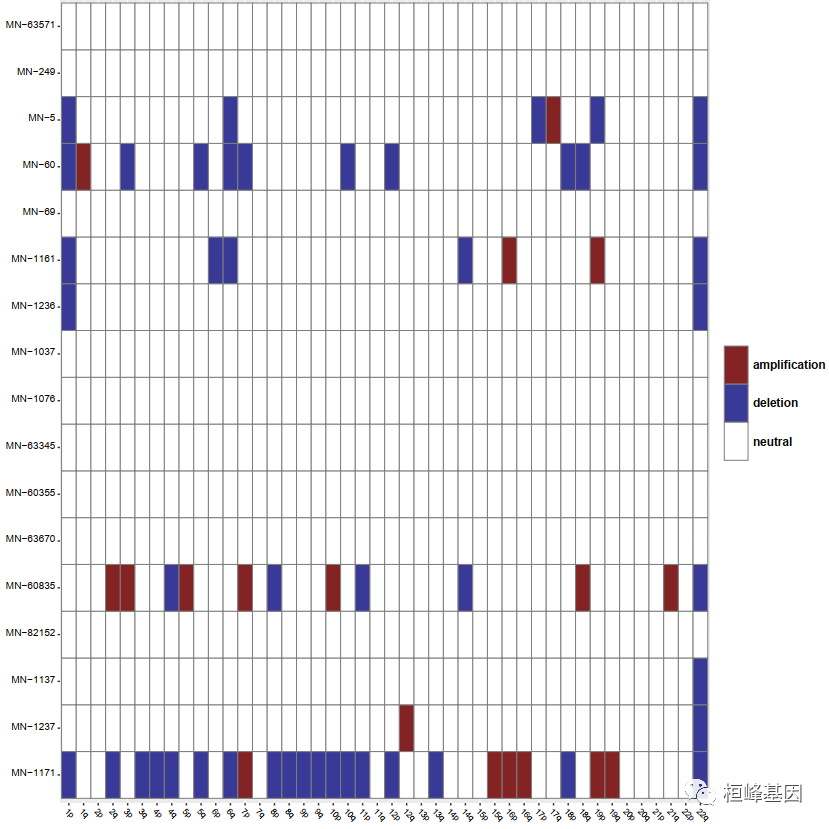
C. plotGEAndGT
绘制从基因分型和RNA-Seq中调用的大规模事件(仅适用于小样本量)
## plot large scale events called from genotyping and rna-seq (can be used only
## with small sample size)
plotGEAndGT(chrMat = finalChrMat, genoMat = genoMat, fileName = "RNASeqAndGT.png")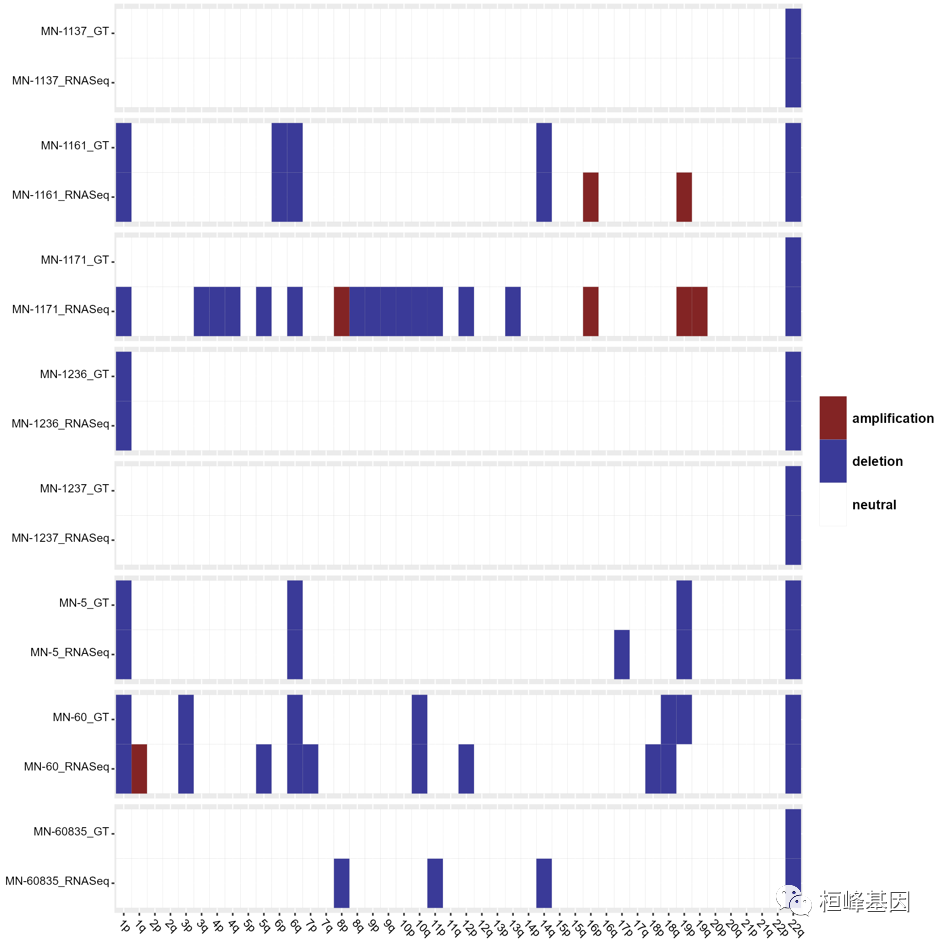
D. plotBAFAllSamples
所有样品的BAF移位信号的可视化。检测BAF移位信号对于分析CNV和LOH事件非常有用,特别是当与表达信号进行比较时。BAF移位图显示了BAF移位信号,使得每一行都是全基因组的BAF移位信号谱。
## plot BAF deviation for all samples together in one plot (can be used only
## with small sample size)
plotBAFAllSamples(loh = obj@loh.median.filtered.data, fileName = "LOHAllSamples.png")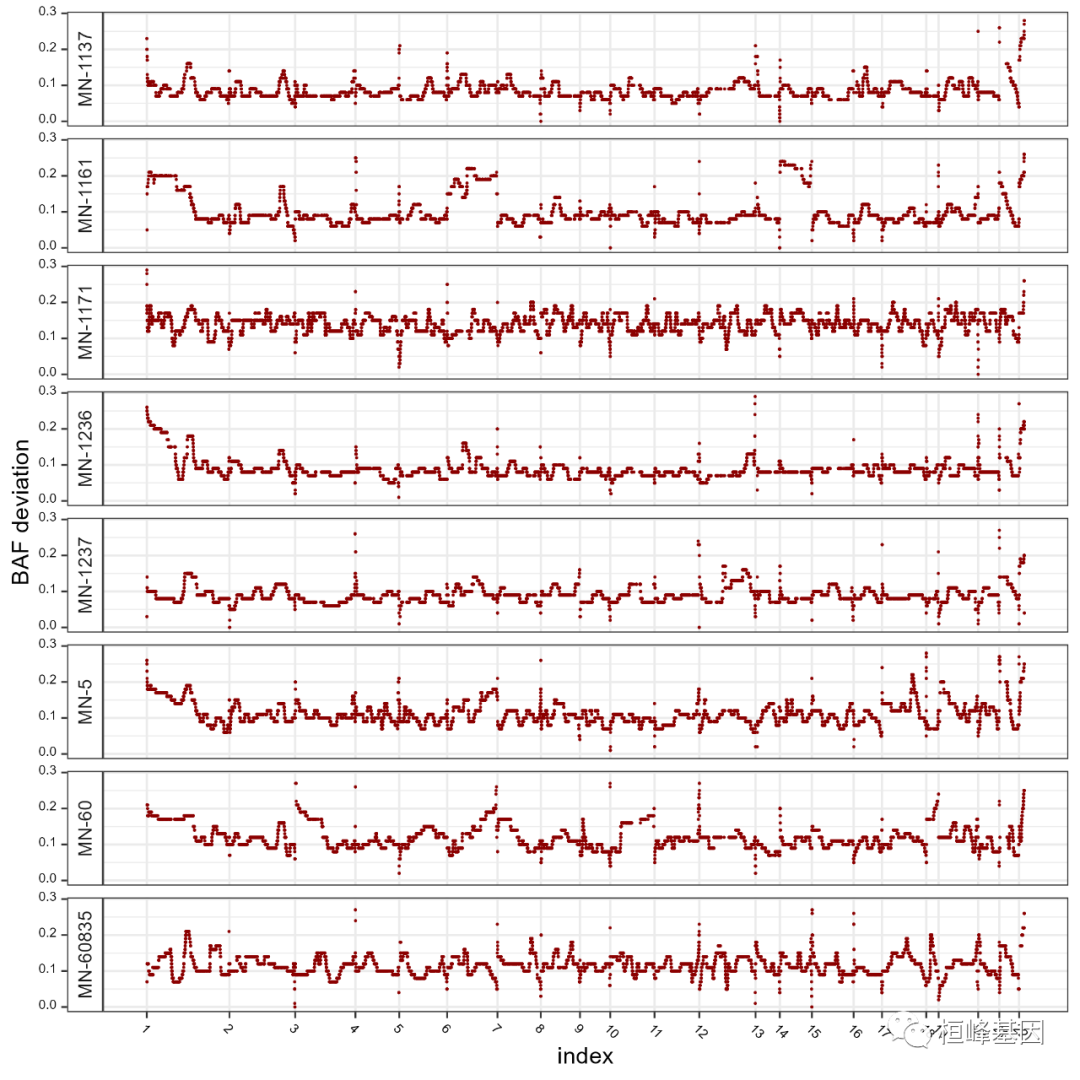
E. plotBAFOneSample
一个样本在不同尺度下BAF移位信号的可视化。该选项绘制了一个样本在不同尺度下的BAF移位信号。类似于表达信号的多尺度平滑,该信息可以全景评估和识别CNV和LOH事件。
## plot BAF signal in different scales for all samples
plotBAFOneSample(object, fileName = "LohPlotsAllScales.pdf")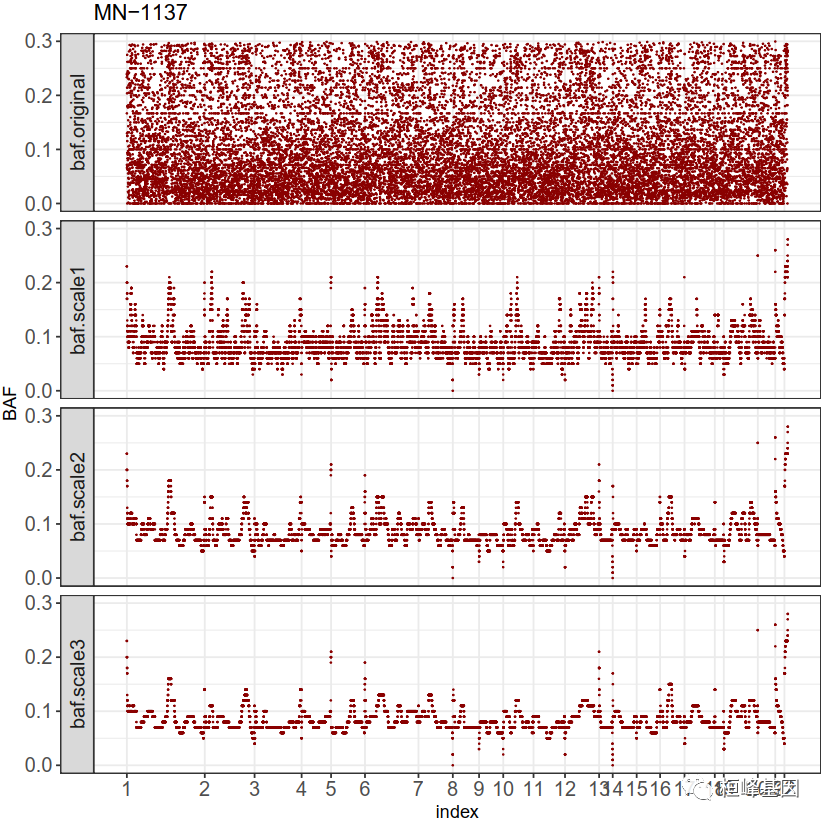
F. plotBAFInSeperatePages
在单独的页面上显示每个样品的BAF偏差。该选项分别为每个样本创建BAF移位信号。这样,用户可以可视化每个样本本身。
plotBAFInSeperatePages(loh = obj@loh.median.filtered.data, folderName = "LOHPlots")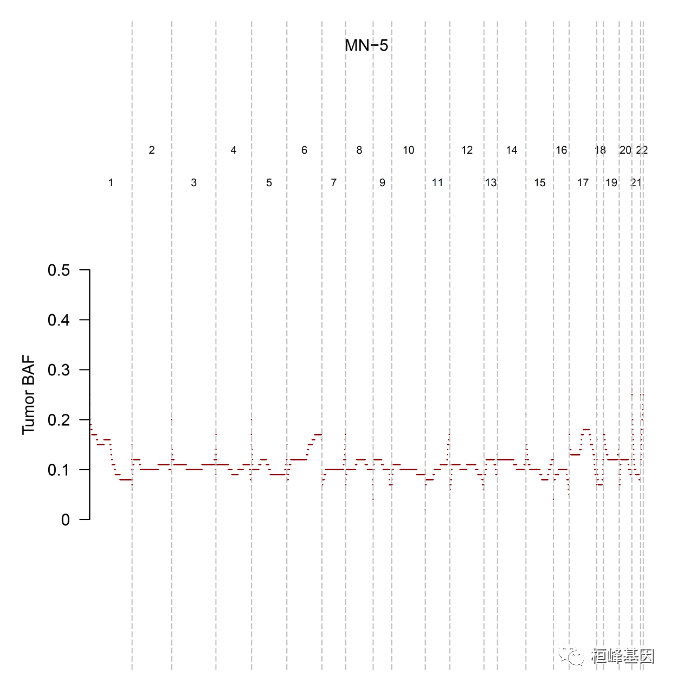
G. plotGEAndBAFOneSample
一个样地中一个样品的基因表达和BAF信号。该选项同时生成基因表达信号和BAF移位信号的可视化,以便用户可以在一个页面中共同评估。
plotGEAndBAFOneSample(object = obj, cnv.scale = 3, loh.scale = 3, sample = "MN-5")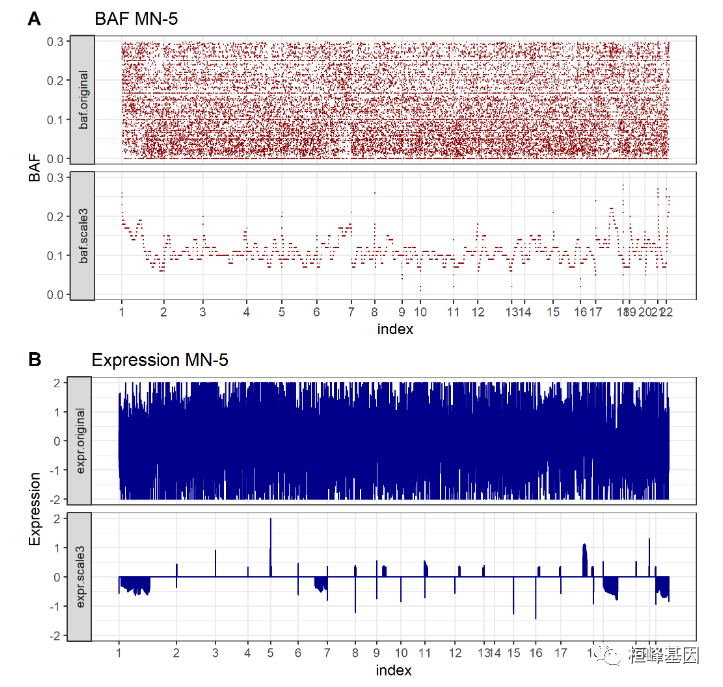
Reference
Serin Harmanci A, Harmanci AO, Zhou X. CaSpER identifies and visualizes CNV events by integrative analysis of single-cell or bulk RNA-sequencing data. Nat Commun. 2020;11(1):89. Published 2020 Jan 3. doi:10.1038/s41467-019-13779-x
这个工具是不是非常好用?
学会了轻松拿捏数据分析,顿时觉得自己也行了,关注桓峰基因公众号,轻松学生信,高效写文章!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!
http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/








每天10个小知识点)

)



)





