AutoFragDiff 是一个基于片段的,自回归的,口袋条件下的,3D分子生成扩散模型。
AutoFragDiff方法来源于文章《Autoregressive fragment-based diffusion for pocket-aware ligand design》,由加州大学的Mahdi Ghorbani等人于2023年11月3日发表在OpenReview,是NeurIPS2023 Poster文章。
其中,有两名作者来自罗氏。
文章链接为:Autoregressive fragment-based diffusion for pocket-aware ligand design | OpenReview。

一、AutoFragDiff 模型介绍
传统的,基于口袋的3D分子生成网络的常用方法是自回归模式,模型放置原子和原子键是迭代的,逐个进行。
但是这种方式会导致误差的积累,同时生成速度较慢,生成苯环分子也需要6个步骤。而使用基于分子片段的自回归方法可以避免这个问题。
作者使用使用几何矢量感知器和自回归扩散模型 Autoregressive Diffusion Models (ARDMs),以自回归的模式,分子骨架和蛋白质口袋为条件下,逐个预测新分子片段的原子类型和空间坐标。
在这一过程中,AutoFragDiff允许每个片段经历去噪过程,预测原子坐标和原子类型。该方法改善了生成的3D 分子的局部几何形状,同时保持与蛋白质靶标的高预测结合亲和力。 如下示意图:

此外,AutoFragDiff不是依赖固定的片段库,而是动态生成片段,为生成的片段多样性提供了灵活性。
该模型可以从用户提供的起始分子骨架进行支架延伸,即适用于骨架生长/延伸任务,既可以使用于口袋限制下,也可以使用在没有口袋限制的情况。即,AutoFragDiff可以依据提供的分子片段,在有口袋和没有口袋的条件下,进行分子扩展生成完整分子。
二、模型性能
作者统计了AutoFragDiff生成分子中环的键角与二面角分布与CrossDock数据集中分布的Jensen-Shannon Divergence(JSD,该值越小越好),结果显示,AutoFragDiff生成环与真实数据的更像,生成的分子结构更真实,要优于其他模型。如下表:

除了环状的原子键,作者在SI中也提供了其他原子键的JSD相似度结果,如下表。
从表中看,TargetDiff的结果更好一些,但是AutoFragDiff和TargetDiff都优于3DSBDD和Pocket2Mol。

作者同时比较了AutoFragDiff模型与3D-SBDD,Pocket2Mol等模型的Vina score, 多样性,可合成性和成药性指标,如下图。
其中,AutoFragDiff生成分子的Vina score要优于其他对比模型,但是成药性,可合成性,多样性指标上,弱于其他对比模型。

然后作者给了一个具体的例子,1a2g,如下图。感觉生成的分子还行。对比原有的骨架,Vina score也下降很多。

由于是会议的poster文章,作者给出的结果并不多。在SI中,提供了生成分子与口袋原子“碰撞”个数,strain energy,和相互作用原子的信息。
AutoFragDiff 分子与口袋原子的平均冲突为 6.7 次,优于其他基于扩散的模型(TargetDiff 9.2 和 DiffSBDD 平均 11.8)。
非扩散模型 Pocket2Mol 和 3DSBDD 每个分子平均有 5.7 次和 3.9 次冲突,而 CrossDock 数据集的测试集平均有 4.8 次冲突, 如下图:

同样,非扩散模型Pocket2Mol 和 3DSBDD 通常生成比基于扩散的模型具有更低应变能的分子,构象更加合理,如下图。

考虑到相互作用类型,TargetDiff 分子具有最多的氢键供体和受体,而 AutoFragDiff 和 TargetDiff 都显示出最多的疏水性和范德华相互作用,与 CrossDock 测试集分子相当,如下图。

三、模型评测
3.1 环境安装
作者提供了github 链接:
GitHub - keiserlab/autofragdiff
复制代码:
git clone https://github.com/keiserlab/autofragdiff.git项目目录结构:
.
├── LICENSE
├── README.md
├── __init__.py
├── analysis
├── analyze_generated_pocket_mols.py
├── analyze_scaffolds_generated.py
├── assets
├── data
├── extend_scaffold_crossdock.py
├── fpscores.pkl.gz
├── generate_pocket_molecules.py
├── notebooks
├── sample_crossdock_mols.py
├── sample_from_pocket.py
├── sampling
├── src
├── train_anchor_predictor.py
├── train_frag_diffuser.py
└── utils8 directories, 12 files环境安装
conda create -n AutoFragDiff python=3.10
conda activate AutoFragDiff 选择python=3.10避免pytroch_lightning的版本兼容问题。
安装 rdkit, biopython等。
pip3 install rdkit
conda install -c conda-forge openbabel
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidiapip install biopython==1.75
pip install biopandas
pip install networkx
pip install py3dmol
pip install scikit-learn
pip install tensorboard
pip install wandb
pip install tqdm
conda install pytorch-lightning# 以下是pytorch_lightning的参考模块
conda install pytorch-lightning=1.6 -c conda-forge
conda install -c conda-forge packaging
pip install tqdm
pip install pyyaml
pip install protobuf
pip install absl-pyconda install pyg -c pyg
pip install meeko
conda install -c bioconda vina
Meeko 读取 RDKit 分子对象并写入 PDBQT 字符串,将对接输出转换为 RDKit 分子和 SD 文件,而不会丢失键序,在python中经常用于Vina对接的预处理和结果处理。
安装QuickVina2需要另外创建一个环境MGLTools:
wget https://github.com/QVina/qvina/raw/master/bin/qvina2.1
chmod +x qvina2.1 conda create -n MGLTools -c bioconda mgltools安装AutoDockTool
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3安装fpocket,在生成分子时用于确定口袋:
conda config --add channels conda-forge
conda install fpocket注意,在macOS下面mgltools源和fpocket没有符合的包,不适配macOS系统。。。因此,建议本项目使用ubuntu系统测试。
以下均为ubuntu的测试结果。另外,这个项目由于提供的是pip安装方式,并且没有明确的版本信息,pyg,torch等安装包是相互之间有版本依赖关系的,因此安装起来会困难一些,多尝试就好。
3.2 下载作者训练好的checkpoint
作者训练完成的checkpoint文件下载
作者提供了训练好的模型checkpoint,链接为:https://drive.google.com/drive/folders/1DQwIfibHIoFPGJP6aHBGiYRp87bCZFA0

将两个checkpoint都下载,然后放置在./目录下。
3.3 生成分子测试 - 2z3h
作者提供了一个notebook形式的,里面展示了crossdocked_pocket10第一个体系,即2z3h,的基于口袋的分子生成,文件名为sample_for_pocket.ipynb。
sample_for_pocket.ipynb放置在./notebook文件夹内吗。因此,首先将工作目录切换到./notebook内。
测试体系2z3h.pdb,我已经提前下载到文件内,其口袋附近结构如下图(在晶体中,原口袋内有一个小分子BLO,我这里一并放上去了,只作为展示用途,作者提供的案例中并没有):

首先使用fpcoket寻找口袋,在终端运行:
fpocket -f 2z3h.pdb由于这里的2z3h.pdb只包含了口袋,因此,找到的口袋数量不多,一个是小分BEO所在的位置,序号为1,一个在其背后,序号为2,如下图:

对口袋进行加H,生成2z3h_H.pdb,终端运行如下命令:
reduce -Quiet -NOFLIP 2z3h.pdb > 2z3h_H.pdb注:我这的reduce是之前安装的AmberTools23。如果没有安装过,需要安装reduce或者AmberTools23。当然,也可以另外使用pymol等工具加氢以后保存成2z3h_H.pdb。
以下为sample_for_pocket.ipynb里面的内容。
首先导入相关模块:
import numpy as np
import pandas as pd
import os
from tqdm import tqdm
from itertools import combinationsfrom rdkit import Chem
import torch
import time
import shutil
from pathlib import Path
import torch.nn.functional as F
import syssys.path.append('../')from utils.volume_sampling import sample_discrete_number
from utils.volume_sampling import remove_output_files, run_fpocket, extract_values
from utils.templates import get_one_hot, get_pocket
from utils.templates import add_hydrogens, extract_hydrogen_coordinatesfrom src.lightning_anchor_gnn import AnchorGNN_pl
from src.lightning import AR_DDPM
from src.const import prot_mol_lj_rm, CROSSDOCK_LJ_RM
from src.noise import cosine_beta_schedule
from scipy.spatial import distance
from Bio.PDB import PDBParser
from Bio.PDB.Polypeptide import is_aa, three_to_onefrom analysis.reconstruct_mol import reconstruct_from_generated
from analysis.vina_docking import VinaDockingTaskfrom rdkit.Chem import rdmolfiles
from sampling.sample_mols import generate_mols_for_pocketfrom openbabel import openbabel
import tempfilefrom utils.volume_sampling import extract_alpha_spheres_coords
from utils.visuals import write_xyz_file, visualize_3d_pocket_molecule, get_pocket_mol
设定原子,电荷,氨基酸的索引字典,还有就是原子的范德瓦尔斯半径。
atom_dict = {'C': 0, 'N': 1, 'O': 2, 'S': 3, 'B': 4, 'Br': 5, 'Cl': 6, 'P': 7, 'I': 8, 'F': 9}
idx2atom = {0:'C', 1:'N', 2:'O', 3:'S', 4:'B', 5:'Br', 6:'Cl', 7:'P', 8:'I', 9:'F'}
CROSSDOCK_CHARGES = {'C': 6, 'O': 8, 'N': 7, 'F': 9, 'B':5, 'S': 16, 'Cl': 17, 'Br': 35, 'I': 53, 'P': 15}
pocket_atom_dict = {'C': 0, 'N': 1, 'O': 2, 'S': 3} # only 4 atoms types for pocket
amino_acid_dict = {'A': 0, 'C': 1, 'D': 2, 'E': 3, 'F': 4, 'G': 5, 'H': 6, 'I': 7, 'K': 8, 'L': 9, 'M': 10, 'N': 11, 'P': 12, 'Q': 13, 'R': 14, 'S': 15, 'T': 16, 'V': 17, 'W': 18, 'Y': 19}
vdws = {'C': 1.7, 'N': 1.55, 'O': 1.52, 'S': 1.8, 'B': 1.92, 'Br': 1.85, 'Cl': 1.75, 'P': 1.8, 'I': 1.98, 'F': 1.47}定义一个从PDB文件提取口袋的函数get_pocket, 输出是pdb文件,以及原子编号的字典,即上述中的pocket_atom_dict ,提取口袋原子的坐标(pocket_coords)以及原子one-hot(pocket_one_hot)。
def get_pocket(pdbfile, pocket_atom_dict, remove_H=True, ca_only=False):pdb_struct = PDBParser(QUIET=True).get_structure('', pdbfile)# find interacting pocket residues based on distance cutoff# 输入文件已为口袋的PDB文件,无需进行距离判断,仅需提取氨基酸即可pocket_residues = []for residue in pdb_struct[0].get_residues():# 氨基酸坐标res_coords = np.array([a.get_coord() for a in residue.get_atoms()])# 氨基酸,仅使用标准氨基酸if is_aa(residue.get_resname(), standard=True):pocket_residues.append(residue)# 氨基酸排序和对应的氨基酸类型pocket_ids = [f'{res.parent.id}:{res.id[1]}' for res in pocket_residues]if ca_only:# 口袋仅使用CA原子表示try:pocket_one_hot = []pocket_coords = []for res in pocket_residues:for atom in res.get_atoms():if atom.name == 'CA':pocket_one_hot.append(np.eye(1, len(amino_acid_dict),amino_acid_dict[three_to_one(res.get_resname())]).squeeze())pocket_coords.append(atom.coord)pocket_one_hot = np.stack(pocket_one_hot)pocket_coords = np.stack(pocket_coords)except KeyError as e:raise KeyError(f'{e} not in amino acid dict ({pdbfile})')else: # 全原子full_atoms = np.concatenate([np.array([atom.element for atom in res.get_atoms()]) for res in pocket_residues], axis=0)full_coords = np.concatenate([np.array([atom.coord for atom in res.get_atoms()]) for res in pocket_residues], axis=0)full_atoms_names = np.concatenate([np.array([atom.get_id() for atom in res.get_atoms()]) for res in pocket_residues], axis=0)pocket_AA = np.concatenate([([three_to_one(atom.get_parent().get_resname()) for atom in res.get_atoms()]) for res in pocket_residues], axis=0)# removing Hs if presentif remove_H:# 删除H原子h_mask = full_atoms == 'H'full_atoms = full_atoms[~h_mask]pocket_coords = full_coords[~h_mask]full_atoms_names = full_atoms_names[~h_mask]pocket_AA = pocket_AA[~h_mask]try:pocket_one_hot = []for i in range(len(full_atoms)):a = full_atoms[i]aa = pocket_AA[i]atom_onehot = np.eye(1, len(pocket_atom_dict), pocket_atom_dict[a.capitalize()]).squeeze()amino_onehot = np.eye(1, len(amino_acid_dict), amino_acid_dict[aa.capitalize()]).squeeze()is_backbone = 1 if full_atoms_names[i].capitalize() in ['N','CA','C','O'] else 0# 关于pocket_one_hot使用了原子类型,氨基酸类型,是否是骨架三种信息pocket_one_hot.append(np.concatenate([atom_onehot, amino_onehot, (is_backbone,)]))pocket_one_hot = np.stack(pocket_one_hot)except KeyError as e:raise KeyError(f'{e} not in atom dict ({pdbfile})')pocket_one_hot = np.array(pocket_one_hot)return pocket_one_hot, pocket_coords定义输入的pdb名字以及运行设备
pdb = '2z3h.pdb'
dev = 'cuda:0' # cuda device 指定口袋(fpocket找到的序号为1的口袋,上文中canya色)
定义生成分子数
k = 1 # pocket number identified by fpocket
pqr_file = pdb[:-4] + '_out/pockets/pocket' + str(k) + '_vert.pqr'
n_samples = 20 # number of samples to generate 生成10个分子提取口袋原子坐标以及原子的one-hot,以及fpocket填充的球的坐标
pocket_onehot, pocket_coords = get_pocket(pdb, pocket_atom_dict, remove_H=True, ca_only=False)# use fpocket to identify the protein pocket
# NOTE: --------------------------
# fpocket can sometimes give you the wrong pocket, make sure to check the output and visualize the pockettry:if not os.path.exists(pdb[:-4] + '_out'):#shutil.rmtree(pdb[:-4] + '_out', ignore_errors=Trueprint('running fpocket...')run_fpocket(pdb)pqr_file = pdb[:-4] + '_out/pockets/pocket' + str(k) + '_vert.pqr'alpha_spheres = np.array(extract_alpha_spheres_coords(pqr_file))except:raise ValueError('fpocket failed!')创建口袋的grid, 分辨率为1.5A:
# --------------- make a grid box around the pocket ----------------
min_coords = pocket_coords.min(axis=0) - 2.5 #
max_coords = pocket_coords.max(axis=0) + 2.5# 每个球的半径都是1.5
x_range = slice(min_coords[0], max_coords[0] + 1, 1.5) # spheres of radius 1.5 (vdw radius of C)
y_range = slice(min_coords[1], max_coords[1] + 1, 1.5)
z_range = slice(min_coords[2], max_coords[2] + 1, 1.5)grid = np.mgrid[x_range, y_range, z_range]
grid_points = grid.reshape(3, -1).T # This transposes the grid to a list of coordinates提取fpocket生成的α球的坐标:
# make sure the pocket-number is correct and you identified the correct pocket
pqr_file = pdb[:-4] + '_out/pockets/pocket' + str(k) + '_vert.pqr'
# 提取fpocket填充的α球的坐标
alpha_spheres = np.array(extract_alpha_spheres_coords(pqr_file))计算小分子生长空间的grid,利用fpocket的α球的3A范围内,且不得与口袋的grid距离小于2A。
distances_spheres = distance.cdist(grid_points, alpha_spheres)
mask_spheres = (distances_spheres < 3).any(axis=1)
filtered_alpha_points = grid_points[mask_spheres]# remove grid points that are close to the pocket
# 删除小分子空间中距离口袋太近的格点,阈值为2A。
pocket_distances = distance.cdist(filtered_alpha_points, pocket_coords)
mask_pocket = (pocket_distances < 2).any(axis=1)
grids = filtered_alpha_points[~mask_pocket]# 小分子生长空间的格点
grids = torch.tensor(grids)
口袋H原子,以及grid list
# 生长分子时,口袋加H,含有H原子
add_H = Trueif add_H:#add_hydrogens(pdb)prot_name_with_H = pdb[:-4] + '_H.pdb'# 提取H原子的坐标H_coords = extract_hydrogen_coordinates(prot_name_with_H)H_coords = torch.tensor(H_coords).float().to(dev)all_grids = [] # list of grids
all_H_coords = []
for i in range(n_samples):all_grids.append(grids) all_H_coords.append(H_coords)口袋大小,采样生成的每个分子的最大原子数:
pocket_vol = len(grids)
max_mol_sizes = []
# 采样每个分子最大原子数
for i in range(n_samples):max_mol_sizes.append(sample_discrete_number(pocket_vol))pocket_size = len(pocket_coords)max_mol_sizes = np.array(max_mol_sizes)
print('maximum molecule sizes', max_mol_sizes)定义一个LJ相互作用的调度器和参数,因为在分子生成过程中,使用到了LJ相互作用的引导,以避免分子与口袋的碰撞。
# scheduler and parameters for LJ guidance (avoiding clashes with pocket)
prot_mol_lj_rm = torch.tensor(prot_mol_lj_rm).to(dev)
mol_mol_lj_rm = torch.tensor(CROSSDOCK_LJ_RM).to(dev) / 100lj_weight_scheduler = cosine_beta_schedule(500, s=0.01, raise_to_power=2)
weights = 1 - lj_weight_scheduler
weights = np.clip(weights, a_min=0.1, a_max=1.)从口袋原子中采样anchor原子的序号,条件是:与fpocket中的α球的距离小于4.5A,距离最近的7个口袋原子中随机产生。
alpha_spheres_pocket_distances = distance.cdist(pocket_coords, alpha_spheres)
possible_pocket_anchors = np.argsort((alpha_spheres_pocket_distances < 4.5).sum(1))[::-1][:7]
pocket_anchors = np.random.choice(possible_pocket_anchors, size=n_samples, replace=True)先可视化一下口袋:
创建口袋的mol对象
pocket_mol = get_pocket_mol(pocket_coords, pocket_onehot)可视化口袋:
visualize_3d_pocket_molecule(pocket_mol, mol=None, spin=False, optimize_coords=False, sphere_positions2=alpha_spheres.tolist(), sphere_positions1=pocket_coords[possible_pocket_anchors].tolist())按照默认配置,口袋原子为灰色,anchor原子为绿色,fpocket产生的α球为黄色:

加载模型,加载口袋原子的坐标,one-hot。
注意, checkpoint要先下载完毕,然后放置在./目录上,不是当前的运行目录。
pocket_onehot = torch.tensor(pocket_onehot).float()
pocket_coords = torch.tensor(pocket_coords).float()model = AR_DDPM.load_from_checkpoint('../pocket-gvp.ckpt', device=dev)
model = model.to(dev)anchor_model = AnchorGNN_pl.load_from_checkpoint('../anchor-model.ckpt', device=dev)
anchor_model = anchor_model.to(dev)有了口袋原子坐标机器one-hot, 还有口袋anchor原子的one-hot,生成每个分子的最大原子数,以及口袋的grid范围,然后,可运行autodifffrag生成分子。
设定每个分子最多由8个片段组成。
# 运行autofragdiff最大生成8个片段
max_num_frags = 8
x, h, mol_masks = generate_mols_for_pocket(n_samples=n_samples,num_frags=max_num_frags,pocket_size=pocket_size,pocket_coords=pocket_coords,pocket_onehot=pocket_onehot,lig_coords=None,anchor_model=anchor_model,diff_model=model,device=dev,return_all=False,max_mol_sizes=max_mol_sizes,all_grids=all_grids,rejection_sampling=False,pocket_anchors=pocket_anchors,lj_guidance=True,prot_mol_lj_rm=prot_mol_lj_rm,mol_mol_lj_rm=mol_mol_lj_rm,all_H_coords=all_H_coords,guidance_weights=weights)将AutoFragDiff生成的原子坐标及原子one-hot转化为rdkit的mol对象
x = x.cpu().numpy()
h = h.cpu().numpy()
mol_masks = mol_masks.cpu().cpu().numpy()# convert to SDF
all_mols = []
for k in range(len(x)):mask = mol_masks[k]h_mol = h[k]x_mol = x[k][mask.astype(np.bool_)]atom_inds = h_mol[mask.astype(np.bool_)].argmax(axis=1)atom_types = [idx2atom[x] for x in atom_inds]atomic_nums = [CROSSDOCK_CHARGES[i] for i in atom_types]try:mol_rec = reconstruct_from_generated(x_mol.tolist(), atomic_nums)all_mols.append(mol_rec)except:continue查看生成的分子
visualize_3d_pocket_molecule(pocket_mol, mol=all_mols[0], spin=False, optimize_coords=False)输出:

将生成的分子rdkit对象保存成sdf文件,命名为generated_mols.sdf。
# 保存成sdf文件
sdf_path = './generated_mols.sdf'
writer = Chem.SDWriter(sdf_path)
for mol in all_mols:writer.write(mol)
writer.close()虽然定义让AutoFragDiff生成20个分子,但实际上只生成了19个。生成的19分子见如下视频:
上传审核中
这个体系在之前的DrugGPS测评的文章中,有测试过,DrugGPS生成的分子严重偏小,而AutoFragDiff很好的解决了这个问题。
3.4 生成分子测试-3WZE
接下来,对我们之前的3WZE体系进行测试。3WZE是一个激酶底物口袋,如下图。

采样与之前3.3类似的方法,生成分子如下视频:
上传审核中
生成分子的2D结构如下图:


参考分子如下图:

从上述结果来看部分分子还有有点类药,同时有的分子的位置很好位于参考分子的空间。生成分子的构象还是比较合理。
但是大量的分子不类药,同时存在很多C=O基团。此外,也存在明显的为完全占据口袋的情况。
3.5 命令行生成分子
注意,reduce的路径,要先设置好,否则会报错如下:
File "/home//AutoFragDiff/sampling/sample_mols.py", line 103, in generate_mols_for_pocket
all_H_coords[l] = all_H_coords[l] - anchor_pos[l]
RuntimeError: The size of tensor a (0) must match the size of tensor b (3) at non-singleton dimension 0
这是因为口袋没有加氢成功。
在输出信息中,会出现:sh: reduce: command not found
在utils.templates.py文件中,设置好reduce的路径。即,将add_Hydrogens函数修改为:(reduce的路径按照自己的环境设置)
def add_hydrogens(pdf_file):"""Add hydrogens to a PDB file using reduce."""#print('adding hydrogens')out_pdb = pdf_file[:-4] + '_H.pdb'os.system(f'~/anaconda3/envs/AmberTools23/bin/reduce -Quiet -NOFLIP {pdf_file} > {out_pdb}')然后再./目录上运行如下命令,为3WZE口袋生成分子。3WZE.pdb要先放置在./目录中:
python sample_from_pocket.py \--results-path results \--pdb 3WZE.pdb \--anchor-model \anchor-model.ckpt \--diff-model pocket-gvp.ckpt \--n-samples 10 \--device cuda:0 \--pocket-number 1生成输出信息如下:

生成的分子保存在./results路径中,sdf文件名为:pocket__mols.sdf。因为用的上面的3WZE体系,生成结果类似,就不重复展示了。
3.6 扩展分子骨架
作者提供了为crossdock扩展分子骨架的代码,在./目录下运行如下函数:(注:这一部分需要先下载好数据集)
python extend_scaffold_crossdock.py \--data-path ./data/CROSSDOCK \--results-path scaffold-gen \--anchor-model anchor-model.ckpt \--n-samples 20 \--exp-name scaffold-gen \--diff-model pocket-gvp.ckpt \--device cuda:0每个体系生成20个分子。运行输出示例:

每个体系生成的分子保存在.//scaffold-gen/scaffold-gen路径中,默认crossdock测试集中的每个体系生成20个分子。
这里,我选择测试集中的第二个体系:GLMU_STRPN_2_459_0, 口袋为4aaw_A_rec_4ac3_r83_lig_tt_min_0_pocket10.pdb, 原先的小分子为:4aaw_A_rec_4ac3_r83_lig_tt_min_0.sdf,如下图:

代码会自动识别骨架,然后,在骨架基础上进行拓展。拓展生成的20个分子的2D结构如下图:

大部分的骨架拓展都失败了,只成功扩展了4个分子。

而之前的参考分子为:
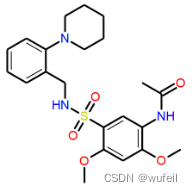
首先,分子扩展的范围不完整,成功率也比较低。扩展的程度未达到参考分子的状态。更多例子,大家可以在./scaffold-gen/scaffold-gen文件夹内查看。
四、测评总结
在口袋条件下,基于自回归扩散模型的3D分子生成方法很多,例如:TargetDiff,很常见。基于分子片段库的3D分子生成方法也有,例如: FLAG和DrugGPS。
基于分子片段的,3D分子生成扩散模型,比较少见,AutoFragDiff将基于分子片段和扩散模型结合起来了,允许分子库中每一个分子片段在采样过程中经历去噪过程。这一点还是比较有意思的,创新性十足。AutoFragDiff既不是传统扩散模型的直接(0,1)采样噪声也不是基于片段那样子预测是哪一个片段。
AutoFragDiff充分利用了扩散模型的强可扩展性与基于分子片段生成分子的高可成药性。但是,在文章展现的结果中,并没有那么理想,成药性上,还有SA打分上,并没有超越SBDD等模型,这一点值得思考。我们的案例测试结果也显示生成的分子比较奇怪。
感觉是分子库中每一个分子片段经历去噪过程的方式不对,只用在了初始化中,在后面的去噪过程中,这些分子片段的信息,被覆盖了,所以效果不佳。

)










 持续更新中)
)



)

