Syn_SegNet: A Joint Deep Neural Network for Ultrahigh-Field 7T MRI Synthesis and Hippocampal Subfield Segmentation in Routine 3T MRI
- Syn_SegNet:用于常规 3T MRI 中超高场 7T MRI 合成和海马亚场分割的联合深度神经网络
- 背景
- 贡献
- 实验
- 为了确定哪个模态分割最优做了个实验
- 消融实验
- 一个小数据集上的对比试验
- 方法
- 合成网络
- 分割网络
- 损失函数
- Thinking
Syn_SegNet:用于常规 3T MRI 中超高场 7T MRI 合成和海马亚场分割的联合深度神经网络
4866 IEEE JOURNAL OF BIOMEDICAL AND HEALTH INFORMATICS, VOL. 27, NO. 10, OCTOBER 2023
背景
3T 图像的信号对比度和分辨率有限,因此在常规 3T MRI 中自动分割这些子场具有挑战性,本文旨利用超高场 7T MRI 合成来改善 3T MRI 中的海马亚场分割。
贡献
- 首先,我们采用改进的Pix2PixGAN作为合成模型,结合自注意力模块、图像和特征匹配损失以及ROI损失,在海马区域周围生成高质量的7T样MRI。
- 其次,我们利用具有多尺度深度监督的 3D-UNet 变体作为分割子网络,结合利用先前解剖学知识的解剖加权交叉熵损失。
- 这项工作的主要贡献如下: • 我们提出了 Syn_SegNet,一种端到端的多任务深度神经网络,用于在常规 3T MRI 上进行 HS 分割,具有通用分辨率。该网络结合了合成和分割任务,通过合成类 7T MRI 来提高 HS 分割精度。
- 我们对 Pix2PixGAN 进行了修改,包括自注意力机制、图像和特征匹配损失以及聚焦海马体的 ROI 损失,以提高生成的 7T 类 MRI 的质量。
- 我们采用具有多尺度深度监控的 3D U-Net 变体进行 HS 分割,利用合成的 7T 数据和真实的 3T 数据。此外,还设计了解剖学加权交叉熵损失 (AWCloss) 函数,以利用海马体的先验解剖学知识,进一步提高分割性能。
- 通过对配对的 3T-7T MRI 数据集和公共 3T 数据集的消融研究和比较研究,对所提出的方法进行了广泛的验证。结果表明Syn_SegNet在 HS 分割中的有效性。
实验
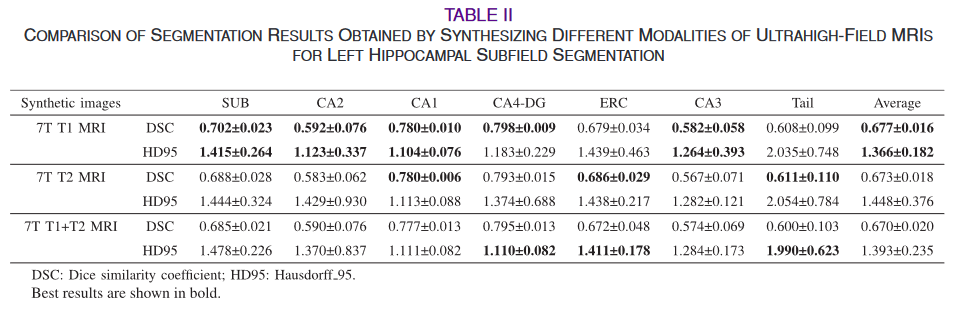
为了确定哪个模态分割最优做了个实验
为了确定最适合HS分割的合成MRI模式,我们比较了3种超高场MRI合成:7T T1 MRI、7T T2 MRI和7T T1 + T2 MRI (即同时合成7T T1 MRI和7T T2 MRI)。将合成的图像与原始输入( 3T T1 + T2 MRI)进行拼接,得到最终的分割结果。本实验是在左HS分割数据上进行的。合成7T T1 MRI获得了最高的平均DSC ( 0.677 )和最低的HD95 ( 1.366 ),在大多数HS分割(见表Ⅱ)中优于其他模态。定性地,与7T T2 MRI (见图8)相比,该网络显示了7T T1 MRI更好的细节恢复。基于这些发现,我们进行了7T T1 MRI合成,用于后续实验。
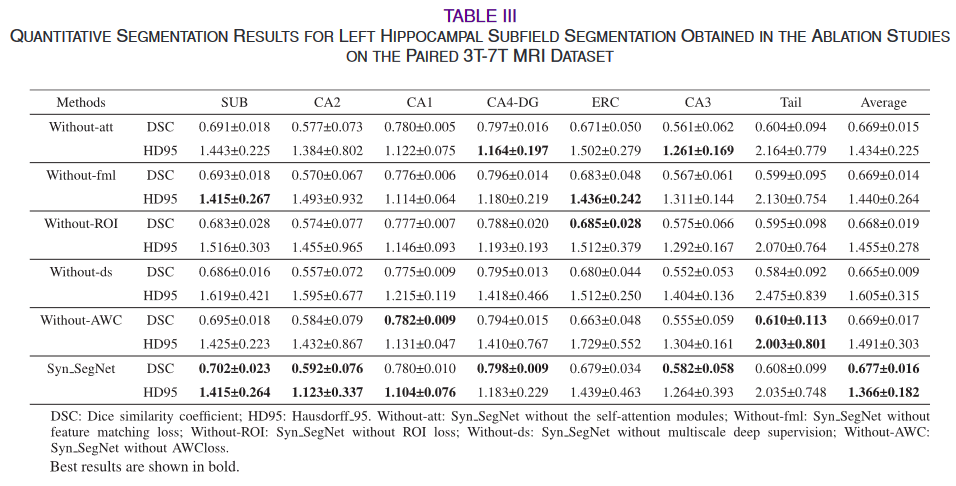
消融实验
AWCLoss涨了0.01,ds涨了0.02,RoiLoss涨了0.02,fml涨了0.01,self-att涨了0.01
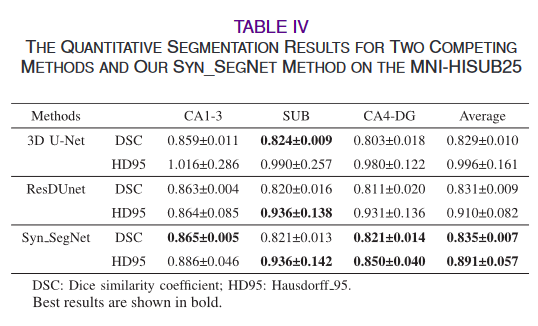
一个小数据集上的对比试验
合成、分割的方法涨了0.002
研究表明,将合成的7T MRI数据与常规3T MRI相结合,提高了HS分割的准确性。我们的研究结果突出了联合训练在合成和分割任务中的优越性。不同的任务之间可以互惠互利,从而提高下游任务的性能。ROI损失起着最重要的作用。通过重点保留ROI内重要的结构和纹理信息,ROI损失增强了合成的7T MRI的质量,从而提高了HS分割的准确性。
方法
输入是3T数据,合成7T数据
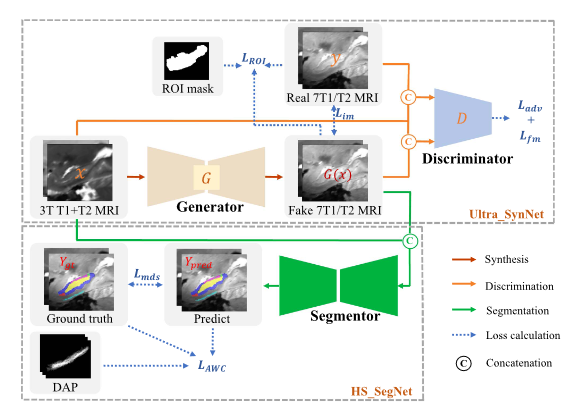
合成网络
简单的生成对抗网络,加了self-Attention

分割网络
3DUNet加上深度监督
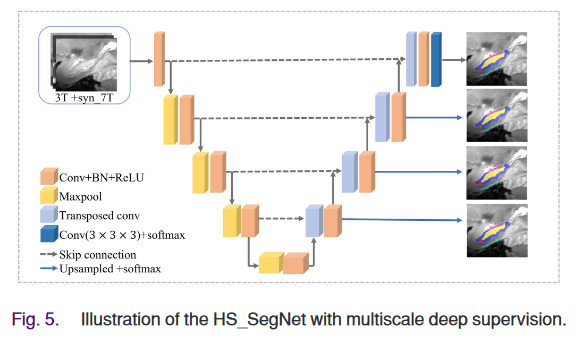
损失函数
确定Syn _ SegNet的最佳超参数值是一个挑战。在评估多个权重组合后,我们设置如下值:

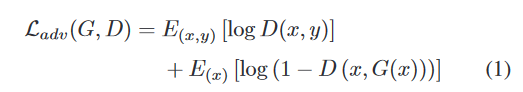
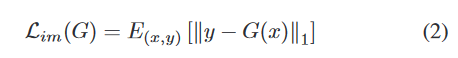
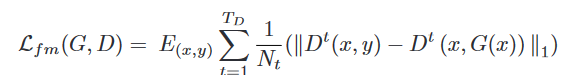
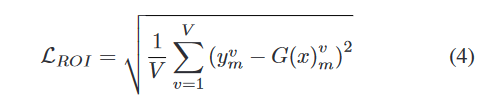
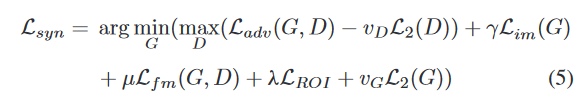
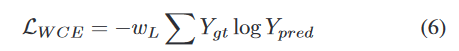
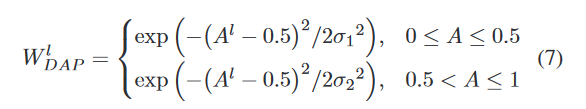
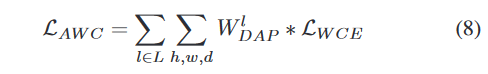
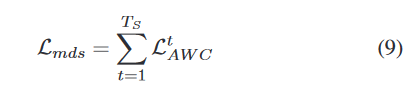
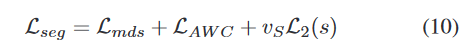
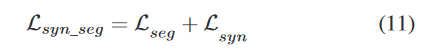
Thinking
损失函数多,生成对抗损失函数的权重是0.001,分割的权重是200,相差好大。为啥捏
此篇重要参考文献:
[36] Y. Huo et al., “Synseg-net: Synthetic segmentation without target modality ground truth,” IEEE Trans. Med. Imag., vol. 38, no. 4, pp. 1016–1025, Apr. 2019.
[41] L. Qu, Y. Zhang, S. Wang, P-T. Yap, and D. Shen, “Synthesized 7T MRI from 3T MRI via deep learning in spatial and wavelet domains,” Med. Image Anal., vol. 62, 2020, Art. no. 101663.
[42] Z. Huang, L. Lin, P. Cheng, L. Peng, and X. Tang, “Multi-modal brain tumor segmentation via missing modality synthesis and modality-level attention fusion,” 2022, arXiv:2203.04586.
[43] Z. Zhang, L. Yang, and Y. Zheng, “Translating and segmenting multimodal medical volumes with cycle-and shape-consistency generative adversarial network,” in Proc. IEEE Conf. Comput. Vis. Pattern Recognit., 2018, pp. 9242–9251.
 => m.Id - n.Id); id是double类型的为什么回报错)
:优选算法详解)

)











:ClickHouse 引擎详解)
)

