原标题:一款好用的进化树可视化编辑软件
iTOL(Interactive Tree Of Life)是一个在线显示和操作的进化树工具。我们可以添加很多自定义的项目,丰富和完善自己的进化树,比如添加柱状图、蛋白结构域、heatmap、基因平行转移(horizontal gene transfer)等。今天讲一下iTOL的具体操作步骤。
网址:http://itol2.embl.de/
打开网址后,iTOL的主界面是酱紫的:
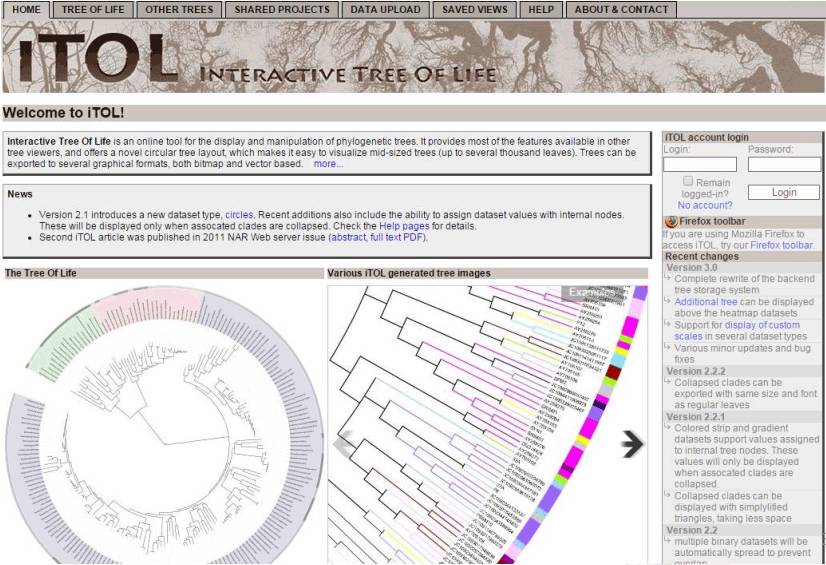
iTOL简介:
目前iTOL版本是3.0
必要工具:带有flash7以上的浏览器,建议用chrome、 firefox、 opera、 safari等,IE(强烈不建议)必须7.0以上版本
必须注册才能构建自己的工作目录
输入主文件:进化树文件(.nwk),最好带有bootstrap的文件
输入辅文件:有一定标准格式的其他文件(下文有示例)
iTOL菜单栏:
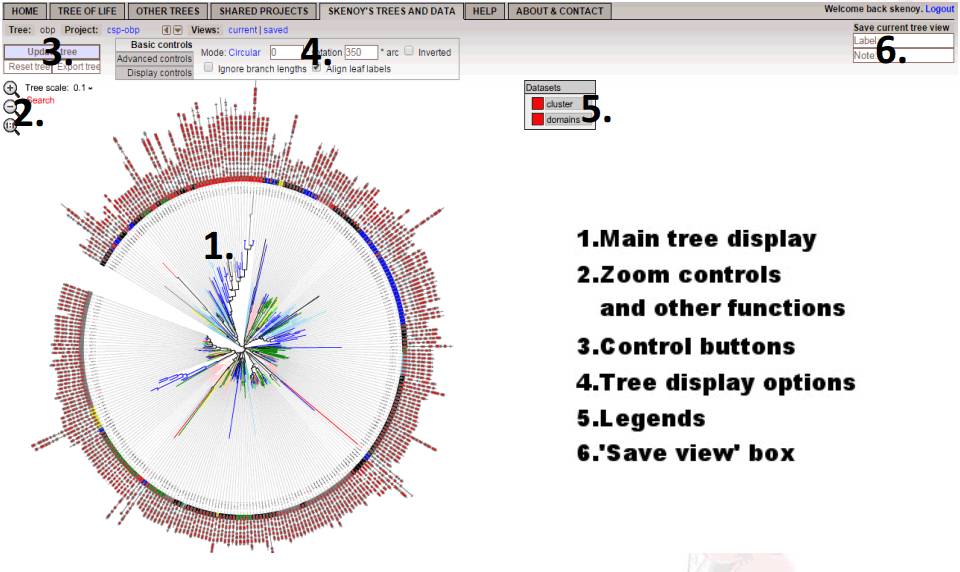
iTOL操作步骤:
注册 /登陆
新建工作区(workspace)
在工作区内新建任务(project)
在project中上传树文件
应用工具修图
注:一个workspace可以建多个project,每个project可以上传一个目的树文件。
1
注册 / 登陆
此处省略100字……
2
新建workspace
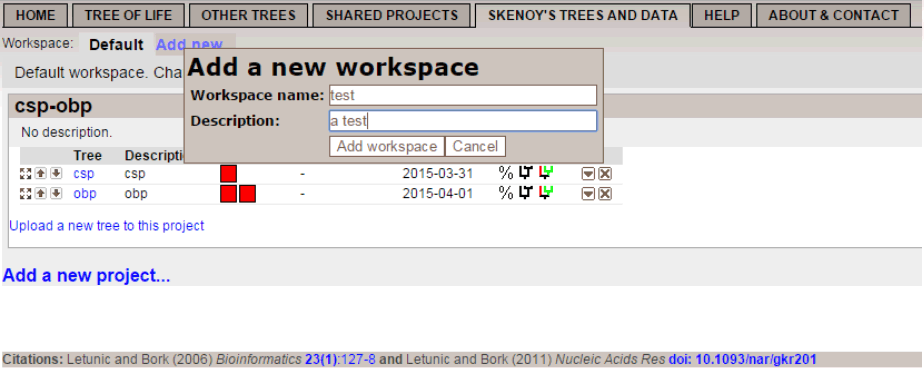
3
新建project
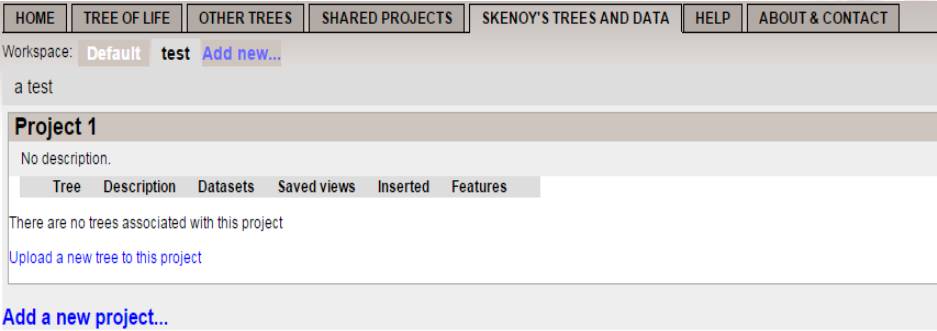
4
在project中上传树文件
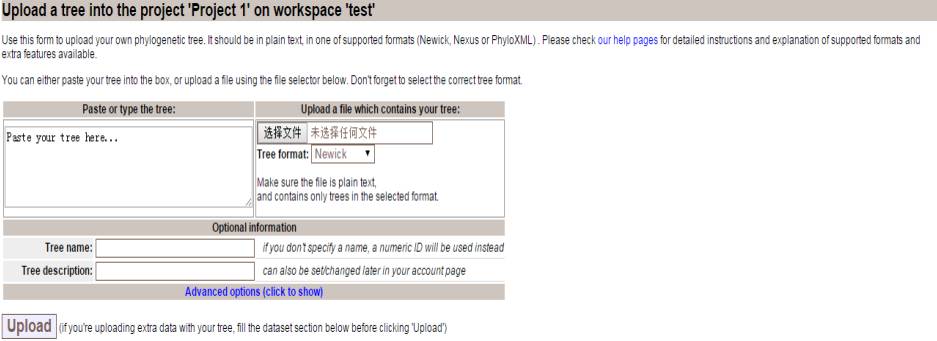
5
应用修图工具
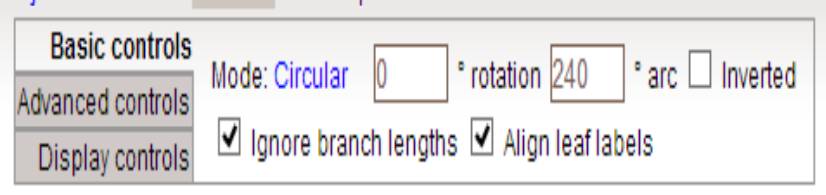
修改图的形状,字体大小和线条粗细:
添加背景颜色(Edit color):
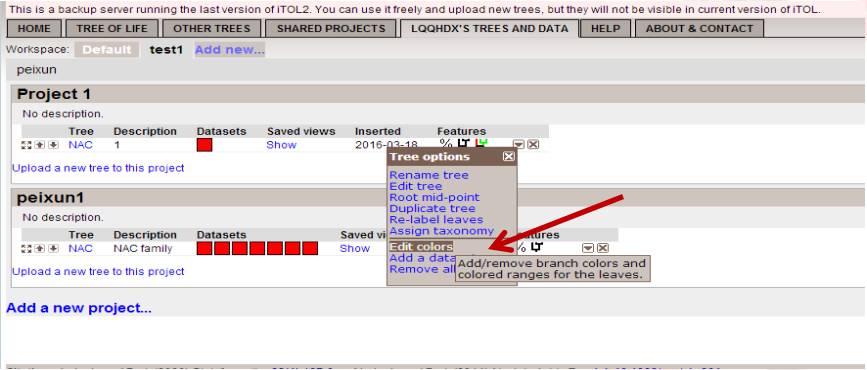
↓
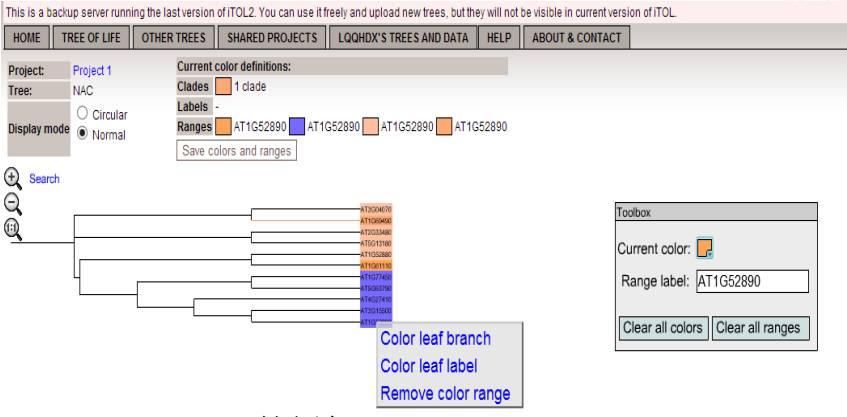
Leaf branch -----枝颜色
Leaf label-----基因ID颜色
Range----背景颜色
注:要使多个基因呈现同一种颜色,需要再Range label中随便输入一个图上的基因ID。
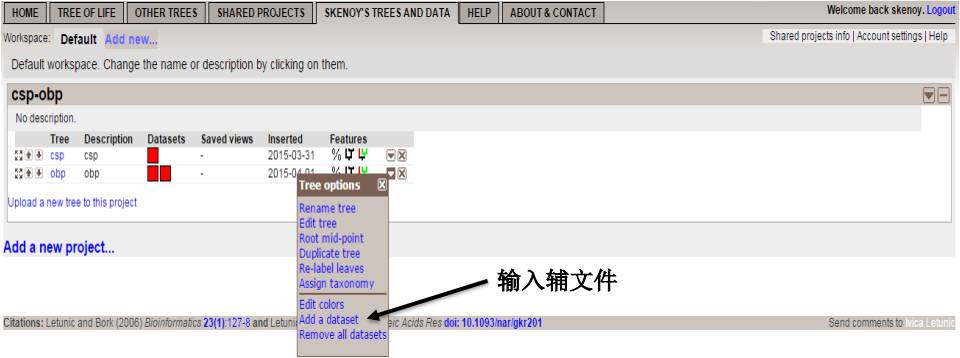
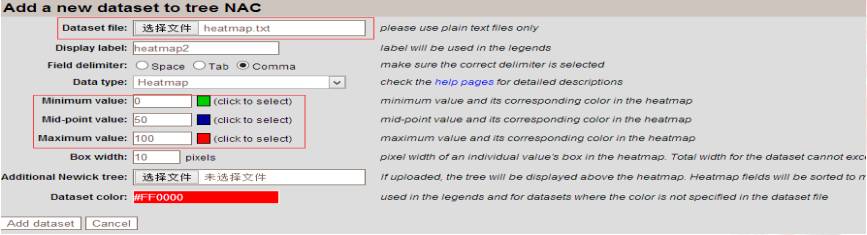
添加表达量:
↓
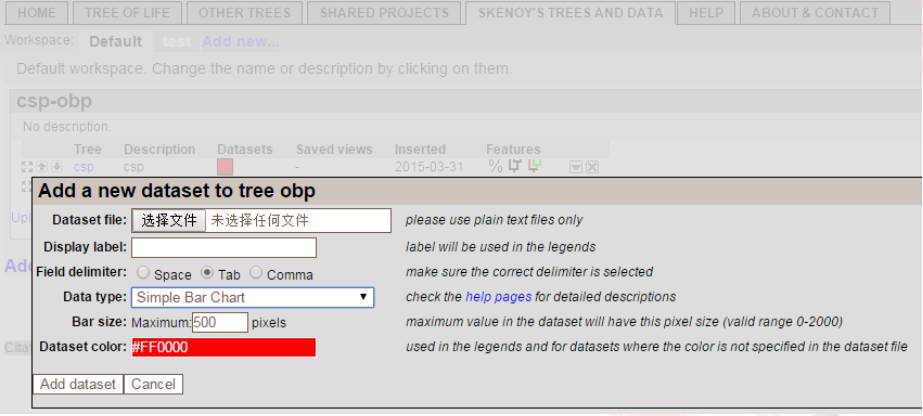
Field delimiter-----选择辅助文件间隔方式
Data type-----选择辅助文件展示方式
辅助文件格式:
LABELS,sample_1,sample_2,sample_3 样本名
COLORS,#ff0000,#00ff00,#0000ff 颜色
AT3G04070,10,20,30
AT1G69490,50,30,10
AT2G33480,20,40,10
AT5G13180,15,10,19 表达量
AT1G52880,19,18,16
AT1G61110,10,20,20
AT1G77450,40,50,50
AT5G63790,19,30,40
AT4G27410,20,30,15
AT3G15500,18,40,30
AT1G52890,10,40,10
↓

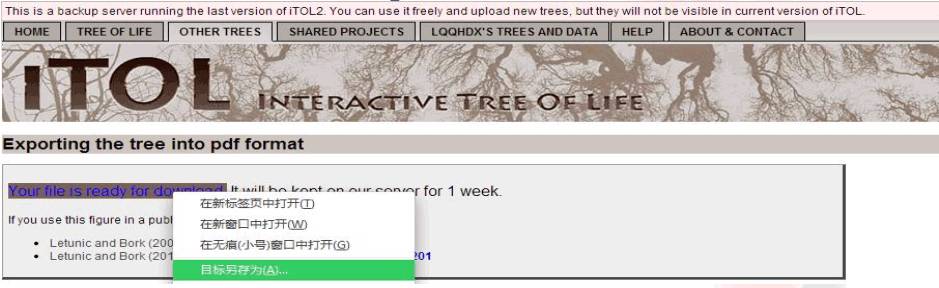
图形保存:
Export tree(选择输出格式输出文件)

↓
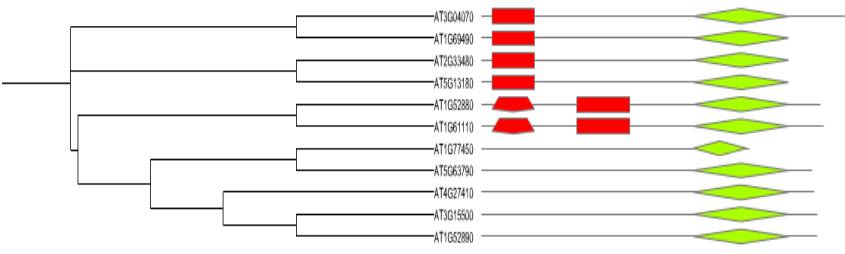
添加蛋白结构域:
AT3G04070,343,RE|10|50|#ff0000|DUF2,DI|200|290|#aaff00|DUF22
基因ID,基因长度,第一个结构域图形展示类型|结构域起始位置|结构域终止位置|图形颜色|结束,第二个结构域图形展示类型|起始位置|终止位置|颜色|结束

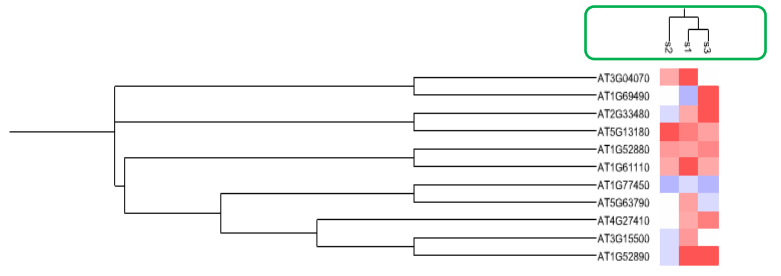
添加热图:
LABELS,s1,s2,s3
AT3G04070,10,20,30
AT1G69490,50,30,10
AT2G33480,20,40,10
AT5G13180,15,10,19
AT1G52880,19,18,16
AT1G61110,10,20,20
AT1G77450,40,50,50
AT5G63790,19,30,40
AT4G27410,20,30,15
AT3G15500,18,40,30
AT1G52890,10,40,10
↓
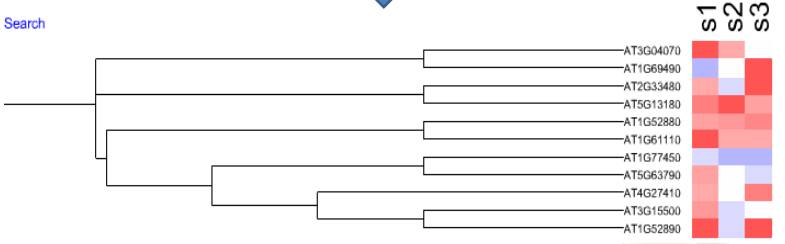
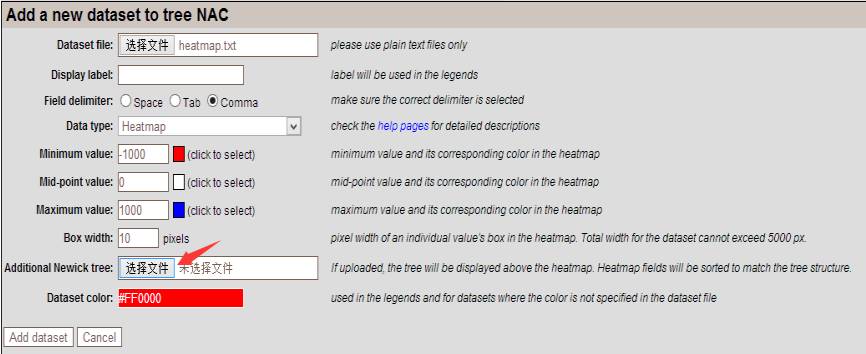
添加样本聚类树:
添加热图时选择添加树文件。文件格式: ((s1,s3),s2)。
大家可以将进化树提交给iTOL,然后按照自己想要的格式进行美化试试看。想学习更多画进化树的技巧,可以来参加基迪奥-华工暑期培训班()呀。好哒,今天就酱紫啦~~返回搜狐,查看更多
责任编辑:







开发实用指南)





如何在分类数据上训练模型?)





