1、下载
https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/

2、解压,配置环境变量
tar zvxf ncbi-blast-2.14.1+-x64-linux.tar.gz解压后改名为 blast配置环境变量,可以不配置 使用的时候直接绝对路径使用(本次使用绝对路径)
vim ~/.bashrc 将下面添加道最后, dir 是解压文件所在的绝对路径,export BLAST_HOME=/dir/blast
export PATH=$PATH:/dir/blast/bin使配置生效source ~/.bashrc测试安装结果blastn -version 如果显示版本信息则说明可正常使用
3、创建数据库
在/dir/blast 下创建文件夹 db/mydb 用来存放不同的数据库
构建数据库:initdb.fasta 是用来构建数据库的源文件
#绝对路径调用,直接使用makeblastdb命令调用可能出现找不到命令的问题,可能是环境变量没有配置好/dir/blast/bin/makeblastdb -in /dir/blast/db/mydb/initdb.fasta -dbtype prot结果:

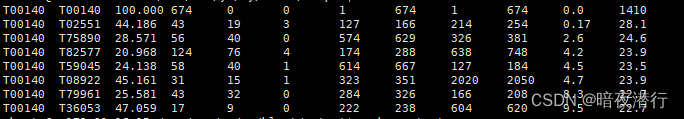
4、测试搜索 test.fasta 是测试序列文件
#采用绝对路径调用#结果输出到终端/dir/blast/bin/blastp -query /dir/blast/output/test.fasta -db /dir/blast/db/mydb/initdb.fasta -outfmt 6#结果输出到文件/dir/blast/bin/blastp -query /dir/blast/output/test.fasta -db /dir/blast/db/mydb/initdb.fasta -outfmt 6 -out /dir/blast/output/test结果:
5.在php中调用
//通过blast获取targetID,然后通过targetID获取详细信息
$input_sequence = $data_json['sequence']; // 从用户前端输入的序列
$file_name = uniqid() . ".fasta";
// 创建临时文件
$tempFile = tempnam("/tmp", $file_name);
//将用户输入的文件写入到临时文件,blast的输入条件只找到了文件输入,不知道可不可以其他形式输入
file_put_contents($tempFile, $input_sequence);
// 执行 BLAST 查询
$output = shell_exec("/dir/blast/bin/blastp -query " . $tempFile . " -db /dir/blast/db/mydb/initdb.fasta -outfmt 6");echo $output;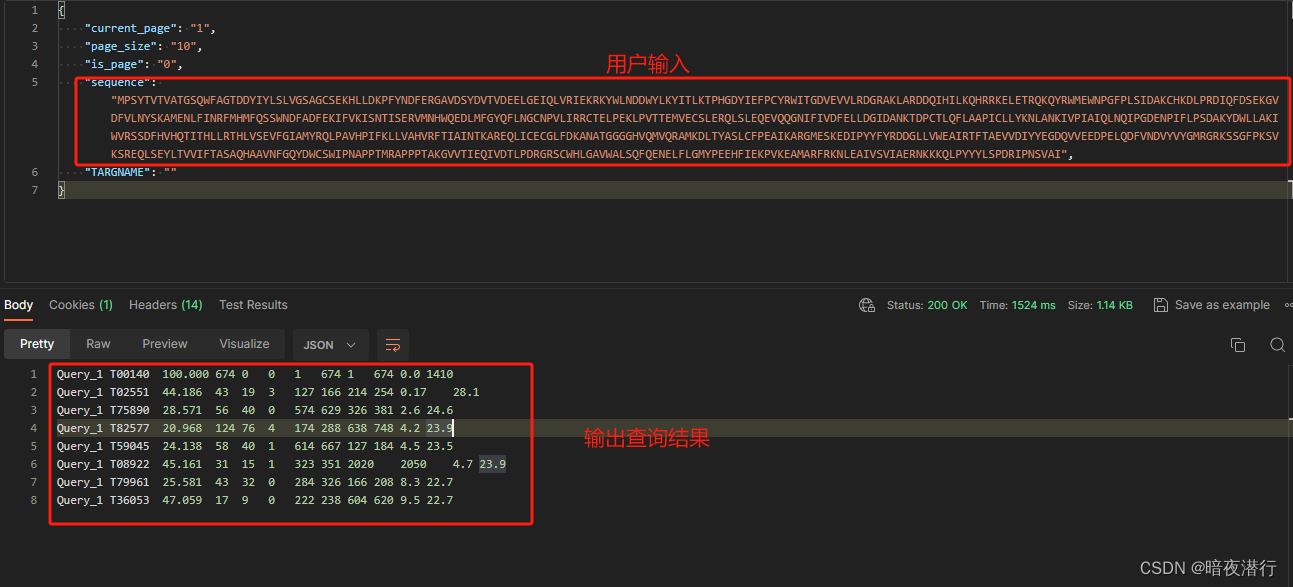





)





函数示例)






)

